-
梯度引导的分子生成扩散模型- GaUDI 评测
GaUDI模型来自于以色列理工Tomer Weiss的2023年发表在预印本ChemRxiv上的工作 《Guided Diffusion for Inverse Molecular Design》。原文链接:Guided Diffusion for Inverse Molecular Design | Materials Chemistry | ChemRxiv | Cambridge Open Engage
GaUDI模型有点像强化学习,目的都是生成特定性质的分子,且效果均非常显著,而且生成过程均存在迭代更新过程。
写在前面
GaUDI模型有点像强化学习,目的都是生成特定性质的分子,且效果均非常显著,而且生成过程均存在迭代更新过程。
但是,二者原理不同。
从迭代过程来说,强化学习生成特定属性的分子迭代发生在生成的批次分子之间,而梯度引导扩散模型是发生在生成一个分子的过程中,因此,从速度上来说,GaUDI模型非常快,很有优势。
从目标函数的形式来说,强化学习是使用目标函数计算生成的分子,获得reward。reward是 一个数值,而是通过数值,调节分子生成器的梯度强度,让分子生成器降低目标分子的损失,于是分子生成器生成更多特定属性的分子。GaUDI模型则不同,目标函数产生的梯度,并未返回到分子生成器中,而是直接用于引导Zt-1,使Zt-1亲向特定属性(方向)。因此,对于任何的分子性质要求,GaUDI模型的分子生成器,即扩散模型,都可以通用,无需重新训练。
从联合概率分布来说,GaUDI模型是联合概率分布,而强化学习不是。GaUDI模型生成的分子更加”平滑“,更具有实用性。强化学习更容易钻模型的漏洞,生成一些奇怪的分子。
一、背景介绍
GaUDI是用于逆向分子设计的一个引导扩散模型,因此文章的题目是《Guided Diffusion for Inverse Molecular Design》。GaUDI对扩散模型中的分子生成器进行了改造,将分子生成过程使用性质预测的函数进行梯度引导,更新Zt-1,使Zt-1更靠近我们期待的性质(例如:logP等)。我们期待的性质可以使单目标也可以是多目标,一切取决于我们的梯度函数f(x,t)。

引导分子生成的梯度来自于目标函数f(x,t), 函数f(x,t)用于根据输入的去噪过程中的Zt-1,预测Zt-1(即:分子)对应的性质。利用预测性质与目标性质之间的损失梯度,更新Zt-1,那么在去噪生成过程中,生成的Zt-1就会越来越靠近我们的期望的性质。当然目标函数f(x,t)需要另外训练。梯度对Zt-1的更新程度可使用调节系数(gradient scalar values, s)来控制。整个去噪采样逻辑如下图:

伪代码如下:

二、文章案例
2.1 多目标优化(全局最优)

作者以 图 A 的 cc-PBHs 为例,目标是增加改分子的稳定性(更低的Erel)。这里作者是进行全局最优的例子,目标函数是生成分子与cc-PBHs 的LUMO, HLG, IP, EA 的MSE加上Erel。作者尝试了不同的调节系数(gradient scalar values, s)结果如图B所示。图C展示了一些具体的例子。
2.2 Out-of-distribution生成
验证GaUDI模型是否可以生成更高的HLG值的分子,限定条件是在相同的环数量下。结果如下图:

图A是数据集中不同环数下的最高HLG分子,图B是生成的分子,图C是不同调节系数下的结果。从结果上来看,生成分子的效果非常显著。
2.3 多目标优化
将目标设置为:ℓ(HLG,IP, EA) = 3 · HLG + IP − EA, 结果如下下图:

整体结果还是非常惊喜的。因为没有任何的强化学习,但是实现了类似的结果,效果非常明显。非常好的弥补了强化学习只能身材个核那个smiles的缺点。
原文代码开源:porannegroup / GaUDI · GitLab
三、模型应用测试
3.1 环境安装
首先,复制代码:
git clone https://github.com/tomer196/GaUDI.git代码目录:
- .
- ├── analyze
- ├── cond_prediction
- ├── data
- ├── edm
- ├── environment.yml
- ├── eval_validity.py
- ├── GaUDI.png
- ├── generation_guidance.py
- ├── __init__.py
- ├── LICENSE
- ├── models_edm.py
- ├── __pycache__
- ├── README.md
- ├── sampling_edm.py
- ├── train_edm.py
- └── utils
安装环境目录,并激活环境:
- conda env create -n GaUDI --file environment.yml
- conda activate GaUDI
如果不行,就分开安装rdkit, pytorch等,注意版本:
- conda install pytorch==2.1.0 torchvision==0.16.0 torchaudio==2.1.0 pytorch-cuda=11.8 -c pytorch -c nvidia
- conda install -c conda-forge rdkit
- pip install matplotlib networkx tensorboard pandas scipy tqdm imageio
安装过程,比较顺利,没有特殊问题。但是centos和win系统比较麻烦,估计要摸索一下。
另外,需要安装几个附属模块:
- pip install tensorboard
- pip install imgaug
3.2 数据准备&下载数据
作者使用到两个数据集:COMPAS 和 PASS,下载链接:
COMPAS:porannegroup / COMPAS · GitLab
需要从上述网址中,下载三个文件,分别是:PASs_csv.tar.gz, PASs_xyz.tar.gz, compas-main-COMPAS-1.zip


在data目录下,创建一个all_data文件夹,将下载的PASs_csv.tar.gz, PASs_xyz.tar.gz, compas-main-COMPAS-1.zip文件上传。然后,分别解压:
- unzip compas-main-COMPAS-1.zip
- tar -zxvf PASs_csv.tar.gz
- tar -zxvf PASs_xyz.tar.gz
- #删除压缩文件
- rm compas-main-COMPAS-1.zip PASs_csv.tar.gz PASs_xyz.tar.gz
然后去更改./data/aromatic_dataloaders.py中get_path函数,关于数据的路径。
- def get_paths(args):
- if not hasattr(args, "dataset"):
- csv_path = args.csv_file
- xyz_path = args.xyz_root
- elif args.dataset == "cata":
- csv_path = "/home/tomerweiss/PBHs-design/data/COMPAS-1x.csv"
- xyz_path = "/home/tomerweiss/PBHs-design/data/peri-cata-89893-xyz"
- elif args.dataset == "peri":
- csv_path = "/home/tomerweiss/PBHs-design/data/peri-xtb-data-55821.csv"
- xyz_path = "/home/tomerweiss/PBHs-design/data/peri-cata-89893-xyz"
- elif args.dataset == "hetro":
- csv_path = "/home/tomerweiss/PBHs-design/data/db-474K-OPV-filtered.csv"
- xyz_path = "/home/tomerweiss/PBHs-design/data/db-474K-xyz"
- elif args.dataset == "hetro-dft":
- csv_path = "/home/tomerweiss/PBHs-design/data/db-15067-dft.csv"
- xyz_path = ""
- else:
- raise NotImplementedError
- return csv_path, xyz_path
改为:
- def get_paths(args):
- if not hasattr(args, "dataset"):
- csv_path = args.csv_file
- xyz_path = args.xyz_root
- elif args.dataset == "cata":
- csv_path = "./data/all_data/compas-main-COMPAS-1/COMPAS-1/COMPAS-1x.csv"
- xyz_path = "/data/all_data/compas-main-COMPAS-1/COMPAS-1/pahs-cata-34072-xyz"
- elif args.dataset == "hetro":
- csv_path = "./data/all_data/db-474K-OPV-filtered.csv"
- xyz_path = "./data/all_data/db-474K-xyz"
- else:
- raise NotImplementedError
- return csv_path, xyz_path
3.3 训练EDM 模型
由于作者没有提供预训练好的checkpoint, 所以需要我们自己训练。回到主目录,然后:
python train_edm.py训练过程了会被记录,将保存在./summary路径下,默认是:cata-test文件夹。整个训练过程,3090显卡需要5h左右。
训练完成后,./summary/cata-test内得到如下目录:
- .
- ├── args.txt
- ├── epoch_0
- ├── epoch_100
- ├── epoch_150
- ├── epoch_200
- ├── epoch_250
- ├── epoch_300
- ├── epoch_350
- ├── epoch_400
- ├── epoch_450
- ├── epoch_50
- ├── epoch_500
- ├── epoch_550
- ├── epoch_600
- ├── epoch_650
- ├── epoch_700
- ├── epoch_750
- ├── epoch_800
- ├── epoch_850
- ├── epoch_900
- ├── epoch_950
- ├── events.out.tfevents.1700200741.a01.3529424.0
- ├── events.out.tfevents.1700200774.a01.3532044.0
- └── model.pt #模型文件
3.4 训练条件模型
同样的,作者没有给checkpoint,条件模型也需要训练。在主目录下,执行:
python cond_prediction/train_cond_predictor.py训练过程将保存在prediction_summary目录中的cata-test文件夹中。训练过程大约也需要4h。训练完成后, prediction_summary的目录如下:
- .
- └── cata-test
- ├── args.txt
- ├── events.out.tfevents.1700211860.a01.63329.0
- └── model.pt
3.5 条件引导扩散的分子生成
3.5.1 使用generation_guidance.py
在完成EDM和条件模型的训练以后,现在可以进行条件引导分子扩散。
首先,需要修改generation_guidance.py文件中的EDM和条件模型路径。将如下内容:
- args = get_edm_args("/home/tomerweiss/PBHs-design/summary/hetro_l9_c196_orientation2")
- cond_predictor_args = get_cond_predictor_args(
- f"/home/tomerweiss/PBHs-design/prediction_summary/"
- f"cond_predictor/hetro_gap_homo_ea_ip_stability_polynomial_2_with_norm"
- )
根据我们前面训练获得的模型,更改为:
- args = get_edm_args("./gaudi/summary/cata-test")
- cond_predictor_args = get_cond_predictor_args(
- f"./gaudi/prediction_summary/cata-test"
- )
在189~193行可以修改,需要生成的分子数量和梯度引导强度。
- ##################### 设置引导梯度的强度,和想要的分子数量 ###################################
- # Set controllable parameters
- args.batch_size = 512 # number of molecules to generate
- scale = 0.6 # gradient scale - the guidance strength
- n_nodes = 10 # number of rings in the generated molecules
- #########################################################################################
然后执行:
python generation_guidance.py就可以生成分子。
但是,存在的问题是:
- plot_graph_of_rings
- plot_rdkit
这两个函数一直无法通过。同时发现,改模型输出的结果不是xyz文件,也不是sdf文件,似乎是图片。这是因为,作者在定义图的时候与我们平常的方法不同,平常的,我们会将一个原子作为一个节点,但是这里作者是将一个环作为一个节点。因此输出的结果比较奇怪。需要其他的转化函数(plot_rdkit),才能输出我们能查看的RDKIT对象。但是我们在运行generation_guidance.py文件时,plot_rdkit文件报错了。所以无法继续运行。
3.5.2 generation_guidance.py文件修正
在研究了其代码和输出结果以后,我们发现代码里面存在变量错误和文件名命名错误。
将以下代码进行修改:
- plot_graph_of_rings(
- x_stable[idx].detach().cpu(),
- atom_type_stable[idx].detach().cpu(),
- filename=f"{dir_name}/{i}.pdf",
- title=f"{best_str}\n {value}",
- dataset=args.dataset,
- )
- plot_rdkit(
- x_stable[idx].detach().cpu(),
- atom_type_stable[idx].detach().cpu(),
- filename=f"{dir_name}/mol_{i}.pdf",
- title=f"{best_str}\n {value}",
- dataset=args.dataset,
- )
修改为:
- try:
- plot_graph_of_rings(
- x_stable[idx].detach().cpu(),
- atom_type[idx].detach().cpu(),
- filename=f"{dir_name}/{i}.png",
- title=f"{best_str}\n {value}",
- dataset=args.dataset,
- )
- plot_rdkit(
- x_stable[idx].detach().cpu(),
- atom_type[idx].detach().cpu(),
- filename=f"{dir_name}/mol_{i}.png",
- title=f"{best_str}\n {value}",
- dataset=args.dataset,
- )
- except:
- pass
使用try语句的原因是,及时通过了有效检测,但是仍然有分子是画不出来的。
生成分子:
python generation_guidance.py运行结束后,将产生./best文件夹目录,有一个以运行日期为名的文件夹,例如:1120_00:42:46_0.6,其中保存对于 Top 5 最优分子的结果,包括节点图以及分子结构。
生成目录示例:
- ./best
- └── 1120_00:42:46_0.6
- ├── 0.png
- ├── 1.png
- ├── 2.png
- ├── 3.png
- ├── 4.png
- ├── all.png
- └── mol_0.png
生成节点图:
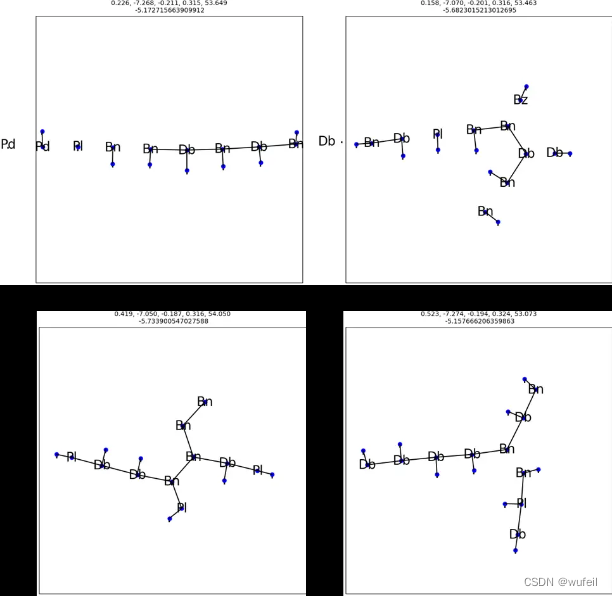
目标函数分子的gap最大,最优分子结构:

由于生成分子结构代码的问题,最终只生成了一个分子的结构图,如下图所示:
- scale=0.8
- stability_dict['mol_valid']=82.00% out of 50
- Mean target function value: -4.7209
- best value: -5.982420921325684, pred: tensor([ 0.2653, -6.9535, -0.1940, 0.3079, 53.0093])
- Mean target function value (from valid): -4.7066
根据输出的结果,最优的分子的目标函数值,gap值为:5.98,但是由于画图的原因,导致无法输出分子结构。能输出分子结构的目标函数值是5.488。
因为在没有引导的时候,即scale参数为0时(在generation_guidance.py文件内调整scale参数的值),相当于直接使用EDM进行分子生成,运行代码输出:
- scale=0
- stability_dict['mol_valid']=100.00% out of 50
- Mean target function value: 0.0738
- best value: -1.7954111099243164, pred: tensor([ 1.1055, -8.5804, -0.2251, 0.3761, 55.9974])
- Mean target function value (from valid): 0.0738
对应的gap的最大值只有1.79,远小于scale为0.8的结果,说明作者提出的梯度引导很符合target函数的要求,效果还不错。但是牺牲的代价是分子的有效率降低了。
3.6 scale梯度调节参数
刚才我们提到scale参数,scale参数是调节引导强度的超参数,当这个值越大时,则生成的分子越满足,越靠近我们的目标函数需求,但是分子的有效率会大大降低,例如,我们将scale设置为2时:只有6%的分子有效。
- scale=2
- stability_dict['mol_valid']=6.00% out of 50
- Mean target function value: -5.2808
- best value: -6.993871212005615, pred: tensor([ 0.2147, -6.5605, -0.1836, 0.2928, 52.0480])
- Mean target function value (from valid): -5.1165
- best value (from stable): tensor([ 0.2562, -7.1488, -0.2008, 0.3158, 52.8709]), score: -5.479823112487793
- best value (from stable): tensor([ 0.3591, -7.2578, -0.1995, 0.3182, 53.5928]), score: -5.1993408203125
- best value (from stable): tensor([ 0.1914, -7.4633, -0.2178, 0.3219, 54.1037]), score: -4.670310020446777
我们将scale设置为4时,报错了,生成了一些奇怪的分子无法计算其gap等分子属性结果,分子的有效率直接降为0:
- scale=4
- stability_dict['mol_valid']=0.00% out of 50
- Mean target function value: -2.2682
- best value: -6.642547130584717, pred: tensor([ 0.3690, -6.6970, -0.1769, 0.3109, 51.9043])
- Mean target function value (from valid): nan
- ~/anaconda3/envs/GaUDI/lib/python3.8/site-packages/numpy/lib/histograms.py:883: RuntimeWarning: invalid value encountered in divide
- return n/db/n.sum(), bin_edges
3.7 条件引导扩散的分子生成(jupyter notebook版本)
作者也提供了一个jupyter notebook的版本,详见文件中的Case_Test.ipynb文件。
导入相关模块:
- import shutil
- import os
- import matplotlib.pyplot as plt
- import torch
- import numpy as np
- import gdown
- from time import time
- from types import SimpleNamespace
- from cond_prediction.train_cond_predictor import get_cond_predictor_model
- from data.aromatic_dataloader import create_data_loaders
- from models_edm import get_model
- from utils.helpers import switch_grad_off, get_edm_args, get_cond_predictor_args
加载预训练模型:
edm_model - 扩散模型,一种在每次迭代时稍微清洁分子的降噪器。
cond_predictor - 条件预测器,预测生成过程中噪声分子的属性。 以时间为条件。
- from cond_prediction.train_cond_predictor import get_cond_predictor_model
- from data.aromatic_dataloader import create_data_loaders
- from models_edm import get_model
- from utils.helpers import switch_grad_off, get_edm_args, get_cond_predictor_args
- # 加载EDM 参数配置
- args = get_edm_args("pre_trained/EDM")
- # 加载条件控制模型参数配置
- cond_predictor_args = get_cond_predictor_args("pre_trained/cond_pred")
- # 数据集的模拟,我们仅需要它来了解数据的形状及其统计数据。
- dataset_mock = SimpleNamespace(
- num_node_features=12,
- num_targets=5,
- mean=torch.tensor([ 1.1734, -9.2780, -0.2356, 0.4042, 53.9615]),
- std=torch.tensor([0.5196, 0.3886, 0.0179, 0.0152, 1.6409]),
- )
- train_loader_mock = SimpleNamespace(dataset=dataset_mock)
- # 加载EDN模型
- edm_model, nodes_dist, prop_dist = get_model(args, train_loader_mock, only_norm=True)
- # 加载条件控制模型
- cond_predictor = get_cond_predictor_model(cond_predictor_args, dataset_mock)
- # 设置模型中的参数为不可训练
- switch_grad_off([edm_model, cond_predictor])
定义生成的内容:
batch_size - 有多少分子 - 影响运行时间。
n_rings 每个分子中环的数量。
Guiding_scale - 对具有较低目标功能的分子的引导强度 - 梯度标量影响生成分子的有效性与其目标分数之间的权衡。
它越高,有效分子的百分比就会减少。 然而,生成的有效分子将更接近定义的目标属性。
目标函数 - 我们想要最小化的函数。 预测属性的任意组合。
- ### 定义分子限制和目标函数 ###
- batch_size = 10
- n_rings = 8
- guidance_scale = 1
- def target_function(_input, _node_mask, _edge_mask, _t):
- pred = cond_predictor(_input, _node_mask, _edge_mask, _t)
- pred = prop_dist.unnormalize(pred)
- gap = pred[:, 0]
- homo = pred[:, 1]
- ea = pred[:, 2] * 27.2114 # hartree to eV
- ip = pred[:, 3] * 27.2114 # hartree to eV
- return -gap
- # return ip + ea + 3 * gap
生成分子:
- from sampling_edm import sample_guidance
- # 初始化生成分子,由于这里的模型是一个节点是一个环,所以节点比较奇怪
- nodesxsample = torch.Tensor([n_rings] * batch_size).long()
- # nodesxsample = nodes_dist.sample(args.batch_size)
- start_time = time()
- x, one_hot, node_mask, edge_mask = sample_guidance(
- args,
- edm_model,
- target_function,
- nodesxsample,
- scale=guidance_scale,
- )
- print(f"Generated {x.shape[0]} molecules in {time() - start_time:.2f} seconds")
使用 RDKit 评估分子的有效性并过滤掉无效分子:
- target_function_values = get_target_function_values(
- x, one_hot, target_function, node_mask, edge_mask, edm_model
- )
- pred = prop_dist.unnormalize(predict(cond_predictor, x, one_hot, node_mask, edge_mask, edm_model))
绘制最佳分子(具有较低的目标函数值)。 生成的分子表示为环图 (GOR),并带有表示环方向的加法节点。
- from utils.plotting import plot_grap_of_rings_inner, plot_rdkit
- best_idxs = target_function_values.argsort()
- n_plot = min(3, x.shape[0])
- fig, axes = plt.subplots(1, n_plot, figsize=(5*n_plot, 7))
- for i, idx in enumerate(best_idxs[:n_plot]):
- pred_i = pred[idx]
- title = f"HLG: {pred_i[0]:.2f}, HOMO: {pred_i[1]:.2f}, EA: {pred_i[2]:.2f}, IP: {pred_i[3]:.2f}"
- plot_grap_of_rings_inner(axes[i], x[idx], one_hot.argmax(2)[idx], title=title, dataset=args.dataset, )
- plt.show()
输出:
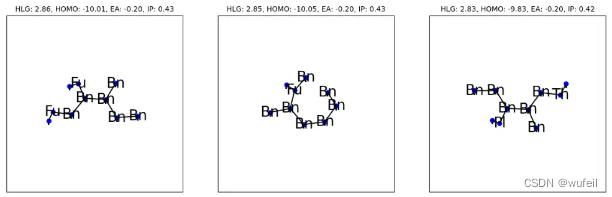
转换成完整的分子结构:
- fig, axes = plt.subplots(1, n_plot, figsize=(5*n_plot, 7))
- for i, idx in enumerate(best_idxs[:n_plot]):
- pred_i = pred[idx]
- title = f"HLG: {pred_i[0]:.2f}, HOMO: {pred_i[1]:.2f}, EA: {pred_i[2]:.2f}, IP: {pred_i[3]:.2f}"
- plot_rdkit(x[idx], one_hot.argmax(2)[idx], axes[i], title=title, dataset=args.dataset)
- plt.show()
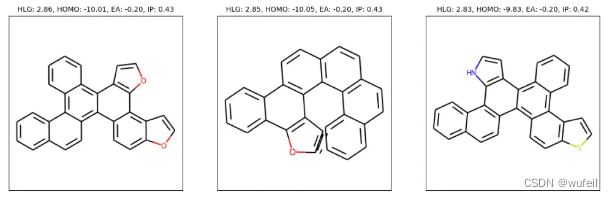
四、总结
1. GaUDI是一个梯度引导的分子生成扩散模型,可以在不重新训练分子生成模型的基础上,通过目标函数的梯度引导分子生成,从而产生特定属性的分子。文中以Gap等分子属性作为目标,验证了其方法的可行性。
2. 对于药物设计,分子设计等而言,该方法不能直接使用。首先,该模型对分子的表示,以环为节点,不符合我们几何神经网络中关于分子的设定,导致分子还原存在困难。这也是为什么上述结果中,有着GOA图的原因,以及有大量分子无法输出结构的原因。数据集中的分子均为多环结构,不是药物设计中常用的分子结构,模型的偏差很严重。
结论是,该模型不推荐,难用不友好,但是该方法提供了非常好的概念验证,值得进一步研究学习。
-
相关阅读:
手撕520页PDF高级文档,成功“挤掉”7年开发架构师,牛逼
【读论文】DIVFusion: Darkness-free infrared and visible image fusion
记一次JVM参数调优经历
2022宁夏杯D 包装纸箱设计
Spring Security 实现动态权限菜单方案(附源码)
《Vue.js实战》8.2实战
Java面试题(JVM篇)
中国净初级生产力年度合成产品NPP(MYD17A3H.006)
港科夜闻|国务院发文指出将打造重大科技创新平台,稳步推进粤港澳教育合作,加快与香港科大、中科院共建省实验室...
浅入浅出 1.7和1.8的 HashMap
- 原文地址:https://blog.csdn.net/wufeil7/article/details/134541056
