-
plink遗传数据质控--每个个体QC、每个marker(变异)质控、全基因组关联meta分析QC
0.4 遗传数据质控
0.4.1 每个个体QC(per-individual QC)
主要有5步
- DNA质量差(call率低,缺失基因型);
- 常染色体杂合度高,表明可能样品污染或杂合度低,这可能是近亲繁殖造成的。
- 性别信息不一致
- 重复的或相关的
- 来自不同祖先的群体
1)低质量基因型个体的鉴定
5%的缺失基因型比例
plink --bfile 1kg_hm3 --mind 0.05 --make-bed --out 1kg_hm3_mind005- 1
2)常染色体杂合度鉴定
在分析中排除了杂合度高和杂合度低的个体。
--het,计算杂合度,主要生成两个文件,1kg_hm3_het.het, 1kg_hm3_het.logplink --bfile 1kg_hm3 --het --out 1kg_hm3_het- 1
每个样本的杂合度计算为杂合子基因型call数与非缺失calls总数之比。
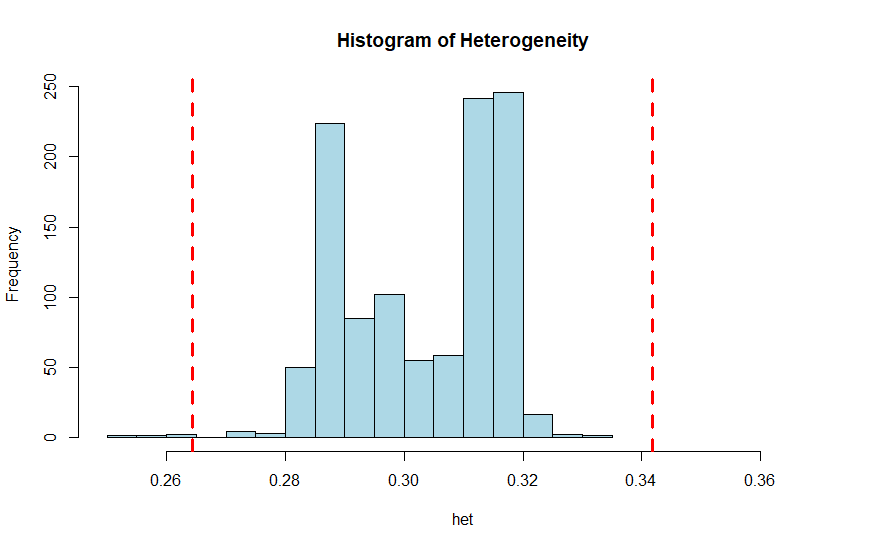
--exclude,可以将具有非常高或非常低的平均杂合度值的异常值去掉,规则是去除那些偏离特定样本杂合性平均值士3个标准差的个体。杂合性统计数据的分布
library(tidyverse) heterogeneity_stats <- read.table("../1kg_hm3_het.het",header = T) colnames(heterogeneity_stats) het <- (heterogeneity_stats$N.NM.-heterogeneity_stats$O.HOM.)/heterogeneity_stats$N.NM. min <- mean(het)-3*sd(het) max <- mean(het)+3*sd(het) hist(het,col="lightblue",xlim=c(0.25,0.37), main="Histogram of Heterogeneity") abline(v = c(min,max), col = "red", lwd=3, lty=2)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
3)识别性别信息不一致的个体
男性只有一个X染色体副本,因此性染色体中的任何标记都不可能是杂合的。
plink --bfile hapmap-ceu --check-sex --out hapmap_sexccheck- 1
生成.sexcheck文件, F为X染色体近交系数,雄性的X染色体近交系系数应大于0.8,雌性的X染色体近交系系数应小于0.2。
$ head hapmap_sexccheck.sexcheck FID IID PEDSEX SNPSEX STATUS F 1334 NA12144 1 1 OK 0.9999 1334 NA12145 2 2 OK -0.06528 1334 NA12146 1 1 OK 0.9999 1334 NA12239 2 2 OK 0.05498 1340 NA06994 1 1 OK 0.9999 1340 NA07000 2 2 OK -0.1001 1340 NA07022 1 1 OK 0.9998 1340 NA07056 2 2 OK -0.03786 1341 NA07034 1 1 OK 0.9999- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
4)识别重复的或相关的个体
即无意重复【inadvertent duplications】和神秘联系[cryptic relatedness](近亲)。
见第9章
5)不同血统的个体的鉴定:群体分层
主要是进行PCA分析。
0.4.2 每个marker(变异)质控
主要有5步
- 排除低检出率SNP
- 去除低等位基因频率的SNP
- 识别和排除具有极端偏离哈代温伯格平衡的变异
- 在病例对照研究中,排除组间检出call率极不同的snp
- 在输入snp的情况下,排除了低输入质量的变异的研究
1)低检出率SNP
call率低于95%的变异被排除在分析之外。用命令
--genoplink --bfile 1kg_hm3 --geno 0.05 --make-bed --out 1kg_hm3_geno- 1
2)低等位基因频率SNP
排除低等位基因频率的原因,首先,在低MAF的情况下,缺乏检测任何真正的snp-性状关联的能力。其次,这些snp往往更容易出现基因分型错误。
--maf# 去掉MAF小于0.01的位点plink --bfile 1kg_hm3 --maf 0.01 --make-bed --out 1kg_hm3_maf- 1
如果有大样本,如100,000,MAF阈值可为0.01;如果N=10,000,MAF为0.05.
3)偏离哈温平衡
HWE假设一个无限大的种群,没有选择、突变或迁移,并且如果没有违反任何条件,基因型和等位基因频率在世代中是恒定的。
--hweplink --bfile 1kg_hm3 --hwe 0.00001 --make-bed --out 1kg_hm3_hwe- 1
产生四个新文件,.log .bed .fam .bim
排除指标根据你是二元的还是定量的(例如,连续的)特征而不同。
对于二元性状,规则通常在cases中,HWE p-value < 1 0 − 10 10^{-10} 10−10,在control中< 1 0 − 6 10^{-6} 10−6。
对于数量性状,HWE p值< 1 0 − 6 10^{-6} 10−6。
4)整合不同QC的文件
plink --bfile 1kg_hm3 \ # 输入二进制文件 --mind 0.03 \ # 包含至少97%完整的高基因分型个体 #有3%的SNP都是缺失的,那么就删掉该个体 --geno 0.05 \ # call率低于95%的变异被排除在分析之外 #一个SNP在个体中5%都是缺失的,那么就删掉该SNP --maf 0.01 \ # 去掉MAF小于0.01的位点 --hwe 0.00001 \ --exclude individuals_failQC.txt \ # 去除未完成个体水平QC的个体 --make-bed --out 1kg_hm3_QC- 1
- 2
- 3
- 4
- 5
- 6
- 7
0.4.3 全基因组关联meta分析QC
1)filter
- 使用一个通用的参考来使所有文件中变异的碱基对位点一致。
- 评估关联结果的信息,特别是来自大量文件的信息。效应等位基因有缺失信息的变异、可变等位基因变异、效应估计值、标准误差或p值不可信的变异将从样本中删除。
- 非双等位基因变异或单型,都被排除在最终结果列表之外。
- 来自罕见变异的结果通常是有问题的,可能会影响结果。MAF低于1%被剔除,
- 输入质量影响相关性分析结果质量。输入质量低于0.7的变异被剔除。
2)诊断检查
等位基因频率图
检查变异(1)都以相同的方式编码,在等位基因频率和链取向上没有错误,(2)在研究中有相似的等位基因频率。

y是期望等位基因频率,X是来自参考群体的等位基因频率。在这里我们只绘制等位基因频率差异为0.2的点
森林图
QQ图
参考:
An Introduction to Statistical Genetic Data Analysis. -
相关阅读:
字典服务的设计与管理
Qt Quick读取本地文件并显示成表格
[2022强网杯] polydiv和gamemaster
Ruby on Rails Post项目设置网站初始界面
SQL优化记录
16/32/64位操作系统下,各种变量类型分别占多少字节
持续测试简介
SpringMVC(二)@RequestMapping注解
opencv 直方图比较
PMI-ACP(103:1-16)
- 原文地址:https://blog.csdn.net/ziixiaoshenwang/article/details/126945860