-
基于Python实现的遗传算法求TSP问题
遗传算法求TSP问题
目录
人工智能第四次实验报告 1
遗传算法求TSP问题 1
一 、问题背景 1
1.1 遗传算法简介 1
1.2 遗传算法基本要素 2
1.3 遗传算法一般步骤 2
二 、程序说明 3
2.3 选择初始群体 4
2.4 适应度函数 4
2.5 遗传操作 4
2.6 迭代过程 4
三 、程序测试 5
3.1 求解不同规模的TSP问题的算法性能 5
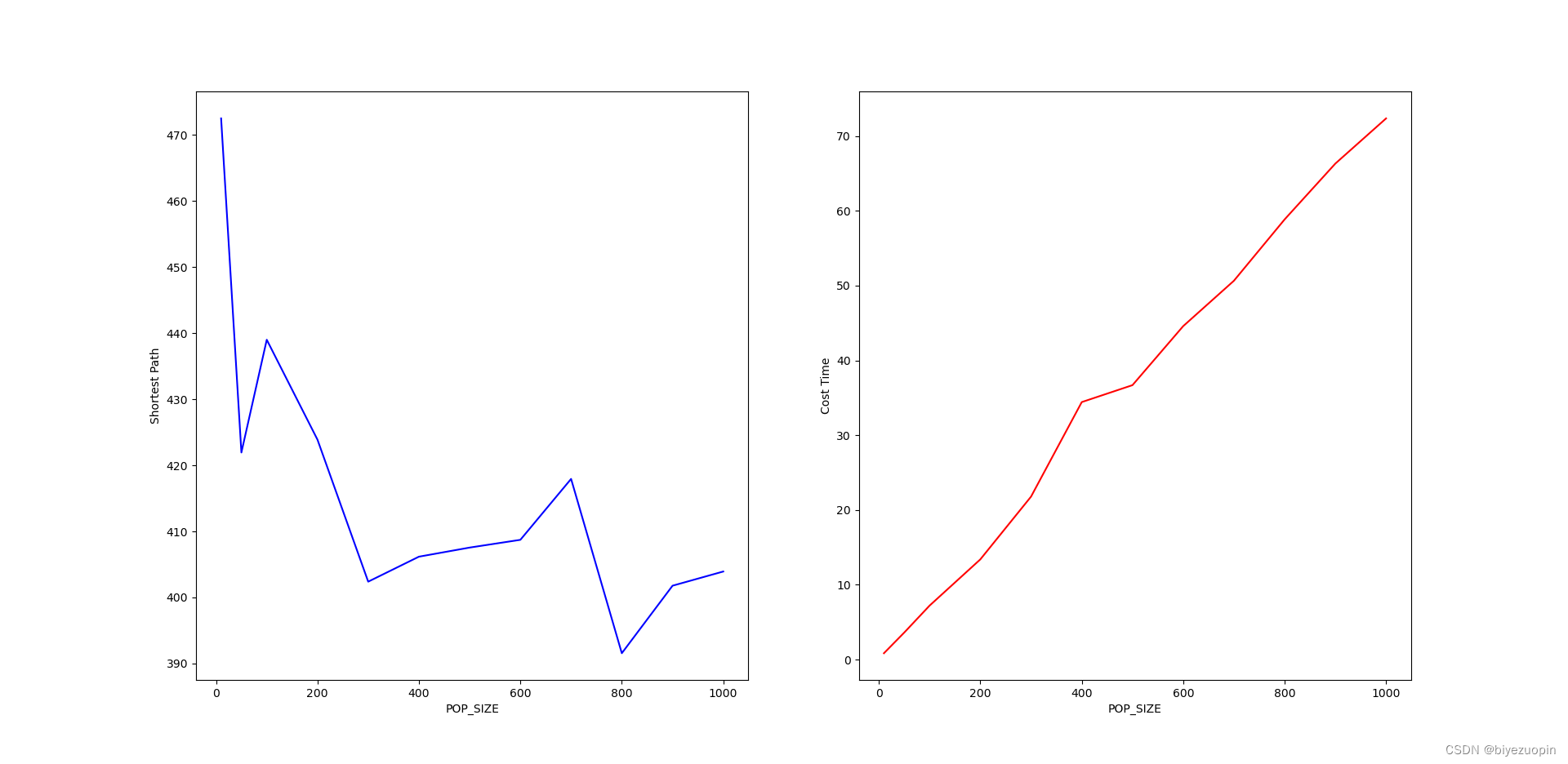
3.2 种群规模对算法结果的影响 5
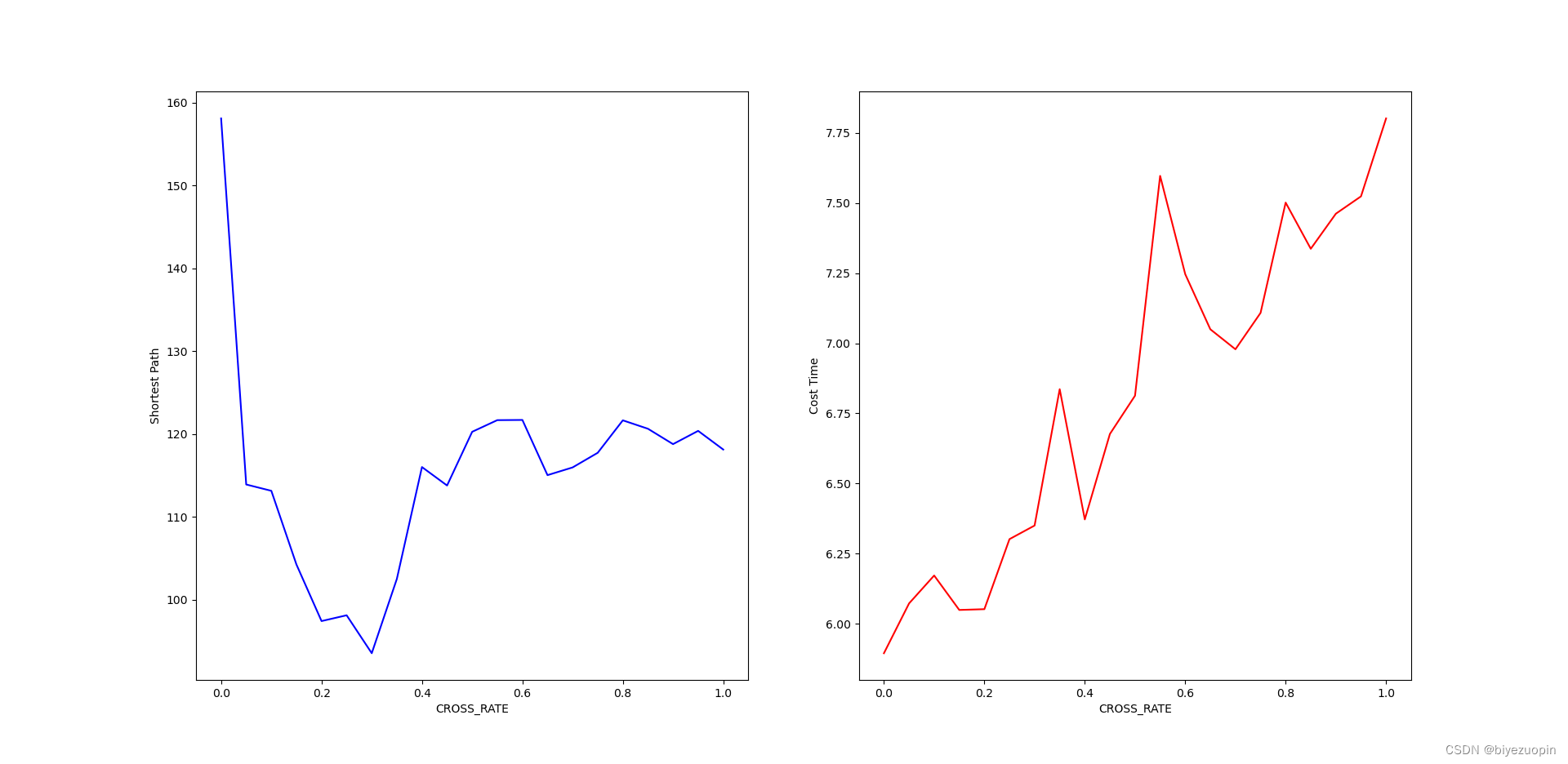
3.3 交叉概率对算法结果的影响 6
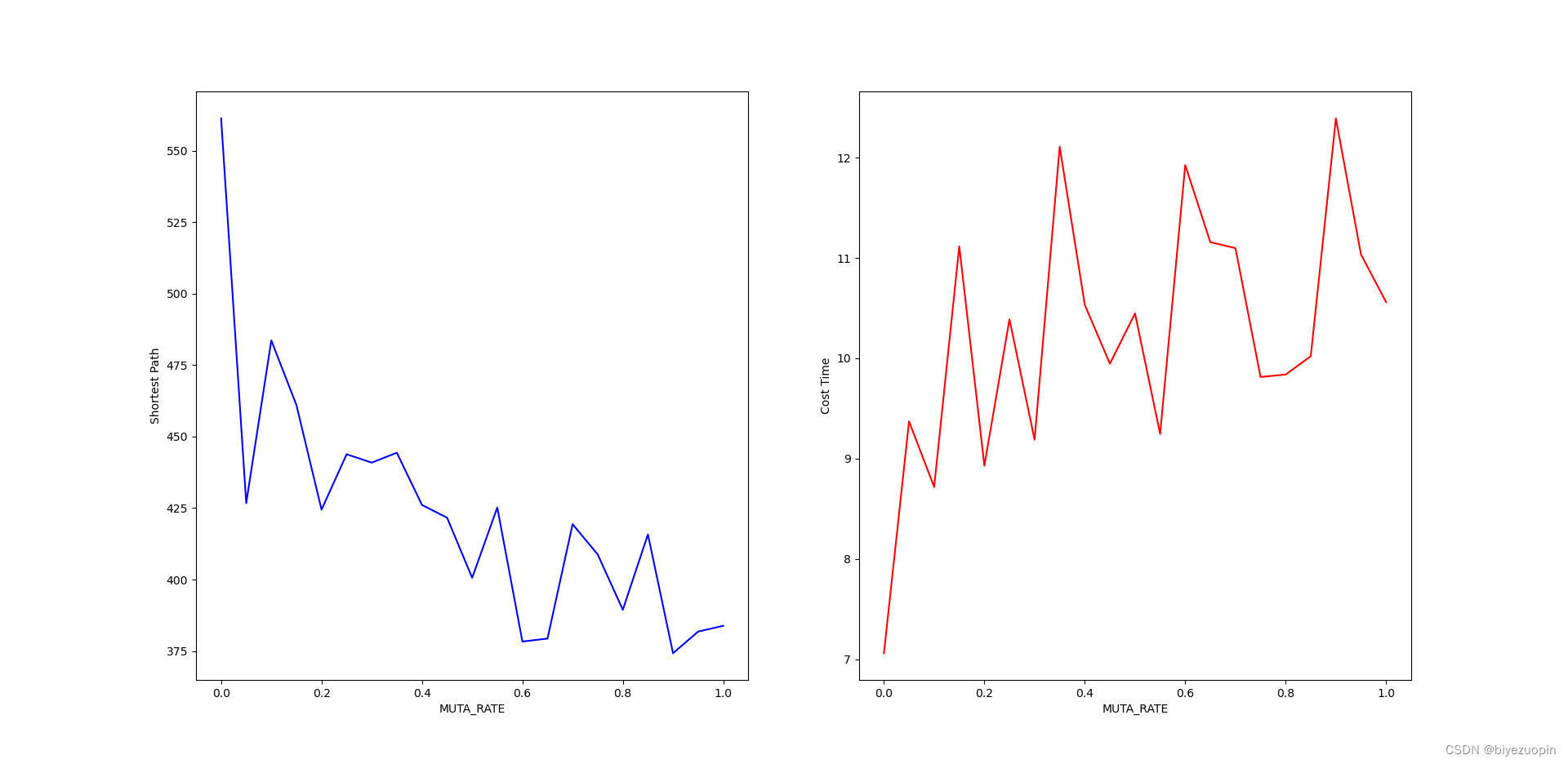
3.4 变异概率对算法结果的影响 7
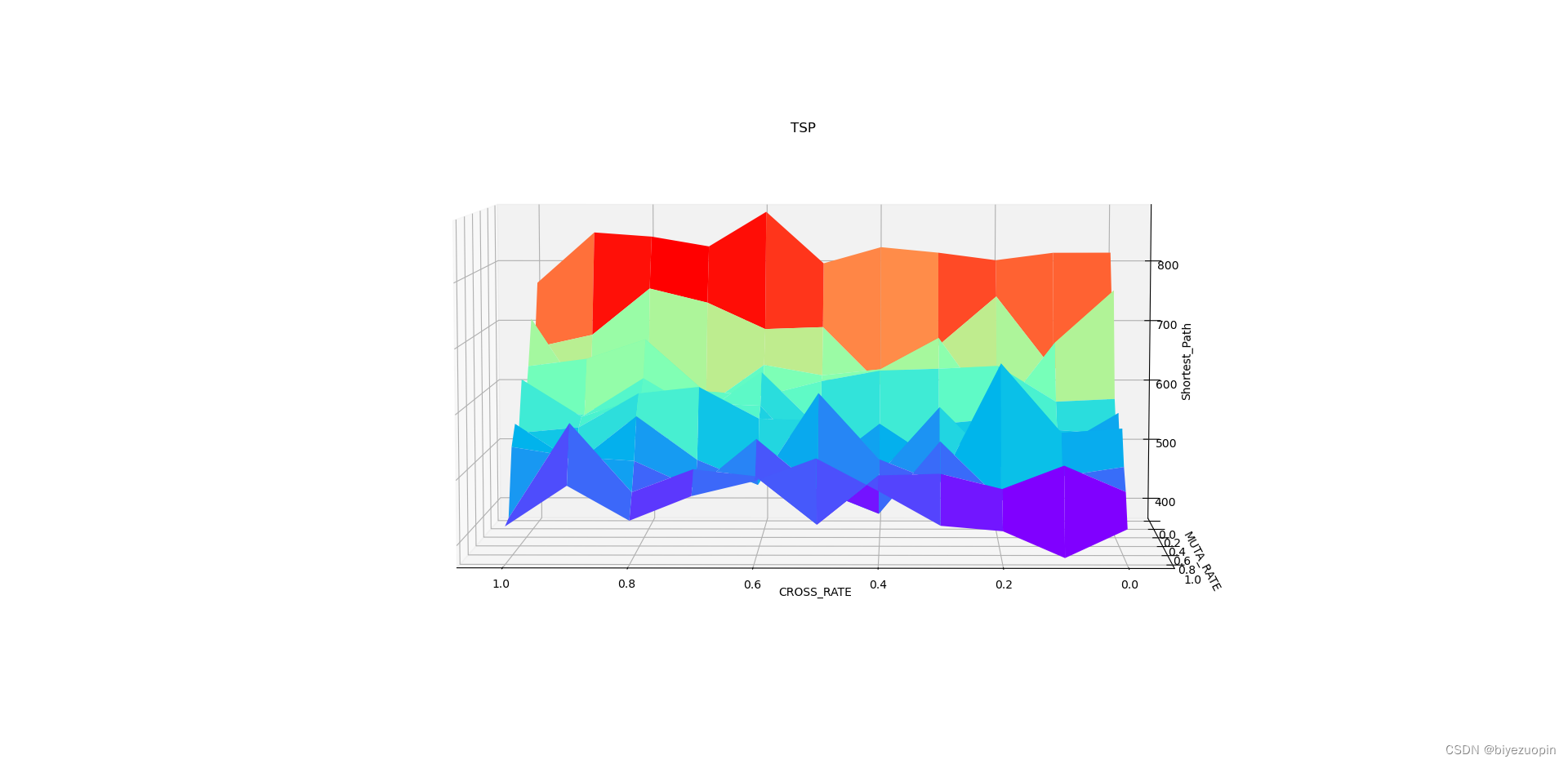
3.5 交叉概率和变异概率对算法结果的影响 7
四 、算法改进 8
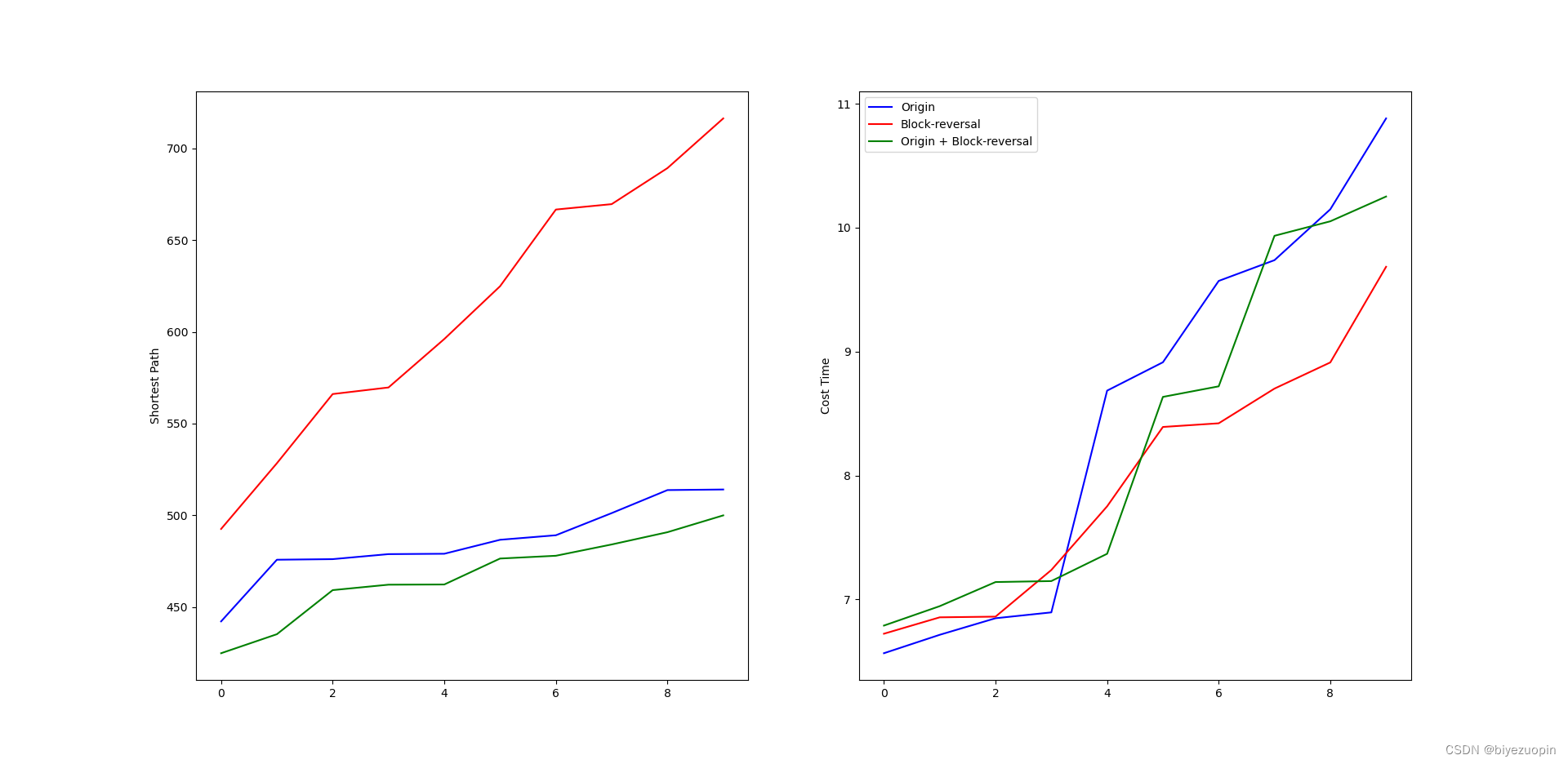
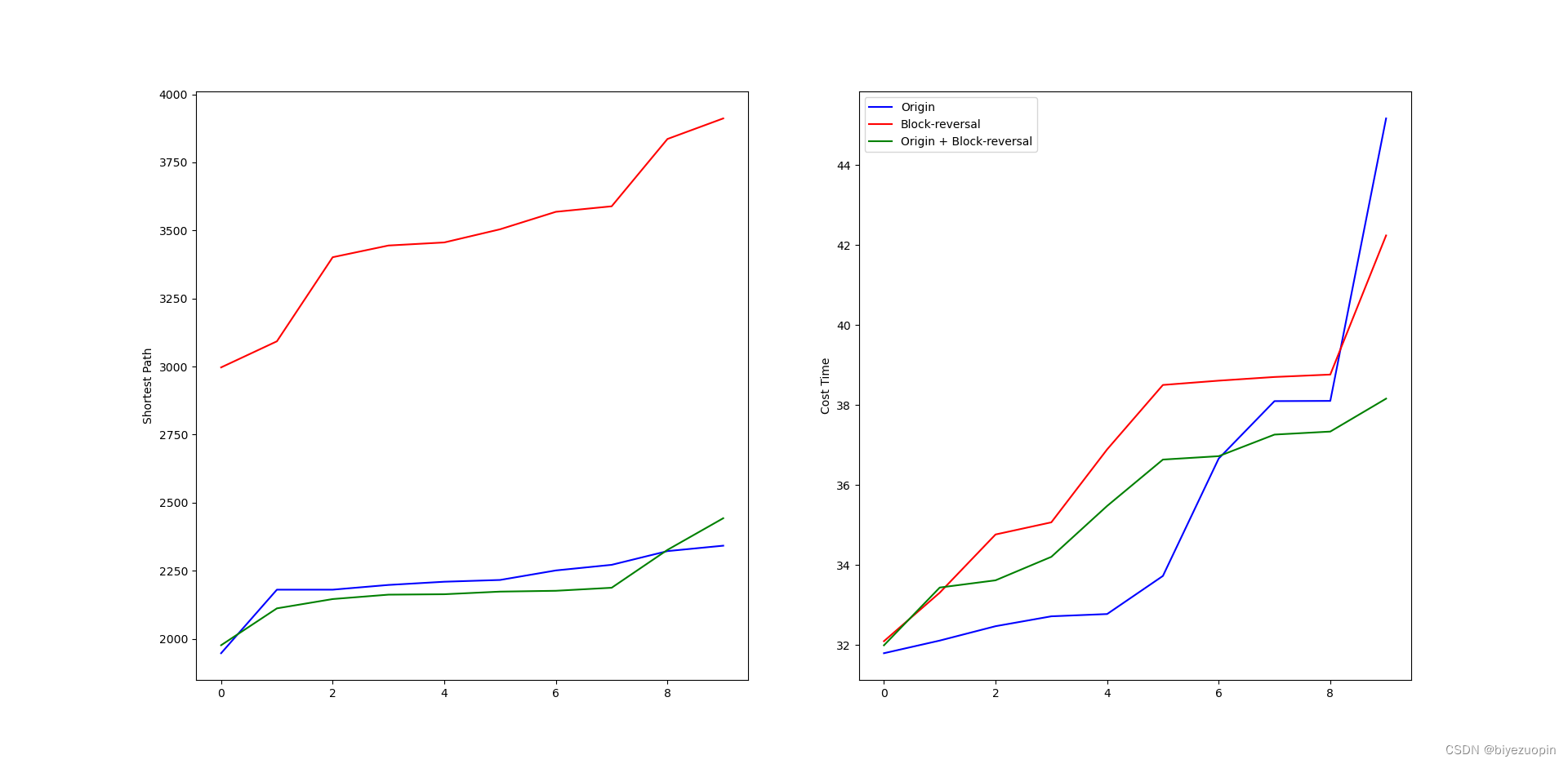
4.1 块逆转变异策略 8
4.2 锦标赛选择法 9
五 、实验总结 10
一 、问题背景
1.1遗传算法简介
遗传算法是一种进化算法,基于自然选择和生物遗传等生物进化机制的一种搜索算法,其通过选 择、重组和变异三种操作实现优化问题的求解。它的本质是从原问题的一组解出发改进到另一组较好的 解,再从这组改进的解出发进一步改进。在搜索过程中,它利用结构和随机的信息,是满足目标的决策 获得最大的生存可能,是一种概率型算法。
遗传算法主要借用生物中“适者生存”的原则,在遗传算法中,染色体对应的是数据或数组,通常由 一维的串结构数据来表示。串上的各个位置对应一个基因座,而各个位置上所取的值对等位基因。遗传 算法处理的是基因型个体,一定数量的个体组成了群体。群体的规模就是个体的数目。不同个体对环境 的适应度不同,适应度打的个体被选择进行遗传操作产生新个体。本文转载自http://www.biyezuopin.vip/onews.asp?id=16719每次选择两个染色体进行产生一组新 染色体,染色体也可能发生变异,得到下一代群体。
1.2遗传算法基本要素
1.参数编码:可以采用位串编码、实数编码、多参数级联编码等
2.设定初始群体:
1.启发 / 非启发给定一组解作为初始群体
2.确定初始群体的规模
3.设定适应度函数:将目标函数映射为适应度函数,可以进行尺度变换来保证非负、归一等特性
4.设定遗传操作:
1.选择:从当前群体选出一系列优良个体,让他们产生后代个体
2.交叉:两个个体的基因进行交叉重组来获得新个体
3.变异:随机变动个体串基因座上的某些基因
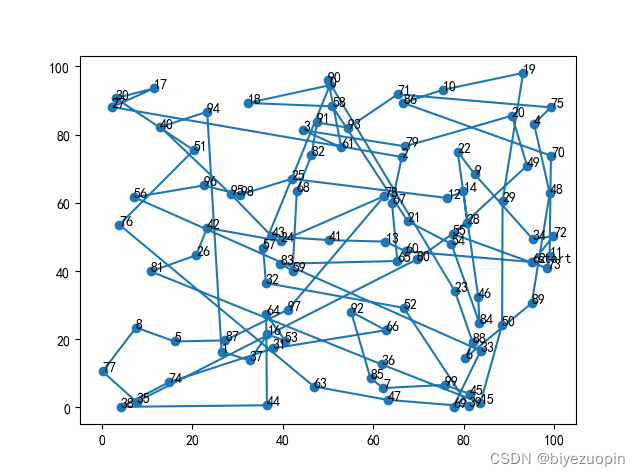
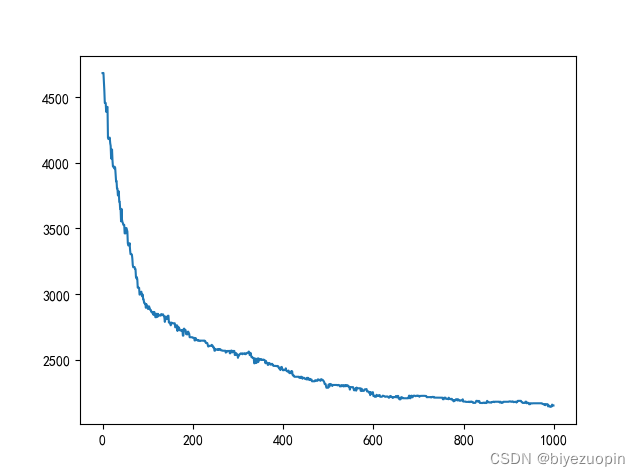
5.设定控制参数:例如变异概率、交叉程度、迭代上限等。import numpy as np import random import matplotlib.pyplot as plt import copy import time from matplotlib.ticker import MultipleLocator from scipy.interpolate import interpolate CITY_NUM = 20 City_Map = 100 * np.random.rand(CITY_NUM, 2) DNA_SIZE = CITY_NUM #编码长度 POP_SIZE = 100 #种群大小 CROSS_RATE = 0.6 #交叉率 MUTA_RATE = 0.2 #变异率 Iterations = 1000 #迭代次数 # 根据DNA的路线计算距离 def distance(DNA): dis = 0 temp = City_Map[DNA[0]] for i in DNA[1:]: dis = dis + ((City_Map[i][0]-temp[0])**2+(City_Map[i][1]-temp[1])**2)**0.5 temp = City_Map[i] return dis+((temp[0]-City_Map[DNA[0]][0])**2+(temp[1]-City_Map[DNA[0]][1])**2)**0.5 # 计算种群适应度,这里适应度用距离的倒数表示 def getfitness(pop): temp = [] for i in range(len(pop)): temp.append(1/(distance(pop[i]))) return temp-np.min(temp) + 0.000001 # 选择:根据适应度选择,以赌轮盘的形式,适应度越大的个体被选中的概率越大 def select(pop, fitness): s = fitness.sum() temp = np.random.choice(np.arange(len(pop)), size=POP_SIZE, replace=True,p=(fitness/s)) p = [] for i in temp: p.append(pop[i]) return p # 4.2 选择:锦标赛选择法 def selectII(pop, fitness): p = [] for i in range(POP_SIZE): temp1 = np.random.randint(POP_SIZE) temp2 = np.random.randint(POP_SIZE) DNA1 = pop[temp1] DNA2 = pop[temp2] if fitness[temp1] > fitness[temp2]: p.append(DNA1) else: p.append(DNA2) return p # 变异:选择两个位置互换其中的城市编号 def mutation(DNA, MUTA_RATE): if np.random.rand() < MUTA_RATE: # 以MUTA_RATE的概率进行变异 # 随机产生两个实数,代表要变异基因的位置,确保两个位置不同,将2个所选位置进行互换 mutate_point1 = np.random.randint(0, DNA_SIZE) mutate_point2 = np.random.randint(0,DNA_SIZE) while(mutate_point1 == mutate_point2): mutate_point2 = np.random.randint(0,DNA_SIZE) DNA[mutate_point1],DNA[mutate_point2] = DNA[mutate_point2],DNA[mutate_point1] # 4.1 变异:在父代中随机选择两个点,然后反转之间的部分 def mutationII(DNA, MUTA_RATE): if np.random.rand() < MUTA_RATE: mutate_point1 = np.random.randint(0, DNA_SIZE) mutate_point2 = np.random.randint(0, DNA_SIZE) while (mutate_point1 == mutate_point2): mutate_point2 = np.random.randint(0, DNA_SIZE) if(mutate_point1 > mutate_point2): mutate_point1, mutate_point2 = mutate_point2, mutate_point1 DNA[mutate_point1:mutate_point2].reverse() # 4.1 变异:调用 I 和 II def mutationIII(DNA, MUTA_RATE): mutationII(DNA, MUTA_RATE) mutation(DNA, MUTA_RATE) # 交叉变异 # muta = 1时变异调用 mutation; # muta = 2时变异调用 mutationII; # muta = 3时变异调用 mutationIII def crossmuta(pop, CROSS_RATE, muta=1): new_pop = [] for i in range(len(pop)): # 遍历种群中的每一个个体,将该个体作为父代 n = np.random.rand() if n >= CROSS_RATE: # 大于交叉概率时不发生变异,该子代直接进入下一代 temp = pop[i].copy() new_pop.append(temp) # 小于交叉概率时发生变异 if n < CROSS_RATE: # 选取种群中另一个个体进行交叉 list1 = pop[i].copy() list2 = pop[np.random.randint(POP_SIZE)].copy() status = True # 产生2个不相等的节点,中间部分作为交叉段,采用部分匹配交叉 while status: k1 = random.randint(0, len(list1) - 1) k2 = random.randint(0, len(list2) - 1) if k1 < k2: status = False k11 = k1 # 两个DNA中待交叉的片段 fragment1 = list1[k1: k2] fragment2 = list2[k1: k2] # 交换片段后的DNA list1[k1: k2] = fragment2 list2[k1: k2] = fragment1 # left1就是 list1除去交叉片段后剩下的DNA片段 del list1[k1: k2] left1 = list1 offspring1 = [] for pos in left1: # 如果 left1 中有与待插入的新片段相同的城市编号 if pos in fragment2: # 找出这个相同的城市编号在在原DNA同位置编号的位置的城市编号 # 循环查找,直至这个城市编号不再待插入的片段中 pos = fragment1[fragment2.index(pos)] while pos in fragment2: pos = fragment1[fragment2.index(pos)] # 修改原DNA片段中该位置的城市编号为这个新城市编号 offspring1.append(pos) continue offspring1.append(pos) for i in range(0, len(fragment2)): offspring1.insert(k11, fragment2[i]) k11 += 1 temp = offspring1.copy() # 根据 type 的值选择一种变异策略 if muta == 1: mutation(temp, MUTA_RATE) elif muta == 2: mutationII(temp, MUTA_RATE) elif muta == 3: mutationIII(temp, MUTA_RATE) # 把部分匹配交叉后形成的合法个体加入到下一代种群 new_pop.append(temp) return new_pop def print_info(pop): fitness = getfitness(pop) maxfitness = np.argmax(fitness) # 得到种群中最大适应度个体的索引 print("最优的基因型:", pop[maxfitness]) print("最短距离:",distance(pop[maxfitness])) # 按最优结果顺序把地图上的点加入到best_map列表中 best_map = [] for i in pop[maxfitness]: best_map.append(City_Map[i]) best_map.append(City_Map[pop[maxfitness][0]]) X = np.array((best_map))[:,0] Y = np.array((best_map))[:,1] # 绘制地图以及路线 plt.figure() plt.rcParams['font.sans-serif'] = ['SimHei'] plt.scatter(X,Y) for dot in range(len(X)-1): plt.annotate(pop[maxfitness][dot],xy=(X[dot],Y[dot]),xytext = (X[dot],Y[dot])) plt.annotate('start',xy=(X[0],Y[0]),xytext = (X[0]+1,Y[0])) plt.plot(X,Y) # 3.2 种群规模对算法结果的影响 def pop_size_test(): global POP_SIZE ITE = 3 # 每个值测试多次求平均数以降低随机误差 i_list = [10, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000] b_list = [] t_list = [] for i in i_list: print(i) POP_SIZE = i time_cost = 0 min_path = 0 for j in range(ITE): time_start = time.time() ans = tsp_solve() min_path += min(ans) time_end = time.time() time_cost += time_end - time_start b_list.append(min_path / ITE) t_list.append(time_cost / ITE) show_test_result(i_list, b_list, t_list, "POP_SIZE") # 3.3 交叉概率对算法结果的影响 def cross_rate_test(): global CROSS_RATE ITE = 3 # 每个值测试多次求平均数以降低随机误差 i_list = range(0, 21) b_list = [] t_list = [] ii_list = [] # [0, 0.05, 0.1, ... 0.95, 1] for i in i_list: print(i) CROSS_RATE = 0.05 * i ii_list.append(CROSS_RATE) time_cost = 0 min_path = 0 for j in range(ITE): time_start = time.time() ans = tsp_solve() min_path += min(ans) time_end = time.time() time_cost += time_end - time_start b_list.append(min_path / ITE) t_list.append(time_cost / ITE) show_test_result(ii_list, b_list, t_list, "CROSS_RATE") # 3.4 变异概率对算法结果的影响 def muta_rate_test(): global MUTA_RATE ITE = 3 # 每个值测试多次求平均数以降低随机误差 i_list = range(0, 21) b_list = [] t_list = [] ii_list = [] # [0, 0.05, 0.1, ... 0.95, 1] for i in i_list: print(i) MUTA_RATE = 0.05 * i ii_list.append(MUTA_RATE) time_cost = 0 min_path = 0 for j in range(ITE): time_start = time.time() ans = tsp_solve() min_path += min(ans) time_end = time.time() time_cost += time_end - time_start b_list.append(min_path / ITE) t_list.append(time_cost / ITE) show_test_result(ii_list, b_list, t_list, "MUTA_RATE") # 3.5 交叉概率和变异概率对算法结果的影响 def cross_muta_test(): s = np.array([0, 0.1, 0.2, 0.3, 0.4, 0.5, 0.6, 0.7, 0.8, 0.9, 1.0]) X, Y = np.meshgrid(s,s) Z = np.zeros(shape=(11, 11)) global MUTA_RATE global CROSS_RATE for i in range(11): for j in range(11): print(str(i) + ":" + str(j)) CROSS_RATE = X[0,i] MUTA_RATE = Y[0,j] ans = tsp_solve() Z[i, j] = min(ans) ax = plt.axes(projection='3d') ax.plot_surface(X, Y, Z, rstride=1, cstride=1,cmap='rainbow', edgecolor='none') ax.set_xlabel("CROSS_RATE") ax.set_ylabel("MUTA_RATE") ax.set_zlabel("Shortest_Path") ax.set_title('TSP') plt.show() # 3.2-3.4 生成参数测试结果的可视化图表 def show_test_result(i_list, b_list, t_list, msg): ax1 = plt.subplot(121) ax1.plot(i_list, b_list, 'b') ax1.set_xlabel(msg) ax1.set_ylabel("Shortest Path") ax2 = plt.subplot(122) ax2.plot(i_list, t_list, 'r') ax2.set_xlabel(msg) ax2.set_ylabel("Cost Time") plt.show() # 求解TSP问题并返回最大值 # muta 指定变异方式,sel 指定选择方式 def tsp_solve(muta=1, sel=1): pop = [] li = list(range(DNA_SIZE)) for i in range(POP_SIZE): random.shuffle(li) l = li.copy() pop.append(l) best_dis = [] # 进行选择,交叉,变异,并把每代的最优个体保存在best_dis中 for i in range(Iterations): # 迭代N代 pop = crossmuta(pop, CROSS_RATE, muta=muta) fitness = getfitness(pop) maxfitness = np.argmax(fitness) best_dis.append(distance(pop[maxfitness])) if sel == 1: pop = select(pop, fitness) # 选择生成新的种群 elif sel == 2: pop = selectII(pop, fitness) # 选择生成新的种群 return best_dis # 4.1 块逆转变异策略对比测试 def opt1_test(): ITE = 20 # 测试次数 i_list = range(ITE) b_list = [] # 每次求出的最短路径 t_list = [] # 每次求解的耗时 b_listII = [] t_listII = [] b_listIII = [] t_listIII = [] for i in i_list: print(i) # I. 原两点互换异策略 time_start = time.time() b_list.append(min(tsp_solve(muta=1))) time_end = time.time() t_list.append(time_end - time_start) # II. 块逆转变异策略 time_startII = time.time() b_listII.append(min(tsp_solve(muta=2))) time_endII = time.time() t_listII.append(time_endII - time_startII) # III. 同时使用上述两种编译策略 time_startIII = time.time() b_listIII.append(min(tsp_solve(muta=3))) time_endIII = time.time() t_listIII.append(time_endIII - time_startIII) # 做排序处理,方便比较 b_list.sort() t_list.sort() b_listII.sort() t_listII.sort() b_listIII.sort() t_listIII.sort() ax1 = plt.subplot(121) ax1.plot(i_list, b_list, 'b', label="Origin") ax1.plot(i_list, b_listII, 'r', label="Block-reversal") ax1.plot(i_list, b_listIII, 'g', label="Origin + Block-reversal") ax1.set_ylabel("Shortest Path") ax2 = plt.subplot(122) ax2.plot(i_list, t_list, 'b', label="Origin") ax2.plot(i_list, t_listII, 'r', label="Block-reversal") ax2.plot(i_list, t_listIII, 'g', label="Origin + Block-reversal") ax2.set_ylabel("Cost Time") plt.legend() plt.show() # 4.2 锦标赛选择策略对比测试 def opt2_test(): ITE = 20 # 测试次数 i_list = range(ITE) b_list = [] # 每次求出的最短路径 t_list = [] # 每次求解的耗时 b_listII = [] t_listII = [] b_listIII = [] t_listIII = [] for i in i_list: print(i) # I. 原赌轮盘选择策略 time_start = time.time() b_list.append(min(tsp_solve(sel=1))) time_end = time.time() t_list.append(time_end - time_start) # II. 锦标赛选择策略 time_startII = time.time() b_listII.append(min(tsp_solve(sel=2))) time_endII = time.time() t_listII.append(time_endII - time_startII) # III. 锦标赛选择策略 + 两点互换变异 + 块逆转变异策略 time_startIII = time.time() b_listIII.append(min(tsp_solve(sel=2,muta=3))) time_endIII = time.time() t_listIII.append(time_endIII - time_startIII) # 做排序处理,方便比较 b_list.sort() t_list.sort() b_listII.sort() t_listII.sort() b_listIII.sort() t_listIII.sort() ax1 = plt.subplot(121) ax1.plot(i_list, b_list, 'b', label="Origin") ax1.plot(i_list, b_listII, 'r', label="Tournament") ax1.plot(i_list, b_listIII, 'g', label="Tournament + Block-reversal + Origin") ax1.set_ylabel("Shortest Path") ax2 = plt.subplot(122) ax2.plot(i_list, t_list, 'b', label="Origin") ax2.plot(i_list, t_listII, 'r', label="Tournament") ax2.plot(i_list, t_listIII, 'g', label="Tournament + Block-reversal + Origin") ax2.set_ylabel("Cost Time") plt.legend() plt.show() # 3.1 原程序的主函数 - 求解不同规模的TSP问题的算法性能 def ori_main(): time_start = time.time() pop = [] # 生成初代种群pop li = list(range(DNA_SIZE)) for i in range(POP_SIZE): random.shuffle(li) l = li.copy() pop.append(l) best_dis= [] # 进行选择,交叉,变异,并把每代的最优个体保存在best_dis中 for i in range(Iterations): # 迭代N代 pop = crossmuta(pop, CROSS_RATE) fitness = getfitness(pop) maxfitness = np.argmax(fitness) best_dis.append(distance(pop[maxfitness])) pop = select(pop, fitness) # 选择生成新的种群 time_end = time.time() print_info(pop) print('逐代的最小距离:',best_dis) print('Totally cost is', time_end - time_start, "s") plt.figure() plt.plot(range(Iterations),best_dis) # 4.1 块逆转变异策略运行效果展示 def opt1_main(): time_start = time.time() pop = [] # 生成初代种群pop li = list(range(DNA_SIZE)) for i in range(POP_SIZE): random.shuffle(li) l = li.copy() pop.append(l) best_dis= [] # 进行选择,交叉,变异,并把每代的最优个体保存在best_dis中 for i in range(Iterations): # 迭代N代 pop = crossmuta(pop, CROSS_RATE, muta=3) fitness = getfitness(pop) maxfitness = np.argmax(fitness) best_dis.append(distance(pop[maxfitness])) pop = select(pop, fitness) # 选择生成新的种群 time_end = time.time() print_info(pop) print('逐代的最小距离:',best_dis) print('Totally cost is', time_end - time_start, "s") plt.figure() plt.plot(range(Iterations),best_dis) if __name__ == "__main__": ori_main() # 原程序的主函数 opt1_main() # 块逆转变异策略运行效果展示 plt.show() plt.close() # opt1_test() # 块逆转变异策略对比测试 # opt2_test() # 锦标赛选择策略对比测试 # pop_size_test() # POP_SIZE 种群规模参数测试 # cross_rate_test() # CROSS_RATE 交叉率参数测试 # muta_rate_test() # MUTA_RATE 变异率参数测试 # cross_muta_test() # 交叉率和变异率双参数测试- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
- 59
- 60
- 61
- 62
- 63
- 64
- 65
- 66
- 67
- 68
- 69
- 70
- 71
- 72
- 73
- 74
- 75
- 76
- 77
- 78
- 79
- 80
- 81
- 82
- 83
- 84
- 85
- 86
- 87
- 88
- 89
- 90
- 91
- 92
- 93
- 94
- 95
- 96
- 97
- 98
- 99
- 100
- 101
- 102
- 103
- 104
- 105
- 106
- 107
- 108
- 109
- 110
- 111
- 112
- 113
- 114
- 115
- 116
- 117
- 118
- 119
- 120
- 121
- 122
- 123
- 124
- 125
- 126
- 127
- 128
- 129
- 130
- 131
- 132
- 133
- 134
- 135
- 136
- 137
- 138
- 139
- 140
- 141
- 142
- 143
- 144
- 145
- 146
- 147
- 148
- 149
- 150
- 151
- 152
- 153
- 154
- 155
- 156
- 157
- 158
- 159
- 160
- 161
- 162
- 163
- 164
- 165
- 166
- 167
- 168
- 169
- 170
- 171
- 172
- 173
- 174
- 175
- 176
- 177
- 178
- 179
- 180
- 181
- 182
- 183
- 184
- 185
- 186
- 187
- 188
- 189
- 190
- 191
- 192
- 193
- 194
- 195
- 196
- 197
- 198
- 199
- 200
- 201
- 202
- 203
- 204
- 205
- 206
- 207
- 208
- 209
- 210
- 211
- 212
- 213
- 214
- 215
- 216
- 217
- 218
- 219
- 220
- 221
- 222
- 223
- 224
- 225
- 226
- 227
- 228
- 229
- 230
- 231
- 232
- 233
- 234
- 235
- 236
- 237
- 238
- 239
- 240
- 241
- 242
- 243
- 244
- 245
- 246
- 247
- 248
- 249
- 250
- 251
- 252
- 253
- 254
- 255
- 256
- 257
- 258
- 259
- 260
- 261
- 262
- 263
- 264
- 265
- 266
- 267
- 268
- 269
- 270
- 271
- 272
- 273
- 274
- 275
- 276
- 277
- 278
- 279
- 280
- 281
- 282
- 283
- 284
- 285
- 286
- 287
- 288
- 289
- 290
- 291
- 292
- 293
- 294
- 295
- 296
- 297
- 298
- 299
- 300
- 301
- 302
- 303
- 304
- 305
- 306
- 307
- 308
- 309
- 310
- 311
- 312
- 313
- 314
- 315
- 316
- 317
- 318
- 319
- 320
- 321
- 322
- 323
- 324
- 325
- 326
- 327
- 328
- 329
- 330
- 331
- 332
- 333
- 334
- 335
- 336
- 337
- 338
- 339
- 340
- 341
- 342
- 343
- 344
- 345
- 346
- 347
- 348
- 349
- 350
- 351
- 352
- 353
- 354
- 355
- 356
- 357
- 358
- 359
- 360
- 361
- 362
- 363
- 364
- 365
- 366
- 367
- 368
- 369
- 370
- 371
- 372
- 373
- 374
- 375
- 376
- 377
- 378
- 379
- 380
- 381
- 382
- 383
- 384
- 385
- 386
- 387
- 388
- 389
- 390
- 391
- 392
- 393
- 394
- 395
- 396
- 397
- 398
- 399
- 400
- 401
- 402
- 403
- 404
- 405
- 406
- 407
- 408
- 409
- 410
- 411
- 412
- 413
- 414
- 415
- 416
- 417
- 418
- 419
- 420
- 421
- 422
- 423
- 424
- 425
- 426
- 427
- 428
- 429
- 430
- 431
- 432
- 433
- 434
- 435
- 436
- 437
- 438
- 439
- 440
- 441
- 442
- 443
- 444
- 445
- 446
- 447
- 448
- 449
- 450
- 451
- 452
- 453
- 454
- 455
- 456
- 457
- 458
- 459
- 460
- 461
- 462
- 463
- 464
- 465
- 466
- 467
- 468
- 469















-
相关阅读:
LeetCode 695. 岛屿的最大面积
一款基于.Net开发、开源、支持多平台云存储文件管理器
一文深度解读边缘计算产业发展前景
如何优化 Bash 脚本的执行效率?
电商直播增加人气,留人技巧有哪些
Vue3实战(1)
看完就明白/dev/sda、/dev/hda是什么了
5G+AI+XR:将成为开启空间互联网的钥匙
什么是数据描述统计分析?(Descriptive Analysis)
c#关于类,对象的例子实现
- 原文地址:https://blog.csdn.net/sheziqiong/article/details/126803212
