-
西电数据挖掘实验3——复杂网络社团检测
一、实验内容
复杂网络是描述复杂系统的有力工具,其中每个实体定义成一个节点,实体间的交互关系定义为边。复杂网络社团结构定义为内紧外松的拓扑结构,即一组节点的集合,集合内的节点交互紧密,与外界节点交互松散。复杂网络社团结构检测广泛的应用于信息推荐系统、致癌基因识别、数据挖掘等领域。
本实验利用两类数据:模拟数据与真实数据。模拟数据有著名复杂网络学者Mark Newmann所提出,该网络包括128个节点,每个节点的度为16,网络包含4个社团结构,每个社团包含32个节点,每个节点与社团内部节点有 k 1 k_1 k1个节点相互链接,与社团外部有 k 2 k_2 k2个节点相互链接 ( k 1 + k 2 = 16 k_1+k_2=16 k1+k2=16)。通过调节参数 k 2 k_2 k2( k 2 = 1 , 2 , . . . , 8 k_2=1,2,...,8 k2=1,2,...,8)增加社团构建检测难度。
真实数据集:跆拳道俱乐部数据由34个节点组成,由于管理上的分歧,俱乐部分解成两个社团。
二、分析及设计
Step1:导入网络数据
利用邻接矩阵 A A A来存储网络,其中 A i j A_{ij} Aij表示第 i i i个节点与第 j j j个节点的是否有边相互链接,1表示有,0表示没有。
在本次实验中,我利用Python中的networkx包处理网络数据。networkx能够有效地组织与管理图数据结构,并且其中封装了很多与图操作相关的函数调用,能够提高本次实验程序的编写效率。
Step2:根据网络结构特征给出节点相似性度量指标
给定节点 i i i, 其邻居节点定义为与该节点相链接的所有节点组成的集合,即 N ( i ) = { j ∣ A i j = 1 , j = 1 , 2 , . . . , n } N(i)=\{j|A_{ij}=1,j=1,2,...,n\} N(i)={j∣Aij=1,j=1,2,...,n}。给定一对节点 ( i , j ) (i,j) (i,j),其相似性定义如下:
S i j = ∣ N ( i ) ∩ N ( j ) ∣ ∣ N ( i ) ∪ N ( j ) ∣ S_{ij} = \frac{|N(i) \cap N(j)|}{|N(i) \cup N(j)|} Sij=∣N(i)∪N(j)∣∣N(i)∩N(j)∣
其中 ∣ N ( i ) ∩ N ( j ) ∣ |N(i) \cap N(j)| ∣N(i)∩N(j)∣表示集合 N ( i ) ∩ N ( j ) N(i) \cap N(j) N(i)∩N(j)中元素的个数。Step3:采用贪婪算法提取模块
随机选择一个未聚类的节点作为当前社团C,提取出社团C所有未聚类的邻居节点 N ( c ) N(c) N(c)。选择使得社团密度降低最小的那个节点 v v v添加到社团 C C C,更新当前社团为 C = C ∪ v C = C \cup v C=C∪v(若某节点已经找不到其未聚类的邻居节点,则认为该节点自成一个社团),持续该过程直到当前社团的密度小于某个阈值。当一个社团提取完成后,将其加入存放总社团的集合中,即 C l u b s = C l u b s ∪ C Clubs = Clubs \cup C Clubs=Clubs∪C。此后,再从剩余的未被分类的节点中任选一个出来作为新社团的初始节点,重复进行上述操作,直到所有节点均被归类到某一社团中,算法结束。计算过程中我使用社团中所有节点对的相似度之和除以节点对总数(组合数)再除以2来定义的社团密度,具体函数表达式如下:
D e n s i t y = ∑ i , j s i j C l e n ( c ) 2 / 2 ∈ ( 0 , 2 ) Density = \frac{\sum_{i,j} s_{ij}}{C_{len(c)}^2 / 2} \in(0,2) Density=Clen(c)2/2∑i,jsij∈(0,2)
其中 l e n ( c ) len(c) len(c)是社团 c c c中的节点个数, C l e n ( c ) 2 C_{len(c)}^2 Clen(c)2是从社团 c c c的节点中任选2个节点的组合数, i , j i,j i,j是从 c c c中任取2个节点的组合对应的节点标号。Step4:采用Cytoscape工具,可视化聚类结果
由于Cytoscape对于被导入的文件中的数据格式有一定的要求,所以我先用Python对karate.gml中的数据进行了处理,输出了符合Cytoscape导入数据规范的边数据,然后再将边数据导入txt文件中,最后导入Cytoscape。导入Cytoscape后,根据Python计算出的社团分类结果,将这34个节点分别着色,每个社团中的节点着同色,最终完成可视化操作。
三、详细实现
由于本次实验需要处理图数据,对图中节点进行相关操作,而Python中有很强大的networkx包便于我们构建和操作复杂的图结构,故我选择用Python编写本次实验的程序。具体代码实现如下(所有重要语句均已给出相应的注释):
1.先导入本实验需要用到的包:
import random import copy import numpy as np import networkx as nx import matplotlib.pyplot as plt- 1
- 2
- 3
- 4
- 5
2.定义函数show_info(),显示一下社团网络的相关信息:
# 显示图G的相关信息 def show_info(G): # 输出所有节点和所有边 print("nodes:", G.nodes(), '\n') print("edges:", G.edges(), '\n') # 输出节点总数和边总数 nodes_num = G.number_of_nodes() edges_num = G.number_of_edges() print("number of nodes:", nodes_num) print("number of edges:", edges_num)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
3.定义函数clac_s(),利用题目中给出的相似性计算公式计算相似性矩阵:
# 计算相似性矩阵 def calc_s(G): # 初始化s nodes_num = G.number_of_nodes() sim = np.zeros((nodes_num,nodes_num)) # 开始计算s for i in range(1, nodes_num + 1): for j in range(1, nodes_num + 1): # 根据定义计算i,j的相似性 sim[i-1][j-1] = len(G.adj[i].keys() & G.adj[j].keys()) / len(G.adj[i].keys() | G.adj[j].keys()) print('图G的相似性矩阵为:') print(sim) return sim- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
4.定义函数calc_density()计算社团密度:
# 计算社团密度(采用基于相似度度量的密度) def calc_density(c, s): # 求出社团中的点数和边数 v = c.number_of_nodes() e = c.number_of_edges() # 若社团中只有一个点,则密度最大,为1 if len(c) == 1: return 1.0 # 初始化总相似度 sum_sim = 0.0 for node_i in list(c.nodes()): for node_j in list(c.nodes()): # 不计算自身 if (node_i != node_j) & (node_j.__index__() > node_i.__index__()): sum_sim = sum_sim + s[node_i-1][node_j-1] # 最后除以社团c中node_i,node_j的组合数再除以系数 density_2 = sum_sim / ( ( v * (v - 1) ) / 4 ) # 最后返回社团密度 return density_2- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
5.定义函数find_nbrs()求出社团所有未聚类的邻居节点:
# 求社团未聚类的邻居节点 def find_nbrs(G, G_copy, c): # 初始化 nbrs = [] for node in list(c.nodes()): # node的邻居应该在G中找 node_nbrs = list(G.adj[node].keys()) nbrs = list(set(nbrs) | (set(node_nbrs))) # 未聚类的邻居节点应该在G_copy中找,最后以数值形式存放在列表里 final_nbrs = list(set(nbrs) & (set(list(G_copy.nodes())))) return final_nbrs- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
6.定义核心函数club_julei()利用贪心算法求解所有社团,算法思想在上文中已经阐述,算法细节见注释:
# 这里是初始化全局变量picked_node picked_node = 0 # 利用贪心算法找到所有的社团——核心函数 def club_julei(G, s, t): # 初始化列表存放所有社团列表 clubs = [] # 深拷贝G,否则对G_copy操作也会同时影响G G_copy = copy.deepcopy(G) # 当G_copy的节点列表不为空时循环寻找社团,G_copy节点列表为空时说明所有的社团均已被找到 while(G_copy.nodes()): # 注意列表下标从0开始 c = nx.Graph() idx = random.randint(0, len(G_copy)-1) # 随机选择一个G_copy中的节点初始化社团c(G_copy中节点都是未被聚类的) randpick_node = list(G_copy.nodes())[idx] c.add_node(randpick_node) # 从G_copy中删去该节点 G_copy.remove_node(randpick_node) # 死循环构造社团直到社团密度小于阈值 while(1): # 计算原始社团密度 density_old = calc_density(c, s) # 求当前社团未聚类的邻居节点 candinodes = find_nbrs(G, G_copy, c) if len(candinodes) == 0: clubs.append(list(c.nodes())) break minval = 1.0 global picked_node # 全局变量picked_node # 寻找使社团密度降低最小的那个节点 for node in candinodes: c.add_node(node) # 计算加入该节点后的社团密度 density_new = calc_density(c, s) # 计算社团密度减小量 dec = density_old - density_new # 若dec<minval,更新数据 if dec < minval: minval = dec picked_node = node # 测试完当前结点后删除该节点,再循环测下一个 c.remove_node(node) # 循环结束后picked_node已找到,将该节点加入社团c # 这里判断是否为0是为了应对picked_node未被赋值的情况 if picked_node == 0: continue else: c.add_node(picked_node) # 同时在G中删除该点,因为该点已被分类 if picked_node in list(G_copy.nodes()): G_copy.remove_node(picked_node) # 若当前社团的密度小于阈值,则将其加入到clubs列表中 if calc_density(c, s) < t: #print(calc_density(c, s)) clubs.append(list(c.nodes())) break # 返回所有社团 return clubs- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
四、实验结果
社团聚类结果随着社团密度阈值选取的不同(0.2, 0.4, 0.5)而变化的情况如下图所示:

我用Cytoscape绘制了阈值t分别取0.2,0.4,0.5时的社团聚类结果图(其余阈值下画法类似,不再展示结果),效果如下:t = 0.2时的社团聚类结果:
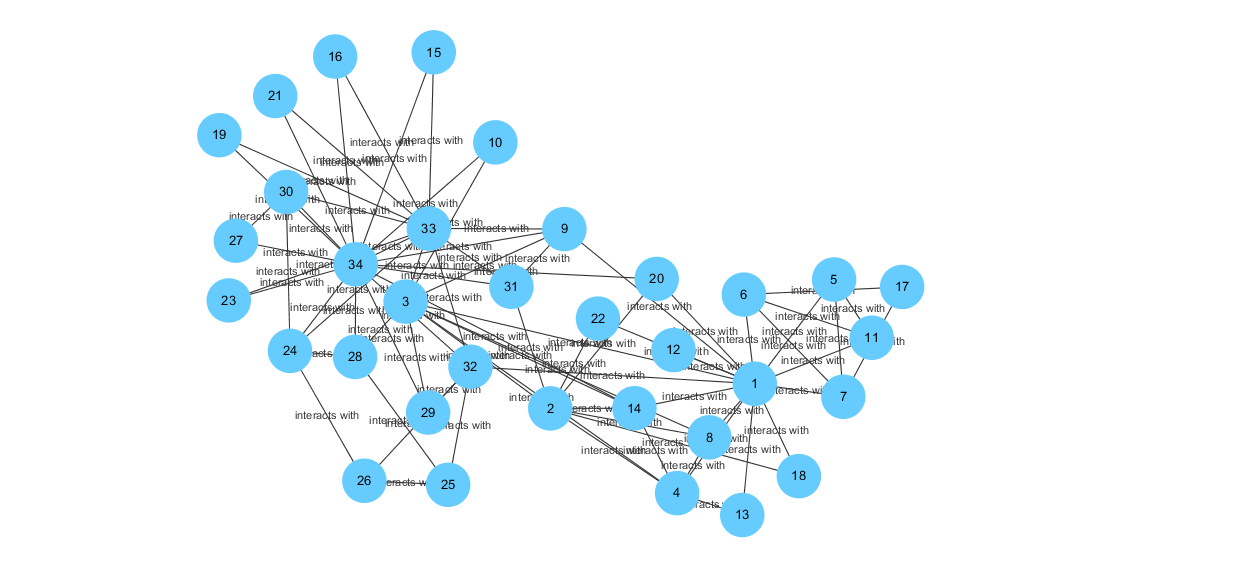
t = 0.4时的社团聚类结果:

t = 0.5时的社团聚类结果:

分析上图可知,当阈值t = 0.4左右时,社团聚类的效果最好。当密度阈值设定为一个比较小的数时(如小于0.1),所有节点被分到同一个社团中;当密度阈值稍微增大后(如0.2-0.5),将会出现更多的社团,当密度阈值取到比较大的值后(如大于0.6),基本上一个社团中只存在两三个节点。当然,即使在同一阈值下,每次运行程序得到的社团聚类结果也是不同的,这是因为每次随机选取的初始节点不同,最终合成的社团也会不同。
在实验中,我尝试过用基本的图密度定义: d e n s i t y = 2 ∣ E ∣ ∣ V ∣ ⋅ ∣ V − 1 ∣ density = \frac{2|E|}{|V|·|V-1|} density=∣V∣⋅∣V−1∣2∣E∣去计算社团的密度,也尝试过用图密度+基于相似性的密度去计算社团密度,但是后来发现这两种方法划分社团的效果并不理想,具体表现为当阈值还不是很大(如0.4)时就已经划分出很多小社团了,不符合我们的预期。
-
相关阅读:
免费版Photoshop2024智能人像磨皮插件
flink 复postgresql数据库数据
云架构(五)BBF模式
MySQL中对varchar类型的列进行统计分析
[Vue3:组件通信)子组件props接收和watch监听,emit发送父组件 (添加修改设置成绩,添加、删除选课记录)
Integer、Long 等包装类 == 值判断、地址判断与缓存
C++项目——云备份-④-服务端配置信息模块设计与实现
shell脚本数组
docker的使用以及注意事项
非零基础自学Java (老师:韩顺平) 第7章 面向对象编程(基础部分) 7.2 成员方法
- 原文地址:https://blog.csdn.net/qq_45717425/article/details/125480880
