-
良/恶性乳腺肿瘤预测(逻辑回归分类器)
案例:良/恶性乳腺肿瘤预测
1.1 简介
本案例使用逻辑回归分类器对乳腺肿瘤进行良性/恶性预测,并对预测模型进行指标测算与评价。
这里数据集采用乳腺癌数据集,原始的数据集下载地址为:https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/breast-cancer-wisconsin.data。
数据特征如下:

1.2 代码
将每个属性的特征量化为1~10的数值进行表示。首先,导入数据并显示前5条数据。
1.2.1 导入数据集
import pandas as pd import numpy as np column_names=['number','Cl_Thickness','Unif_cell_size','Unif_cell_shape','Marg_Adhesion','Sing_epith_cell_size','Bare_nuclei','Bland_chromation','Norm_nuclei','Mitoses','Class'] data=pd.read_csv('breast-cancer-wisconsin.data',names=column_names) display(data.head())- 1
- 2
- 3
- 4
- 5

1.2.2 浏览数据的基本信息
data.info()- 1

1.2.3 查看数据的基本统计信息
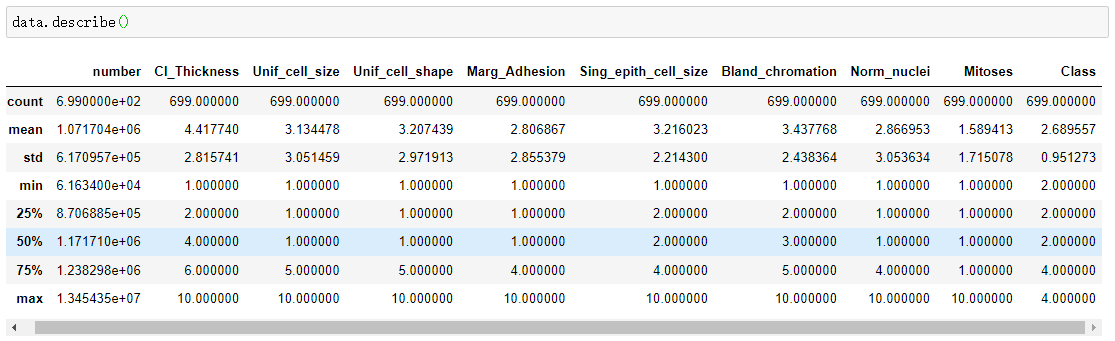
data.describe()- 1
1.2.4 统计数据属性中的缺失值
data.isnull().sum()- 1

如果存在缺失数据,需要丢弃或填充。该数据集中并没有缺失值。这里我们采取删除缺失值的方法
data=data.replace(to_replace='?',value=np.nan) data=data.dropna(how='any') print(data.shape)- 1
- 2
- 3

1.2.5 将数据集划分为训练集和测试集
from sklearn.model_selection import train_test_split # 划分训练集与测试集 X_train,X_test,y_train,y_test=train_test_split(data[column_names[1:10]],data[column_names[10]],test_size=0.25,random_state=33) print('训练样本的数量和类别分布:\n',y_train.value_counts())- 1
- 2
- 3
- 4

1.2.6 标准化数据
每个维度的特征数据方差为1,均值为0,使得预测结果不会被某些维度过大的特征值主导。
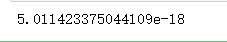
from sklearn.preprocessing import StandardScaler ss=StandardScaler() X_train=ss.fit_transform(X_train) X_test=ss.transform(X_test) print(X_train.mean())- 1
- 2
- 3
- 4
- 5
1.2.7 分别用LogisticRegression与SGDClassifier构建分类器
from sklearn.linear_model import LogisticRegression from sklearn.linear_model import SGDClassifier lr=LogisticRegression() sgdc=SGDClassifier() lr.fit(X_train,y_train) lr_y_predict=lr.predict(X_test) sgdc.fit(X_train,y_train) sgdc_y_predict=sgdc.predict(X_test)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
1.2.8 分析LR分类器性能
from sklearn.metrics import classification_report print('Accuracy of LR Classifier:',lr.score(X_test,y_test)) print(classification_report(y_test,lr_y_predict,target_names=['Benign','Malignant']))- 1
- 2
- 3

1.2.9 SGD分类器性能分析
print('Accuracy of SGD Classifier:',sgdc.score(X_test,y_test)) print(classification_report(y_test,sgdc_y_predict,target_names=['Benign','Malignant'])) # print(classification_report(y_test,sgdc_y_predict))- 1
- 2
- 3
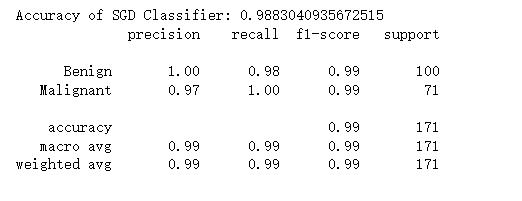
precision 精确率
recall 召回率
f1_score F1值
macro avg 宏观平均值
weighted avg 加权平均值
-
相关阅读:
使用反射拼接SQL语句 和 使用 反射 + 注解 拼接SQL语句
Unity中实现HybridCLR热更新
开箱即用的Appimage是什么以及如何建立快捷方式
STC15单片机-数码管显示PCB板温度(TM1620驱动芯片使用介绍)
【vue2第十一章】v-model的原理详解 与 如何使用v-model对父子组件的value绑定 和修饰符.sync
记录wisemodel上传失败
opencv边缘-边界处理
FDM3D打印系列——Blue Mary
启动-异常学习笔记-代码导读
2022年Java秋招面试必看的 | ZooKeeper面试题
- 原文地址:https://blog.csdn.net/qq_43753724/article/details/125472866