-
数据集 | 基于计算机视觉的医学影像处理数据集
计算机视觉分支-医学影响相关数据集概况。
2D图像数据集
1.1.MIMIC-III (The Medical Information Mart for Intensive Care III)
重症监护医疗信息集市 III ( MIMIC-III ) 数据集是一个大型、去标识化和公开可用的医疗记录集合。数据集中的每条记录都包含 ICD-9 代码,用于识别诊断和执行的程序。每个代码都被划分为子代码,这些子代码通常包括特定的环境细节。该数据集由 112,000 条临床报告记录(平均长度 709.3 个标记)和 1,159 个顶级 ICD-9 代码组成。每个报告平均分配到 7.6 个代码。数据包括生命体征、药物、实验室测量值、护理提供者绘制的观察结果和注释、体液平衡、程序代码、诊断代码、成像报告、住院时间、生存数据等。
1.2.CheXpert
大型标记数据集推动了深度学习方法在各种医学成像任务上实现专家级性能。我们展示了 CheXpert,这是一个包含 65,240 名患者的 224,316 张胸片的大型数据集。我们设计了一个标记器来自动检测放射学报告中是否存在 14 个观察结果,从而捕获射线照片解释中固有的不确定性。我们研究了使用不确定性标签训练卷积神经网络的不同方法,这些网络在给定可用的正面和侧面射线照片的情况下输出这些观察的概率。在由 3 名董事会认证的放射科医师手动注释的 200 项胸部放射学研究的验证集上,我们发现不同的不确定性方法对不同的病理有用。然后,我们在一个测试集上评估我们的最佳模型,该测试集由 500 个胸部放射学研究组成,由 5 位董事会认证的放射科医师共识注释,并将我们的模型与另外 3 位放射科医师在检测 5 种选定病理方面的表现进行比较。在心脏肥大、水肿和胸腔积液中,模型 ROC 和 PR 曲线位于所有 3 个放射科医师操作点之上。我们将数据集作为标准基准向公众发布,以评估胸片判读模型的性能。该数据集可在 https://stanfordmlgroup.github.io/competitions/chexpert 免费获得。在心脏肥大、水肿和胸腔积液中,模型 ROC 和 PR 曲线位于所有 3 个放射科医师操作点之上。我们将数据集作为标准基准向公众发布,以评估胸片判读模型的性能。该数据集可在 https://stanfordmlgroup.github.io/competitions/chexpert 免费获得。在心脏肥大、水肿和胸腔积液中,模型 ROC 和 PR 曲线位于所有 3 个放射科医师操作点之上。我们将数据集作为标准基准向公众发布,以评估胸片判读模型的性能。该数据集可在 https://stanfordmlgroup.github.io/competitions/chexpert 免费获得。
1.3.DRIVE(用于血管提取的数字视网膜图像)
用于血管提取的数字视网膜图像( DRIVE ) 数据集是用于视网膜血管分割的数据集。它由总共 40 张 JPEG 彩色眼底图像组成;其中病理异常7例。这些图像来自荷兰的糖尿病视网膜病变筛查项目。图像是使用佳能 CR5 非散瞳 3CCD 相机获得的,FOV 等于 45 度。每个图像分辨率为 584*565 像素,每个颜色通道(3 个通道)有 8 位。
这组 40 幅图像平均分为 20 幅图像作为训练集和 20 幅图像作为测试集。在这两个集合中,对于每个图像,都有一个直径约为 540 像素的圆形视场 (FOV) 掩模。在训练集中,对于每张图像,已经应用了由眼科专家进行的一次手动分割。在测试集中,对于每个图像,两个不同的观察者应用了两个手动分割,其中第一个观察者分割被接受为性能评估的基本事实。
数据集下载地址:Introduction - Grand Challenge
数据
25_training:色素上皮改变,可能是蝶形黄斑病变伴中央凹色素性瘢痕,或脉络膜病变,无糖尿病视网膜病变或其他血管 异常。
26_training:背景糖尿病视网膜病变、色素上皮萎缩、视盘周围萎缩
32_training:背景糖尿病视网膜病变
03_test:背景糖尿病视网膜病变
08_test:色素上皮变化、中央凹色素性瘢痕或脉络膜病变、无糖尿病视网膜病变或其他血管 异常14_test:背景糖尿病视网膜病变 17_test:背景糖尿病视网膜病变每个图像都经过 JPEG 压缩。
使用具有 45 度视场 (FOV) 的佳能 CR5 非散瞳 3CCD 相机获取图像。每个图像是使用 8 位每个颜色平面以 768 x 584 像素捕获的。每个图像的 FOV 是圆形的,直径约为 540 像素。对于此数据库,图像已在 FOV 周围裁剪。对于每个图像,都提供了一个描述 FOV 的掩码图像。
这组 40 张图像分为训练集和测试集,均包含 20 张图像。对于训练图像,可以对脉管系统进行单次手动分割。对于没有注释的测试用例,您将能够将您的预测提交到该站点并将它们与黄金标准进行比较。此外,每个视网膜图像都有一个掩模图像,指示感兴趣的区域。所有手动分割脉管系统的人类观察者均由经验丰富的眼科医生指导和培训。他们被要求标记他们至少 70% 确定它们是容器的所有像素。



1.4.fastMRI(快速磁共振成像)
fastMRI数据集包括两种类型的 MRI 扫描:膝关节 MRI 和大脑(神经)MRI,并包含训练、验证和掩蔽测试集。NYU Langone 提供的去识别成像数据集包含几个子数据集组中的原始 k 空间数据。这些数据的管理是 IRB 批准的研究的一部分。原始数据和 DICOM 数据已分别通过转换为与供应商无关的 ISMRMD 格式和 RSNA 临床试验处理器进行去识别化。此外,通过对元数据和图像内容的抽查,手动检查每个 DICOM 图像是否存在任何意外的受保护健康信息 (PHI)。 膝关节核磁共振:从 3 和 1.5 特斯拉磁体获得的 1,500 多个完全采样的膝关节 MRI 的数据和来自 3 或 1.5 特斯拉的 10,000 个临床膝关节 MRI 的 DICOM 图像的数据。原始数据集包括有和没有脂肪抑制的冠状质子密度加权图像。DICOM 数据集包含带和不带脂肪抑制的冠状质子密度加权、带脂肪抑制的轴向质子密度加权、矢状质子密度和带脂肪抑制的矢状 T2 加权。 脑核磁共振成像:来自 6,970 个完全采样的脑核磁共振成像数据,这些数据是在 3 和 1.5 特斯拉磁体上获得的。原始数据集包括轴向 T1 加权、T2 加权和 FLAIR 图像。一些 T1 加权采集包括造影剂的入院。
1.5.ChestX-ray14
ChestX-ray14是一个医学成像数据集,包含 30,805 名(从 1992 年到 2015 年收集的)独特患者的 112,120 张正面 X 射线图像,具有文本挖掘的 14 个常见疾病标签,这些标签是通过 NLP 从文本放射学报告中挖掘出来的技巧。它通过增加六种额外的胸部疾病来扩展 ChestX-ray8:水肿、肺气肿、纤维化、胸膜增厚和疝气。
1.6.LIDC-IDRI
LIDC -IDRI数据集包含来自四位经验丰富的胸部放射科医师的病变注释。LIDC-IDRI 包含来自 1010 名肺部患者的 1018 份低剂量肺部 CT。
1.7.CORD-19
CORD-19 是一个免费资源,包含数万篇关于 COVID-19、SARS-CoV-2 和相关冠状病毒的学术文章,供全球研究界使用。
1.8.BraTS 系列
BraTS 2015
BraTS 2015 数据集是用于脑肿瘤图像分割的数据集。它由 220 个高级别胶质瘤 (HGG) 和 54 个低级别胶质瘤 (LGG) MRI 组成。四种 MRI 方式是 T1、T1c、T2 和 T2FLAIR。分段的“基本事实”提供了大约四种肿瘤内类别,即水肿、增强肿瘤、非增强肿瘤和坏死。
BraTS 2017
BRATS2017 数据集。它包含 285 次脑肿瘤 MRI 扫描,每次扫描有四种 MRI 模式,即 T1、T1ce、T2 和 Flair。该数据集还提供脑肿瘤的完整掩码,带有 ED、ET、NET/NCR 的标签。分割评估基于三个任务:WT、TC 和 ET 分割。
BraTS 2018
BraTS 2018是一个数据集,提供由医生注释的多模态 3D 脑 MRI 和地面实况脑肿瘤分割,每个病例由 4 种 MRI 模态(T1、T1c、T2 和 FLAIR)组成。注释包括 3 个肿瘤亚区——增强肿瘤、瘤周水肿、坏死和非增强肿瘤核心。注释被组合成 3 个嵌套的子区域——整个肿瘤 (WT)、肿瘤核心 (TC) 和增强肿瘤 (ET)。使用各种 MRI 扫描仪从 19 个机构收集数据
1.9.STARE(视网膜结构分析)
STARE(视网膜结构化分析)数据集是用于视网膜血管分割的数据集。它包含 20 个相同大小 (700×605) 的彩色眼底图像。对于每个图像,提供了两组注释。
1.10.GENIA
GENIA语料库是在 GENIA 项目范围内编译和注释的生物医学文献的主要集合。该语料库的创建是为了支持分子生物学领域信息提取和文本挖掘系统的开发和评估。
该语料库包含 1,999 个 Medline 摘要,使用 PubMed 查询选择了三个 MeSH 术语“人类”、“血细胞”和“转录因子”。语料库已经用不同级别的语言和语义信息进行了注释。
GENIA 语料库和相应子语料库中的主要注释类别是:
- 词性注释
- 选区(短语结构)句法注释
- 术语注释
- 事件注释
- 关系注解
- 共指注解
1.11.LUNA(胸部 CT )
LUNA挑战赛使用最大的公开可用的胸部 CT 扫描参考数据库 LIDC-IDRI 数据集为自动结节检测算法提供数据集。在LUNA16中,参与者开发他们的算法并在以下两个轨道之一上传他们对 888 CT 扫描的预测:1)应开发完整 CAD 系统的完整结节检测轨道,或 2)提供的一组假阳性减少轨道的结节候选者应分类。
1.12.HAM10000(皮肤病变)
HAM10000是一个包含 10000 张训练图像的数据集,用于检测色素性皮肤病变。作者收集了来自不同人群的皮肤镜图像,通过不同的方式获取和存储。
1.13.LUNA16(肺结节分析)
LUNA16 (肺结节分析)数据集是用于肺分割的数据集。它由 1,186 个肺结节组成,在 888 次 CT 扫描中进行了注释。
1.14.Kvasir-SEG(胃肠息肉图像)
Kvasir-SEG 是一个开放获取的胃肠息肉图像和相应分割掩码的数据集,由医生手动注释,然后由经验丰富的胃肠病学家验证。
Kvasir-SEG 数据集(大小 46.2 MB)包含 1000 张息肉图像。Kvasir-SEG 中包含的图像的分辨率从 332x487 到 1920x1072 像素不等。图像及其相应的蒙版存储在两个具有相同文件名的单独文件夹中。图像文件使用JPEG压缩进行编码。
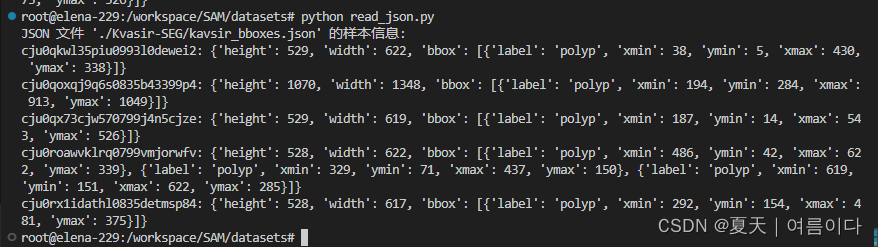
相应图像的边界框(坐标点)存储在 JSON 文件中。
下载命令
- # 下载
- wget https://datasets.simula.no/downloads/kvasir-seg.zip
- # 解压
- unzip kvasir-seg.zip
文件格式

标签文件是json格式
1.15.Medical Segmentation Decathlon
GoogleDrive下载地址【Link】
这里只下载任务一(7.1G)做示范

解压
tar -xvf Task01_BrainTumour.tar

1.16.ChestX-ray8
ChestX-ray8是一个医学成像数据集,包含 32,717 名(从 1992 年到 2015 年收集的)独特患者的 108,948 张正面 X 射线图像,具有文本挖掘的八个常见疾病标签,通过 NLP 从文本放射学报告中挖掘出来技巧。
论文地址:2017 https://arxiv.org/pdf/1705.02315v5.pdf
数据集下载地址:https://nihcc.app.box.com/v/ChestXray-NIHCC
1.17.Bitewing Radiology 数据集
龋齿是一种可传播的牙齿细菌性疾病,会破坏牙齿的结构。牙医主要根据 X 光片来诊断和治疗龋齿。虽然龋齿是一种疾病过程,但该术语通常用于描述射线照相射线可透性。射线照相检查可以提高对龋齿脱矿的最早迹象的检测和诊断。自动龋病检测技术为牙科医生提供潜在的诊断数据,并帮助识别各种疾病的迹象。然而,用于放射照相龋齿诊断的准确和客观的方法却很少被探索。因此,这项挑战的目的是研究在咬翼射线照相中检测龋齿的可能的自动化方法。从 120 名患者中收集了 120 张咬翼图像。在第 1 阶段,我们将在研讨会前发布 40 张带有地面实况数据的图像作为训练数据和 40 张图像作为测试1 数据。在第 2 阶段,将发布 40 张 Test2 数据图像用于现场比赛。
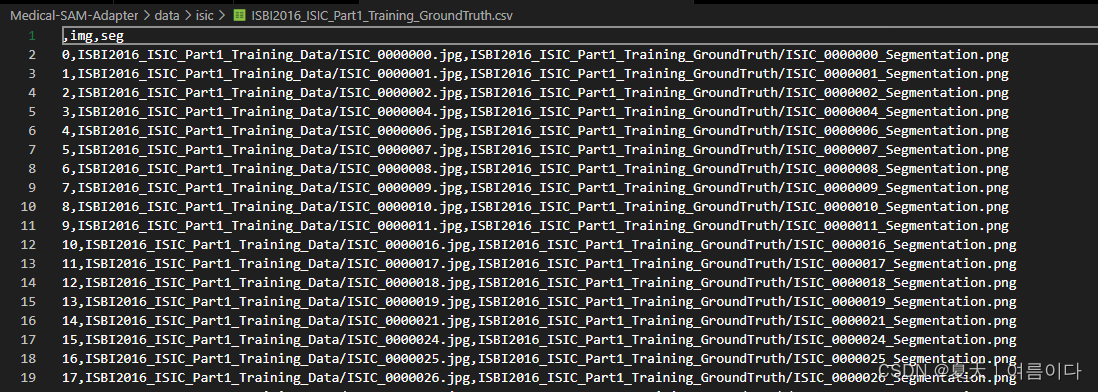
1.18.ISIC Challenge
官网:ISIC Challenge (isic-archive.com)
数据下载: ISIC Challenge
国际皮肤成像合作组织 (ISIC) 是一个 改善黑色素瘤诊断的国际努力,由 国际皮肤数字成像学会 (ISDIS)
- #下载数据集
- wget https://isic-challenge-data.s3.amazonaws.com/2016/ISBI2016_ISIC_Part1_Training_Data.zip
- #wget https://isic-challenge-data.s3.amazonaws.com/2016/ISBI2016_ISIC_Part1_Training_GroundTruth.zip
- wget https://isic-challenge-data.s3.amazonaws.com/2016/ISBI2016_ISIC_Part1_Test_Data.zip
- #wget https://isic-challenge-data.s3.amazonaws.com/2016/ISBI2016_ISIC_Part1_Test_GroundTruth.zip
- #下载csv文件
- https://github.com/KidsWithTokens/MedSegDiff/blob/master/data/isic_csv/ISBI2016_ISIC_Part3B_Test_GroundTruth.csv
- https://github.com/KidsWithTokens/MedSegDiff/blob/master/data/isic_csv/ISBI2016_ISIC_Part3B_Training_GroundTruth.csv
2.3D数据集
2.1.3D-ircadb-01
3D-IRCADb-01 数据库由 10 名女性和 10 名男性 75% 的肝肿瘤患者的 3D CT 扫描组成。 20个文件夹对应20个不同的患者,可以单独下载也可以联合下载。下表提供了图像信息,例如肝脏大小(宽度、深度、高度)或根据 Couninaud 分割的肿瘤位置。它还表明肝脏分割软件可能遇到的主要困难是由于与邻近器官的接触、肝脏的非典型形状或密度,甚至图像中的伪影。
这些文件夹被称为“ 3D-IRCADb-1- number ”( 数字 在 01 和 20 之间变化)。这些文件夹被称为“ 3D-IRCADb- 01- number ”( 数字 在 01 和 20 之间变化)。每个“ 3D-IRCADb-01- number ”文件夹包含 4 个子文件夹,分别称为“ PATIENT_DICOM ”、“ LABELLED_DICOM ”、“ MASKS_DICOM ”和“ MESHES_VTK ””。这些文件夹分别包含 DICOM 格式的匿名患者图像、对应于 DICOM 格式分割的各个感兴趣区域的标记图像、一组新的子文件夹,这些子文件夹对应于包含 DICOM 图像的各个分割感兴趣区域的名称每个掩码,最后是与 VTK 格式的各个感兴趣区域的表面网格对应的所有文件。
论文地址:http://www-sop.inria.fr/geometrica/events/wam/abstract-ircad.pdf
数据集下载地址:Liver segmentation – 3D-ircadb-01 - IRCAD
下载压缩后如图:

每个文件夹里包含
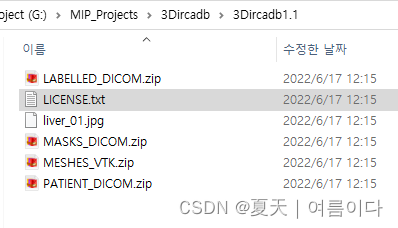
- LABLLED_DICOM.ZIP:
- LICENSE.txt
- liver_01.jpg

- MASKS_DICOM.zip
- MESHES_VTK.ZIP
- PATIENT_DICOM.zip
.vtk结尾的:VTK是使用C++编写的,一种3D文件类型
2.2.BTCV
扩展
【A1】查看json文件前五列代码
- import json
- def print_json_sample(file_path, num_lines=5):
- try:
- with open(file_path, 'r', encoding='utf-8') as file:
- json_data = json.load(file)
- print(f"JSON 文件 '{file_path}' 的样本信息:")
- # 打印文件中的前 num_lines 行
- for i, (key, value) in enumerate(json_data.items()):
- if i >= num_lines:
- break
- print(f"{key}: {value}")
- except FileNotFoundError:
- print(f"文件 '{file_path}' 未找到.")
- except json.JSONDecodeError:
- print(f"无法解码 JSON 文件 '{file_path}'. 请确保文件格式正确.")
- # 用法示例
- file_path = './Kvasir-SEG/kavsir_bboxes.json'
- print_json_sample(file_path)
参考文献
【1】BTCV 数据集介绍 - 知乎 (zhihu.com)
17.Soler, L., A. Hostettler, V. Agnus, A. Charnoz, J. Fasquel, J. Moreau, A. Osswald, M. Bouhadjar, and J. Marescaux. “3D image reconstruction for comparison of algorithm database: A patient specific anatomical and medical image database.” IRCAD, Strasbourg, France, Tech. Rep (2010)
-
相关阅读:
JavaScript:生成器函数
安全协议缺陷
java毕业生设计宠物店管理系统计算机源码+系统+mysql+调试部署+lw
Java操作k8s api示例:使用kubeconfig文件认证;获取所有pod;获取pod内应用容器的启动日志
《计算机导论》课程学习笔记
利用互斥锁解决缓存击穿问题
Python与mqtt的数据读取
火山引擎ByteHouse:如何为OLAP设计高性能向量检索能力?
AJAX 入门笔记
八股文之jdk源码分析
- 原文地址:https://blog.csdn.net/weixin_44649780/article/details/127330818
