-
新视野——宏病毒组ssDNA Virome
噬菌体、病毒研究者的烦恼
1. 病毒与噬菌体分离麻烦,宿主污染严重,数据浪费
2. 基因注释信息缺失,大量未知功能的ORF
3. 想开展溶原性、烈性噬菌体研究,不知如何下手
4. 噬菌体作为一种天然的病原细菌抗生素,前景广阔,是否存在大规模筛选工具?
问题一堆一堆,何处入手?
我们以一篇宏病毒文献为例,寻找破局之道
分为四步

1、抓基础、重基础:收集现有数据
国内大部分研究者对于生物数据库的理解都停留在使用层面,例如常见的NCBI BLAST页面
然而,我们常常忽视数据库整体的状态更新与特色。想建大厦,从基础抓起!
研究的第一步在于收集已有数据,并对已有的病毒完成图进行分类统计。
(1)NCBI RefSeq:< 9000病毒完成图
(2)ICTV:病毒物种分类国际权威数据库

2、找研究重点与Unique Idea
核心思路:自我简化
从上图统计可以看到,按照各种分类标准,病毒可以分为很多类型。此时,如果想一网打尽,那么几乎没有合适的研究手段,很多人在这里被卡住了。。。
简化方式:
(1)只关注带有衣壳蛋白的病毒——表面活性剂可以富集
(2)只关注单链环状DNA病毒——外切酶除去双链DNA,RNA酶去除所有RNA,剩余环状ssDNA后,样品组成得到极大的简化
(3)phi29特异性地对ssDNA进行滚环扩增,获得双链DNA,可以直接进行NGS测序
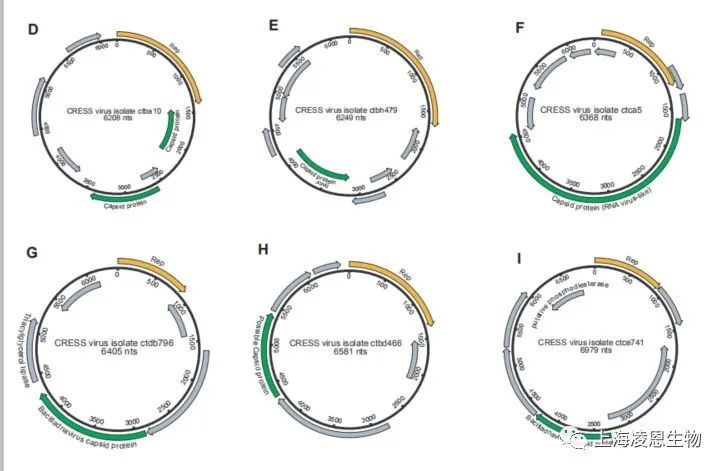
MetaSPades组装后,获得>2500个单链环状DNA完成图,绝大部分都小于10kb。

3、高级分析
高级分析不能贪多,国内很多研究者初期都有非常棒的研究结果。有时为了追求完美,单纯为了增加研究内容,会错失时机,同时也会严重降低文章研究主旨,得不偿失。
(1)用衣壳蛋白的差异性进行物种分类
(2)物种分类重新统计数据
(3)寻找以上两者关联
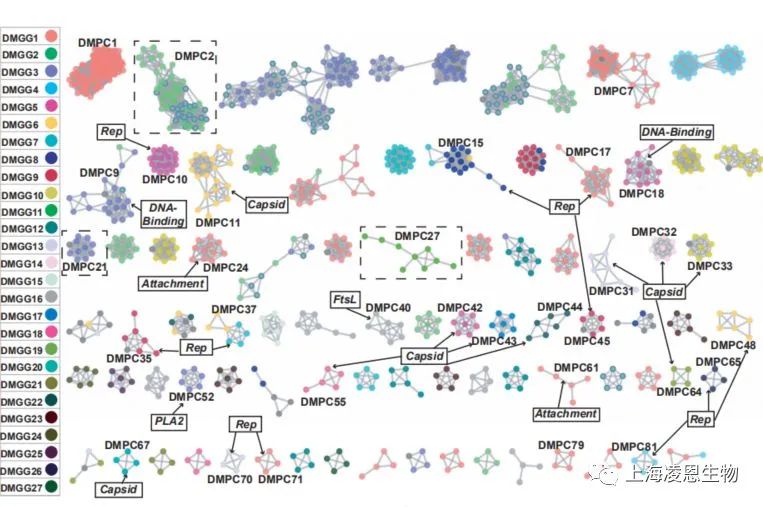

4、主题升华
宏基因组公共数据特别多,找几个作为案例证明:
(1)分析流程的可靠性
(2)获得的2500个完成图存在的广泛性


彩蛋——笔者感想
回帖率:宏基因数据中,reads回帖率比较低,即组装获得的序列还是非常不完整。
在BGI最新研究成果的帮助下,依靠培养组学的技术,暴力解析了kb级别的细菌基因组后,肠道回帖率从50%增加到70%以上,是个非常大的进步。
然而,环境样品没有这么好的运气可以如此暴力破解,因此该研究的意义特别巨大。
我们可以通过实验技术的简化,针对性地解析部分物种,进而获得全貌。日拱一卒无有尽,功不唐捐终入海
欢迎一起讨论关于病毒、宏病毒研究方案!
参考文献:Discovery of several thousand highly diverse circular DNA viruses. bioRxiv, 2019.
-
相关阅读:
决策树实验分析(分类和回归任务,剪枝,数据对决策树影响)
论信息系统项目的整体管理
pytest测试报告邮件发送格式调整(基于Allure的测试报告)
1、k8s问题pod从service中剔除
32【源码】数据可视化:基于 Echarts + Java SpringBoot 动态实时大屏范例 - 监管系统
第三方软件测评单位可为企业带来哪些收益?
ubuntu 安装docker-compose
【ElasticSearch】基于Docker 部署 ElasticSearch 和 Kibana,使用 Kibana 操作索引库,以及实现对文档的增删改查
mac安装hadoop3.2.4
Geom2d_TrimmedCurve解析
- 原文地址:https://blog.csdn.net/SHANGHAILINGEN/article/details/125440657