-
【无标题】
宏基因组组装技术的不断进步,极大促进了不可培养微生物的基因组研究,而基于宏数据组装的基因组,由于其完整性不足和存在嵌合等问题,也一直备受学界争议。最近,PacBio推出的HiFi reads高质量测序技术在这一领域研究中获得了突破进展(以下简称为HiFi-Meta技术)。

研究重点报道了利用PacBio HiFi Reads测序技术开展的基于5例人类粪便样本组装获得的102个的完整单菌基因组(cMAGs)。并在此基础上,对其组装结果的数量、完整性、数据质量、基因组大小等方面进行组装结果的评价。
01、组装效果比较
hifiasm-meta组装效果最好,是目前最佳的三代宏基因组组装软件,毫无争议。
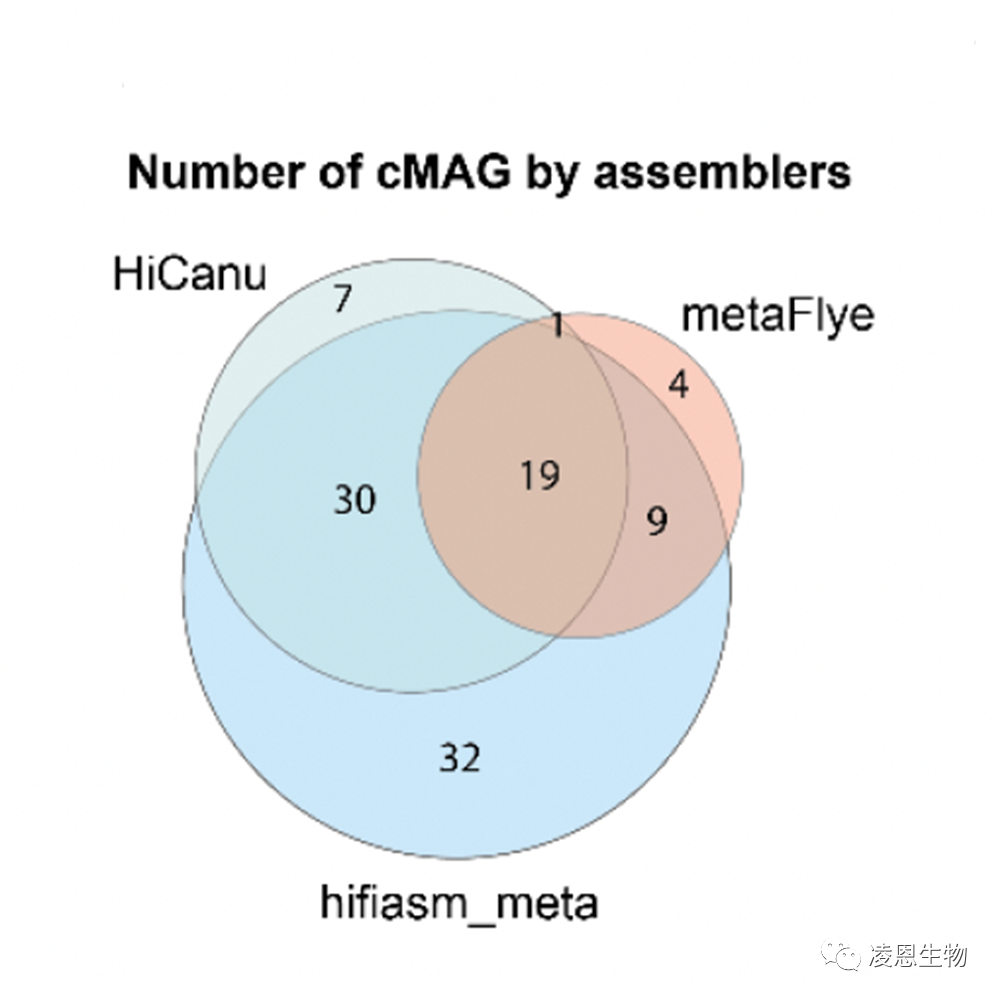
02、HiFi-Meta技术组装效果显著优于Nanopore+NGS研究策略
利用13个人肠道样本,Nanopore+NGS策略仅组装获得6个cMAGs,而基于HiFi-Meta策略,更少的样本量获得的组装结果超出5倍!!

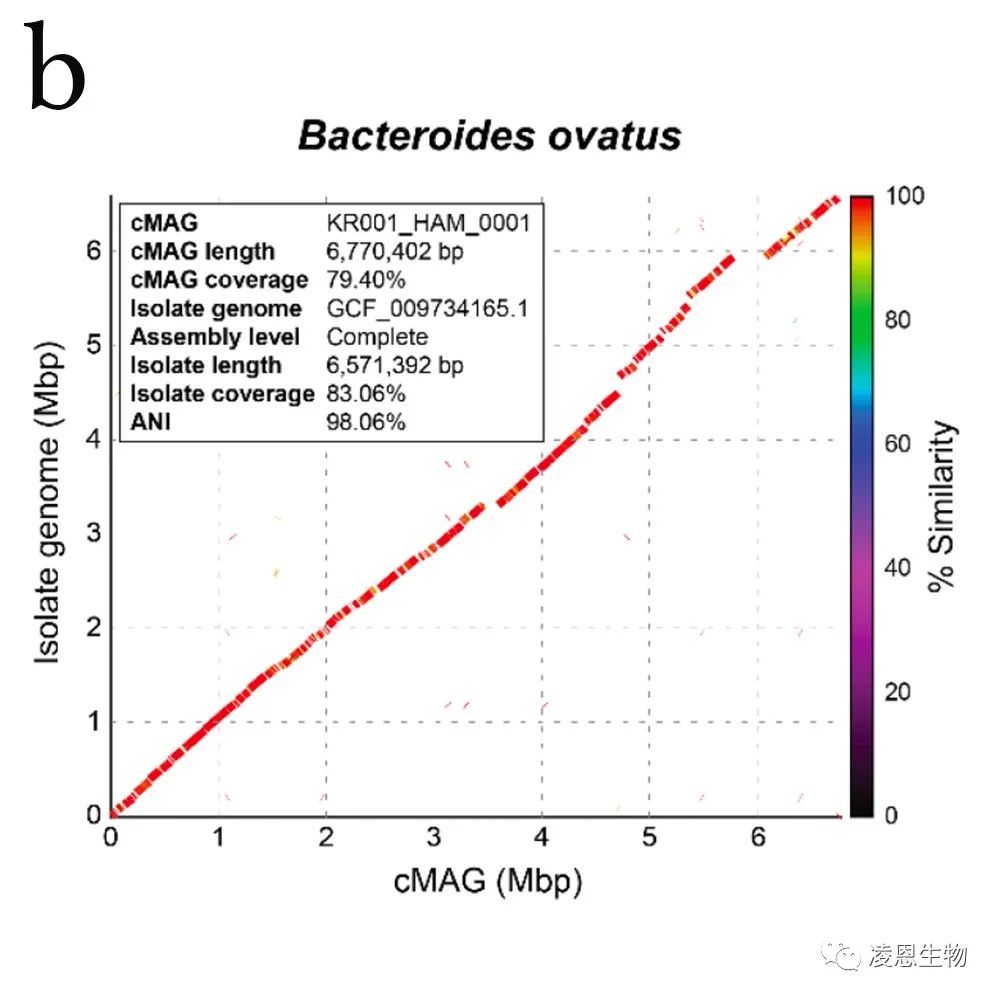
03、HiFi-Meta技术获得cMAGs,完成图质量更优
如下a图为基于宏样本HiFi-Meta组装获得的cMAG与成功分离的单菌的基因组的共线性,组装的准确度近100%。b图为无法分离培养获得的物种基于HiFi-Meta组装的结果与公开近缘菌株的共线性——显著可见,cMAGs的组装准确度完全可以满足单菌比较基因组的分析要求。

04、cMAGs覆盖的物种丰度
基于HiFi-Meta技术组装获得cMAGs中可以获得一些超过6Mb的cMAGs,物种范围涵盖了多个类群;同时,在此过程中首次报道了两大类不可培养的微生物类别(目水平)RF39及TANB77(如下图示),这两个大类此前无相关基因组报告——说明了PacBio HiFi reads测序在宏样本中不仅利于较大型单菌基因组的组装,更适合于宏样本中不能分离培养的新物种的发现及研究。

05、重要关键基因元件
利用宏基因组NGS数据进行MAGs组装时,受限于读长限制,往往是Contigs太短、基因不完整、基因簇破碎,让研究者空有雄心壮志,但又无可奈何陷入巧妇难为无米之炊的窘境。基于HiFi-Meta技术获得的cMAGs则可以轻松跨越基因组岛和rRNA区域,完美囊括NGS测序无法覆盖到的区域,为宏基因组功能研究提供更好的数据支持。

结论:基于PacBio平台的HiFi-Meta测序技术将更有助于宏样本中的单菌(包括不可分离培养的物种的)基因组的完整、精确组装。这篇文章仅针对人类肠道样本进行了测试,各位看官还在等什么呢?抢占先机,制胜科研,HiFi-Meta就找凌恩生物!
-
相关阅读:
基于瞬时频率的语言信号清/浊音判决和高音检测(MATLAB R2021)
Java笔记【方法、数组】
前端项目中,强缓存和协商缓存的配置
OPENCV实战分水岭法二
如何正确计算导入Excel的行数(POI/NPOI)
程序员关于技术应当如何做好职业规划的问题?
vue中FileReader 的使用
直线段扫描算法
信息安全实验二 :使用X-SCANNER扫描工具
Python uWSGI 安装配置
- 原文地址:https://blog.csdn.net/SHANGHAILINGEN/article/details/126718385