-
【听课笔记】复旦大学遗传学_03基因
课程地址:复旦大学遗传学
三、基因与基因突变
1. 基因的概念
1.1 三位一体的基因概念
- 摩尔根,以果蝇为模式生物,1933 年诺贝尔生理学奖或医学奖,现代遗传学奠基人。
- 遗传学模式生物——果蝇:
- 生活史短(卵到成虫 10 天);
- 繁殖率高;
- 饲养简便(玉米培养基);
- 表型丰富;
- 遗传背景清晰;
- 唾腺染色体(双翅目昆虫的重要特征:巨大的唾腺染色体,复制后姐妹染色单体不分开,所以逐渐加粗加大,最后可以直接在光学显微镜下观察。)
- 三位一体的基因概念:
- 基因确切存在
- 基因呈线状排列在染色体上
- 基因决定某一特定性状(基因是功能单位)
- 基因可突变(基因是突变单位)
- 基因可交换(基因是重组单位)
基因在染色体上的证据
-
细胞遗传学的证据:
-
Walther Flemming 细胞遗传学的奠基人之一,发现染色体,并记录了染色体在有丝分裂中的行为
-
Theodor Boveri 和 Walter Sutton 发现减数分裂中染色体行为与孟德尔有关遗传因子的描述完全相符,进而提出染色体理论。
-
果蝇杂交实验证据(摩尔根):


- 统计更多的杂交后代,发现少量例外的白眼雄蝇和红眼雌蝇。
- 果蝇染色体的特殊现象:X 染色体不分离。

- 镜检例外果蝇的染色体组成,完全符合预期。
- 进一步证明果蝇白眼基因 w 在 X 染色体上。
-
基因在染色体上的证据
■ 染色体在有丝分裂中的行为
■ 染色体在减数分裂中的行为
■ 白眼性状伴性遗传
■ 染色体不分离现象
1.2 三位一体的基因概念的修正
(一)基因是突变单位的修正
-
随着越来越多突变果蝇的鉴定,在果蝇白眼红眼之外又发现了杏色眼(apr)。那么问题来了,w 和 apr 是影响同一性状的两个不同基因的突变?还是同一个基因的两种不同突变形式?

-
互补实验:利用不同突变纯合子杂交得到杂合子一代,根据子一代表型是突变型(同一基因的不同突变)还是野生型(不同基因的突变)判断不同突变是否属于同一基因的实验方法。



-
顺反子(
cistron):即在互补实验中的功能单位。 -
如果一个顺反子内部有两个不同位置的突变,那么突变的顺式杂合子 (
cis) 即突变位于同一条染色体上,表型为野生型;反式杂合子 (trans) 即突变位于不同染色体上,表型为突变型。 -
通常,基因和顺反子是近义词。
-
一个基因内部可以有一个以上的顺反子(多肽不同结构功能域之间的互补作用)。
-
两个基因才构成一个顺反子(基因互作,极性突变,即一个基因的突变可能影响到另一个非等位基因的功能。)
-
基因并非突变单位。
(二)基因是重组单位的修正
- 现象:杏色眼和白眼果蝇杂交的 F2 代中除了杏色眼、白眼、浅杏色眼,还有少量野生型红眼果蝇。
- 提出假设:基因内部在不同碱基之间发生交换,产生少量重组类型的配子。详见连锁与交换章节。

- 基因并非重组单位。
- 同点等位基因(
homoallele):在同一个基因内部的相同位点或重叠位点的不同突变基因,同点等位基因之间既不能互补也不能重组。 - 异点等位基因(
heteroallele) :在同一个基因内部的不同位点的不同突变基因,异点等位基因之间不能互补,但可以重组。
(三)基因是功能单位的修正
- 基因是功能单位
- 一个基因一个酶学说 → 一个基因一个多肽(更严谨)
- 基因的概念随着遗传学的发展而变化。
小结:
- 基因在染色体上线性排列
- 基因不是突变的单位,亦非重组单位
- 一个基因一条多肽(或一个结构性 RNA)
2. 基因的基本属性
问题导入
- 调控序列和结构序列中的调控序列如何发挥作用?
- 对于任何一个生物,它每时每刻都在以同一水平表达细胞内的每个基因吗?
2.1 基因是可调控的(操纵子)
乳糖操纵子的基因结构与调控机制

- 包括结构基因、操纵基因以及结构基因的启动子在内的整个连续的 DNA 结构单元称之为 操纵子 (
operon)。 - 调节基因 lacI:编码阻遏蛋白和 LacO 结合。
- 环境中无乳糖时,lacI 编码的阻遏蛋白能够与操纵基因结合,阻断 RNA 聚合酶在启动子上的移动,此时只有非常低水平的基因转录,提供少量结构基因的产物。

- 环境中添加乳糖,乳糖变为异乳糖,异乳糖与阻遏蛋白结合,改变构型,失去与操纵基因结合的能力,RNA 聚合酶转录下游结构基因,开启转录 3 种酶,作用于乳糖,大大促进乳糖的代谢。直至环境中的乳糖被分解完全后,阻遏蛋白又继续发挥作用,抑制操纵子结构基因的转录。

色氨酸操纵子的基因结构与调控机制
- 缺乏色氨酸时,调节基因 trpR 编码的阻遏蛋白无活性,不能与 trpO 结合,RNA 聚合酶结合到 trpP 上,起始基因的转录。

- 色氨酸存在时,能与 trpR 编码的阻遏蛋白结合,成为有活性的阻遏物,结合到 trpO 上,并抑制 RNA 聚合酶转录下游的结构基因。

- 操纵子模型是基因表达的一种负调控机制,调节基因的表达是关闭结构基因表达所必需的。
- 诱导性操纵子:游离的阻遏蛋白是有活性的,可以直接结合操纵基因,关闭基因表达,如乳糖操纵子。
- 抑制性操纵子:游离的阻遏蛋白没有活性,只有在与共抑制物结合形成复合物的情况下,才能与操纵子结合,关闭基因表达,如色氨酸操纵子。
操纵子的遗传分析
操纵子结构的研究方法(早期)
- 利用突变体遗传分析方法揭示乳糖操纵子的结构和调节功能。
- 利用部分二倍体分析大肠杆菌中不同突变等位基因之间的显隐性关系。
- 结构基因和操纵基因的突变分析

- 调节基因的突变分析

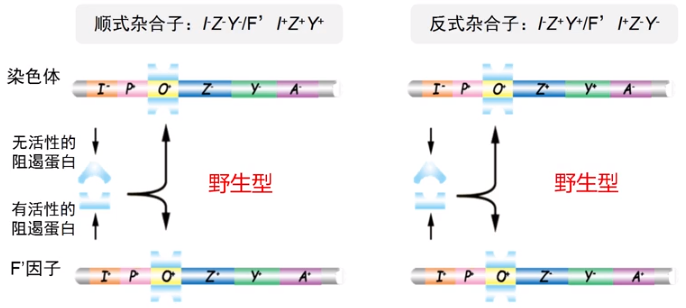
■ 两个基因突变时的表型效应,两个突变基因的顺式杂合子和反式杂合子的表型不一定相同。
■ 操纵基因发挥顺式作用 (cis-acting),它只能影响位于同一条染色体片段上的结构基因的表达。


■ 调节基因发挥反式作用 (trans-acting),它的编码产物能够扩散到不同染色体片段上发挥作用。

代谢抑制
- 代谢抑制 (
catabolite repression) 指的是葡萄糖的存在可抑制其他碳源利用的操纵子的诱导。 - 代谢抑制是基因表达的一种正调控机制,调节基因的表达是打开结构基因表达所必需的。
- 葡萄糖参与乳糖操纵子的转录调节
■ 在乳糖操纵子的启动子上有两个结合位点:lacP(RNA 聚合酶结合位点)、lacC(CAP-cAMP 结合位点)。且 CAP-cAMP 与启动子的结合是乳糖操纵子开启转录所必需的。
■ 当环境中葡萄糖浓度低时,细胞内 cAMP 浓度就高,与 CAP 形成 CAP-cAMP 复合体,复合体结合到乳糖操纵子启动子上,促进 RNA 聚合酶与启动子的结合,开启转录,合成乳糖代谢所需的酶。
■ 当环境中葡萄糖浓度高时,细胞内 cAMP 浓度就低,CAP 不能结合启动子,RNA 聚合酶就无法转录下游的结构基因。
2.2 基因是可割裂的(可变剪接)
割裂基因的发现
- 腺病毒基因的断裂现象(1977 年)
- 腺病毒的分子杂交实验
实验∶腺病毒基因组 DNA 与 Hexon 的 mRNA 进行杂交,电镜观察。
现象:基因组片段可以和 mRNA 形成杂合双链,但在杂合双链的内部溢出 3 个单链 DNA 环。
结论:Hexon 基因内部出现断裂,一些序列在 mRNA 中被删除。

- 真核生物基因的编码序列在染色体上不是连续排列的,而是被内含子间隔开,因此真核生物的基因被称为“割裂基因”(
Split gene)。 - 内含子 (
intron) 指的是成熟的 mRNA 中不出现的基因序列。 - 外显子 (
exon) 是在成熟的 mRNA 中出现的基因序列。
RNA 剪接与可变剪接
- 从 mRNA 初始转录产物中去除内含子的过程被称为 RNA 剪接 (
RNA splicing)。 - mRNA 剪接依赖于特定的 RNA-蛋白质复合物,即剪接复合体 (
spliceosome)。 - tRNA 和 rRNA 也都有特定的剪接方式。酵母 tRNA 依赖于特定的剪切内切核酸酶和剪接连接酶。四膜虫 rRNA 采用自体催化剪接。
- mRNA 的剪接位点
5′剪接位点(通常为GU,简称为 5’SS )。
3′剪接位点(通常为AG,简称为 3’SS )。
剪接分支位点(为A, branchsite ,简称为 BP )。

识别这些剪接位置信号的是一种特殊的非编码 RNA——小核 RNA,SN-RNA 或 U-RNA,五种小核 RNA:U1, U2, U4, U5, U6。
小核 RNA (small nuclear RNA, or U-RNA)先与蛋白质构成核糖核蛋白 (small nuclear ribonu-cleo-proteins , snRNPs),再与其他蛋白质进—步组装成剪接复合体。 - mRNA 的剪接过程
①内含子的 5’未端断裂,并与分支点之间形成分子内磷酸二酯键,呈现一个套索结构;
②内含子的 3’未端断裂,释放出这个套索结构;
③外显子的两个断裂点通过磷酸键重接;
④剪接完成后,snRNP 复合物会被释放,循环使用。

- 可变剪接 (
alternative splicing) 指一个 mRNA 前体在不同的剪接方式下产生不同 mRNA 剪接变异体 (splicing variants) 的现象。人类 95%的割裂基因存在可变剪接现象。 - 案例∶原肌球蛋白 (tropomyosin)

- 可变剪接的方式
①Cassettte exons盒式外显子,又称外显子跳跃,在某些剪接异构体中,该外显子可以被保留,而在另一些异构体中被跳过的现象。
②Mutually exclusive exons互斥外显子
③Competing 5' splice sites竞争性或可变 5’剪接位点
④Competing 3' splice sites竞争性或可变 5’剪接位点
⑤Retained intron内含子保留
⑥Multiple promoters多启动子
⑦Multiple poly(A) sites多 polyA 位点
研究不同物种对剪接方式的偏好,发现在非脊椎动物中,各种剪接方式的比例接近,但在脊椎动物(包括人)中,盒式外显子是最常用的可变剪接方式。

割裂基因的生物学意义
- 内含子参与基因表达调控。可变剪接增加遗传信息多样性,不同的剪接产物可以做不同的时间、不同的空间执行不同的功能,实现高级生命的复杂行为。此外,内含子可以作为顺式调控元件,参与基因的转录表达。
- 内含子也蕴含遗传信息。一个基因的内含子是另一个基因的外显子。如,人的 NF1 基因的第 26 个内含子对应的反义链是 3 个蛋白质编码基因的有义链。这种情况在人类中不常见,但在其他一些较小基因组的低等生物中,可以提高基因组的利用率。此外还发现,内含子可以编码 ncRNA。
- 内含子突变频率高于外显子。内含子没有蛋白质编码功能,所以能够承受较高的突变压力,为基因组的演化积累突变素材。
2.3 基因是可移动的(转座)
转座和转座元件的分类
- 染色体上排列成串的基因并不是固定的,基因可以在染色体上以不规则地方式进行运动,即转座 (
transposition)。 - 能够从基因组的一个位置运动到另一个位置的 DNA 序列被称为可动基因 (
mobile gene), 又称为转座元件 (transposable element)。
★ 转座的方式

Cut-out, paste-inDDE-转座子,剪切-粘贴方式。将转座元件从染色体原有位置上切割下来,并插入新的位置中,依赖特定的转座酶,这类转座酶内都带有 DDE 三氨基酸基序,因此利用这种方式转座的序列称为 DDE 转座子。如,细菌的插入序列、玉米的 Ac-Ds 系统、果蝇的 P 因子等。Copy-out, paste-inLTR-反转录转座子,拷贝-粘贴方式。需要获得一份新的 DNA 拷贝,依赖于反转录酶 RT,RT 以 RNA 转录产物为模板,合成新的 DNA 拷贝,再经过 DDE 转座酶的加工,插入到染色体的新位置。代表性序列:酵母的 Ty1、果蝇的 copia 等。Copy-out, copy-inTP-反转录转座子,拷贝-拷贝方式。需要获得一份新的 DNA 拷贝,依赖于反转录酶 RT,这类转座元件同时编码核酸内切酶,可以直接将反转录产物插入到染色体的新位置而不依赖于 DDE 转座酶。代表性序列:人的 LINE-1 等。
- 这三种转座方式,第一种以 DNA 为中介,也称 DNA 转座子。后两种以 RNA 为中介,称为反转录转座子。
- 反转录转座子 (
retrotransposon): 通过 RNA 为中介,反转录成 DNA 后进行转座的可动元件。转座过程称为反转录转座 (retrotransposition)。
细菌中的转座元件
- 插入序列 (
Insertion Sequence, IS),细菌中的转座元件,最简单的转座元件。IS 两端是短的反向重复序列 IR,一般相同或近乎相同,长度从几个 bp 到几十个 bp 不等;绝大多数 IS 内部含有一个转座酶的编码基因,是结合 IS 元件末端,切割 DNA 并将其插入到靶位点的关键酶。IS 插入宿主基因组后会造成交错切割,从而在插入位置上产生短正向重复序列 (directrepeat, DR)。插入序列如果切离精确,可使 IS 诱发的突变回复为野生型;插入序列的不精确切离,可使插入位置附近的宿主基因发生缺失。

- 复合转座子 (
transposon,Tn) 是一种复合型细菌转座因子。Tn 的两端通常是两个 IS,可以是正向重复或反向重复,两端的 IS 可以作为 Tn 的一部分随同转座,也可以单独发生转座。Tn 的内部携带与转座无关的一些结构基因(如抗药基因)。插入宿主的靶序列后,也在靶序列处形成 DR。
如 Tn9,内部携带克拉霉素基因,末端是正向排列的 IS1;Tn5,内部携带 kana 等抗性基因,末端是反向排列的 IS50;Tn10,内部携带四环素抗性基因,末端是反向排列的 IS10。



- 细菌的转座元件与近年来出现的超级细菌现象之间存在一定的关联。IS、Tn 可以在染色体、质粒等遗传物质中频繁的进行水平交流,并将特定的抗生素抗性基因迅速在细菌的种群中扩散,从而促进病原菌在短期内获得多重抗性,成为超级细菌。
玉米中的转座元件——激活解离系统 (Ac-Ds)
- Ac 基因 (
activator) ,4563np,含有 5 个外显子,能自主移动的调节因子,编码转座酶,两端具有 11np 反向重复序列。 - Ds 基因 (
dissociation): 非自主移动的受体因子,具有结构异质性,不同的 Ds 长度在 0.5 - 4kb 之间,内部转座酶序列发生缺失,不编码转座酶,因此不能自主转座,与 Ac 有同源序列,保留末端的反向重复序列。 - Ac 的基因产物(转座酶)可以扩散到 Ds 元件上,即反式作用与 Ds 元件,与 Ds 结合后帮助 Ds 发生转座。

- Ac-Ds 决定玉米籽粒色斑的遗传




果蝇的 P 因子
- 完整的 P 因子编码的转座酶能结合到 P 因子末端介导其在基因组中的转座
- 黑腹果蝇基因组中还有一些不完整的 P 因子,转座酶基因缺失,不能编码转座酶,但在完整 P 因子存在时,不完整的 P 因子也能在基因组中发生转座。

- P 品系(携带完整Р因子),M 品系(携带不完整Р因子),雄 P 品系和雌 M 品系杂交后代出现杂种不育现象,原因是 P 因子转座导致染色体断裂和重排,生殖细胞发育失败。
- P 因子能自行负调节其转座能力,从而严格控制它在基因组中的数目和稳定性,从而保证了 P 品系果蝇的存活力。
- 为什么雄 P 和雌 M 的后代只有不育的问题,而其他生理结构和功能正常?
只有不育的表型,即突变表型出现了组织差异性,研究发现,只有在生殖细胞中存在 P 因子转座现象,体细胞中 P 因子没有转座现象,原因是细胞特异性可变剪接。生殖细胞中 P 因子转座酶编码基因的成熟 mRNA 保留了第 0、1、2、3 外显子,能编码 87kD 的转座酶,具有转座活性;在体细胞中,一种阻遏性蛋白质结合到转座酶编码基因的 2 号外显子上,抑制了 2、3 外显子间内含子的剪切,成熟 mRNA 中保留了这个内含子,造成移码突变,使得终止密码子提前,得到一条 66kD 的肽段,不具备转座活性。

- 为什么只有雄 P 和雌 M 的后代不育?
母本为 P 品系时杂交后代都是正常的,提示 P 因子的转座活性可能还是受到了卵细胞的细胞质的影响。可能是Р型雌蝇的卵细胞内积累了抑制自身 P 因子随机转座的因子,保护个体发育和种群繁衍,而 M 型果蝇的细胞质中没有抑制机制。研究发现,P 型雌蝇可能通过表达 piRNA (piwi-interacting RNA) 并积累在卵细胞中,传递给子代果蝇抑制 Р 因子的转座。piRNA 一种有功能的 ncRNA,是由 P 因子自身转录表达的,与转座酶 mRNA 之间存在同源性,可以抑制转座酶基因的翻译,促进降解,从而实现转座的抑制。
人类的反转录转座元件 L1
- 人类基因组中有 4 种可动序列,占据人类基因组序列近一半,其中 3 种是反转录转座子。
- 三种反转录转座元件
1)短散在核序列 ( short interspersed nuclear element,SINE)
2)长散在核序列 ( long interspersed nuclear element,LINE)
3)具有长末端重复序列的LTR元件(反转录病毒样元件) - LINE-1 元件
◆ LINE-1 是人类基因组中的代表性 LINE。
◆ LINE-1 全长 6.1kb。两侧有正向重复序列。内部含有两个开放阅读框,编码蛋白的内部含有反转录酶结构域和内切酶结构域。
◆ 全长 LINE-1 具有反转录转座活性。
◆ 人类基因组中有 8×105 拷贝,占基因组 21%。
◆ 基因组中仅 1%的 LINE-1 是全长,截短的 LINE-1 仍能在全长 LINE-1 辅助下转座。

◆ LINE-1 的部分序列插入到了凝血因子Ⅷ的第 14 号外显子中,造成了插入失活,导致甲型血友病。
◆ 哺乳动物神经元细胞和肝脏细胞、心脏细胞中 LINE-1 的转座活性显著不同,可能参与神经元分化、个体发育等。
转座的遗传学效应
- 插入突变:新基因的获得;原有基因的沉默;基因的转录激活/沉默;形成新的正向重复序列。
- 不精确解离导致基因突变。
- 染色体断裂和丢失,在体细胞中造成不稳定的表型变异,在生殖细胞中造成减数分裂不平衡。
- 在基因组中产生大量的重复序列,这些重复序列可以诱导染色体结构变异。染色体上不同位置的同源转座序列之间的交换(非等位同源重组)可诱导染色体缺失、重复、倒位等畸变的发生。
①含有两个正向排列的转座子的染色体片断通过重组可以切除转座序列之间的基因序列,造成染色体缺失。
②含有两个反向排列的转座子的染色体片断通过重组可以形成染色体片断的倒位。
③含有两个正向排列的转座子的染色体可以在姐妹染色单体之间发生重组,结果是一条染色体上的基因加倍,一条发生了丢失。

3. 基因突变
基因突变的分类
(1) 按照基因突变的方向分为正向突变和回复突变。
- 将自然界大量存在的或者实验室中保存的一种标准品系的野生型等位基因形式作为研究生物体基因变异的出发点/标准类型。任何偏离野生型等位基因的变化称为正向突变 (
forward mutation)。 - 与正向变异的概念相对应,从偏离野生型的突变类型向野生类型的变化称为回复突变 (
rescue mutation)或者反向突变 (reverse mutation)。 - 转座子从原位置移动到新位置是一个正向突变,再从新位置精确解离下来就是一个回复突变。
- 造成表型回复的突变可能有两种类型:
△ 真的回复突变,突变发生在同一基因座,跟正突变的位置一样。
△ 抑制因子突变,即突变发生在另一与该基因有相互作用的基因座位上,掩盖了原来突变型的表型效应。
(2)按照基因突变的诱因分为自发突变和诱发突变。
-
自发突变 (
spontaneous mutations) 是生物在自然状态下产生的突变。自发突变可以由 DNA 复制错误、自发损伤、基因转座等多种原因造成。
△ DNA 复制错误:
① 转换 (transition): 嘌呤-嘌呤 嘧啶-嘧啶
② 颠换 (transversion)∶嘌呤-嘧啶 嘧啶-嘌呤
③ 缺失和重复:复制中增加或减少一个或几个碱基
△ 自发损伤:
① 脱嘌呤∶碱基和脱氧核糖之间的糖苷键受到破坏,A 或 G 从 DNA 分子上脱落
② 脱氨基∶胞嘧啶脱氨基变成尿嘧啶,腺嘌呤脱氨基变成次黄嘌呤
③ 氧化性损伤︰细胞内环境中的活性氧化物可以造成 DNA 链的断裂或修饰 -
诱发突变 (
inducedmutations) 是生物暴露在诱变剂中引起的遗传物质的改变。自然环境中的诱变剂包括碱基类似物、烷化剂、嵌合剂、辐射(紫外/电离)等。
△ 碱基类似物︰这些物质由于和碱基在结构上非常类似,可以替代正常碱基掺入到 DNA 分子中,引起 DNA 复制错误。如 5-BU(5-溴尿嘧啶),酮式结构与 A 配对,烯醇式结构与 G 配对。

△ 烷化剂:具有一个或多个活性烷基的化合物。如,甲磺酸乙酯 (EMS) 可以使 DNA 中的碱基发生烷化,造成不正常的碱基配对,如烷化的 G 与 T 配对。

△ 嵌合剂:含有吖啶环的平面分子,可以嵌入到 DNA 双链的碱基对之间,在插入位置引起插入突变。
△ 亚硝酸:有氧化脱氨作用,能使腺嘌呤 A 脱氨基成为次黄嘌呤 H,H 与 C 配对;C 脱氨为 U,U 与 A 配对;G 脱氨为黄嘌呤 X,X 与 C 配对,无诱变作用。
△ 辐射:辐射线(×线、α线、β线、v 线,以及中子、质子等)。辐射所含的能量愈大,诱变的效率更高。辐射剂量的单位用 r(伦琴)表示。电离辐射可诱发基因突变和染色体断裂,它们的频率跟辐射剂量成正比。辐射效应是累积的,低剂量长时间的辐射与高强度短时间辐射同样有害。辐射的诱变效率随生物种类、照射器官和处理时间而有差异。紫外线照射也可诱发突变,但它的穿透力较弱,所以仅用在微生物或体外培养的哺乳动物细胞中。
(3) 按照基因突变的细胞分为体细胞突变和生殖细胞突变。
- 生殖细胞突变发生得越早,影响越多的后代,甚至自身的可育性。
- 体细胞突变发生得越早,突变细胞群越大,嵌合体的表型变化越明显;但不会传递给后代。

基因突变的性质
- 稀有性:自然条件下基因突变率─般较低,并随生物种类和基因而异。
噬菌体和细菌每个世代的自然突变率为 10-8 ~ 10-10;真核生物约为 10-7 ~ 10-9;人 Y 染色体每个世代的突变率为 3.0×10-8 。 - 可逆性:突变型基因可以通过二次突变回复成野生状态,但一般回复突变率低于正向突变率。
- 多向性:一个基因可以以多种方式进行突变,产生不同类型的突变子,得到一系列复等位基因。
基因突变的遗传学效应
- 发生在基因的编码序列内部的突变,其遗传效应主要体现在遗传信息的变化上。
- 发生在基因的非编码序列或者基因间序列的突变,其遗传效应多变。有些突变可能影响了转录/翻译调控序列,并导致基因的表达量或表达谱的变化,但也会有一些突变在表型上没有明显变化。
(1)基因突变对遗传信息的影响
单个碱基突变可造成同义突变、错义突变和无义突变。
- 同义突变 (
synonymous mutation): 不改变氨基酸序列的碱基变化。 - 错义突变 (
missense mutation): 碱基替换导致氨基酸序列发生变化。 - 无义突变 (
nonsense mutation): 编码区的碱基替换后,转录产生终止密码子 (UAG/UAA/UGA),阅读框提前终止。 - 错义突变和无义突变统称为非同义突变 (
nonsynonymous mutation)。 - 碱基插入或缺失对遗传信息的影响是移码突变 (
frameshift mutation) : 由于一个或多个碱基插入或缺失,造成基因的开放阅读框位移,产生完全不同的多肽序列或者提前终止的多肽序列。
(2)基因突变对基因功能的影响
从基因产物的功能对基因突变进行分类,可以有组成型突变、功能丧失型突变和功能获得型突变三大类。-
组成型突变 (
constitutiv-e mutation):突变使得基因产物持续激活,脱离原有的活性调节。如,lacOc。

-
功能丧失型突变 (
loss-of-function mutation):突变破坏了某基因的正常功能,得到无功能或部分功能的产物。功能丧失型突变一般都是隐性突变,即突变杂合子的表型和野生型相同,仅纯合子有突变表型。但是,有时候功能丧失型突变也有显性效应,因为基因组中的一些基因产物具有剂量效应,如果单倍剂量不足,即使是杂合子,也会有突变表型。
-
功能获得型突变 (
gain-of-function mutation) : 突变使得基因获得某种新功能或者原有功能增强,从而产生新表型。功能获得型突变一般都是显性突变,即突变杂合子的表型和野生型不同,表现出突变表型。
4. 基因分型
- 基因型分型 (genotyping) 指的是利用分子生物学方法分析样本的 DNA 序列,从而确定其基因型的实验过程。简言之,就是确定基因的 DNA 序列组成,明确其中是否携带以及携带怎样的突变信息。
- 基因型分型方法
基于分子杂交的检测→基于 DNA 扩增的检测→基于 DNA 测序的检测
四种常见方法:Southern 印迹、DNA 芯片、PCR、DNA 测序
4.1 Southern 印迹
-
Southern 印迹法 (
Southern blot):将 DNA 转移到硝酸纤维素膜上,利用 DNA-DNA 杂交检测特定 DNA 片段的方法。 -
印迹法 (
blot) 是一种将凝胶电泳技术、固定化技术及分子亲和技术融合一体的综合性技术。 -
Southern 印迹的实验流程
① 酶解:获得合适的 DNA 样本,从组织中提取的 DNA 样本分子量较大,先用限制性内切酶消化对提取的 DNA 进行预处理;
② 电泳:利用电泳将 DNA 样本分离,在胶中预先混入核酸的荧光染料,电泳后用紫外线照射可以看到大量连续分布的密集条带;
③ 印迹:将已按大小分离的 DNA 样品转移到固相载体上;
④ DNA 样品杂交:用高浓度的无关 DNA 对膜进行封闭,再与已标记同位素的 DNA 探针杂交,探针识别与之互补的序列并结合在膜上;
⑤ 成像:经放射自显影成像,在全部的 DNA 条带中,只有与对应探针结合的特异性的条带被显现出来。

-
Southern 印迹的关键
■ 通过设计不同的 DNA 探针可实现不同等位基因类型的区分。
■ 探针设计是 Southern blot 实验最为重要的环节之一。
4.2 DNA 芯片
- DNA chip/ DNA 微矩阵 (
DNA microarray):将一系列已知序列的 DNA 探针分子以高密度固定于支持物上,通过与标记的 DNA 样品分子进行杂交实现标记 DNA 的序列分析。

- DNA 芯片的特点
■ 优点:高通量、大规模、平行化、集约化。
■ 缺点:非特异性高(显著高于 Western blot,芯片结果需要额外的实验来验证)。
4.3 PCR
- 聚合酶链式反应 (
Polymerase chain reaction, PCR),是在体外利用 DNA 聚合酶,将少量 DNA 模板通过循环反应特异性扩增的实验方法。 - PCR 过程:模板变性 → 退火结合 → 引物延伸。


- 利用 PCR 方法进行基因型分型
■ 点突变:在突变位置分别设计野生型和突变型等位基因的引物,分别只能扩增出野生型和突变型的等位基因。
■ 插入与缺失:在野生型和突变型等位基因都有的位置上设计通用引物,由于有插入或缺失,突变型和野生型的扩增产物大小不同,可以通过电泳区分。

4.4 DNA 测序
-
DNA 测序 (
DNA sequencing) 即测定 DNA 序列的技术。是当下使用最多的一种基因型分型技术。 -
在等位基因突变方式未知的情况下,不能使用 Southern blot、DNA chip 和 PCR 方法进行基因分型,只能通过 DNA 测序分型。
-
Sanger 测序的实验流程
■ 1. 在 PCR 反应体系中加入少量双脱氧核苷酸 (ddNTP)。由于 DNA 聚合酶不能区分 dNTP 和 ddNTP,所以少量的 ddNTP 会被掺入到 PCR 产物中。
■ 2. ddNTP 可终止延伸反应,得到不同长度的 PCR 产物。 由于 ddNTP 缺少 3’-OH,掺入 ddNTP 后不能继续形成磷酸二酯键,使得延伸反应终止,因此会得到以 ddNTP 结尾的各种长度大小的 PCR 产物。
■ 3. 电泳分离 PCR 产物。通过四种 ddNTP 上携带的荧光探针分子不同来分辨,即可读取正确的 DNA 序列。
■ 4. DNA 序列的读取和分析。后来的测序技术利用荧光探头替代肉眼读序,利用毛细管电泳替代丙烯酰胺电泳,使得传统的 Sanger 测序技术有了飞跃。


-
Sanger 测序的应用
基因组测序
基因型判断和突变分析
基因型分型的应用
- 科学研究
- 遗传测试:
新生儿筛查 ( newborn screening )
产前诊断 ( prenatal diagnosis )
携带者测试 ( carrier testing )
小结:
- 基因是遗传信息的单位一个基因能够编码一段多肽或者一段有结构的 RNA 分子。
- 基因含有调控序列和结构序列
- 真核基因含有外显子和内含子
- 基因有不可动的,也有可动的
- 基因突变可以从多种层次上进行分类,突变能对基因的遗传信息和蛋白质功能产生影响。
- 利用包括 Southern blot、DNA 芯片、PCR 和 DNA 测序等方法可以对不同基因型进行鉴定。
-
相关阅读:
jQuery之事务相关操作
网安之PHP基础作业(5)
Elastic认证考试过程(2022.11.13 06:15)
《XSS-Labs》01. Level 1~10
卡塔尔世界杯倒计时!世界杯直播在哪里观看?美家市场汇总来了!
健康之路押注医药零售:毛利率下滑亏损扩大,医疗咨询人次大幅减少
DevOps|AGI : 智能时代研发效能平台新引擎(上)
【无标题】
[附源码]JAVA毕业设计课程网站设计(系统+LW)
MojoTween:使用「Burst、Jobs、Collections、Mathematics」优化实现的Unity顶级「Tween动画引擎」
- 原文地址:https://blog.csdn.net/zea408497299/article/details/125947777