-
R 笔记 MICE
1 MICE 算法理论部分
MICE(Multiple Imputation by Chained Equations)是一种处理数据集中缺失数据的稳健、信息丰富的方法。 该过程通过一系列迭代的预测模型“填充”(估算)数据集中的缺失数据。
在每次迭代中,数据集中的每个指定变量都使用数据集中的其他变量进行估算。 不断迭代在,直至收敛。
1.1 MICE举例
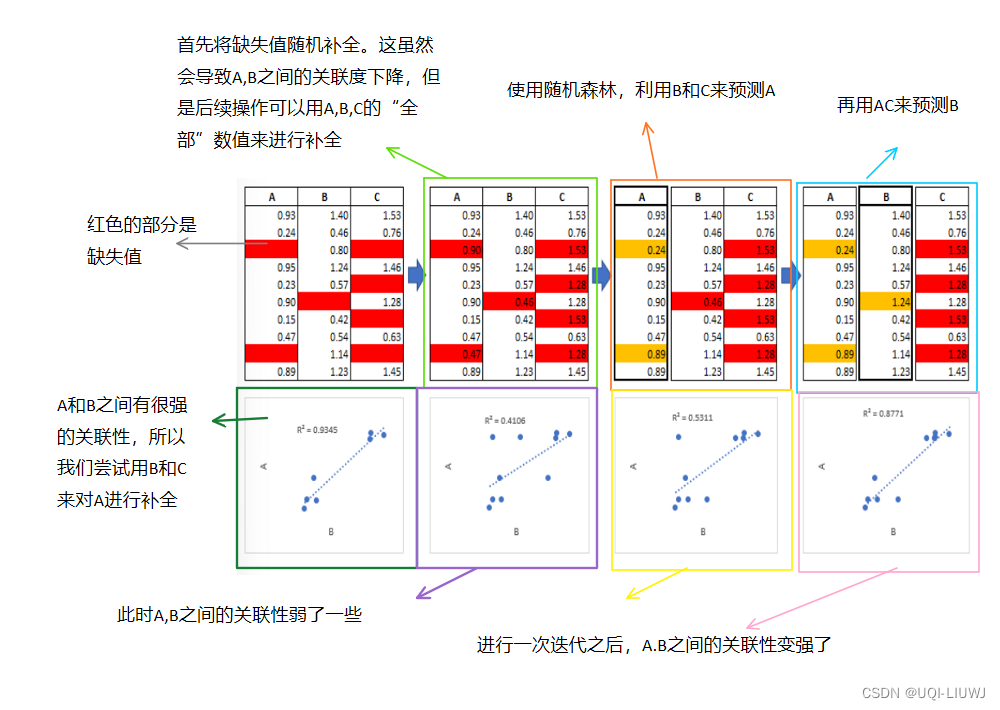
上述这个过程一直持续到所有指定的变量都被插补。 如果没有收敛,则可以运行额外的迭代,尽管通常不超过 5 次迭代是必要的。
插补的准确性取决于数据集中的信息密度。 没有相关性的完全独立变量的数据集不会产生准确的插补。
1.2 PMM,Predictive Mean Matching
MICE 可以使用称为预测均值匹配 (PMM) 的程序来选择要估算的值。 PMM 从原始非缺失数据中选择一个数据点,该数据点的预测值接近缺失样本的预测值。
选择最接近的 N个数据点作为候选值,从中随机选择一个值来进行补全。

2 R语言MICE
2.0 导入包
- library(magrittr)
- library(dplyr)
- library(mice)
- library(missForest)
2.1 导入数据
- data(iris)
- summary(iris)
- # Sepal.Length Sepal.Width Petal.Length
- # Min. :4.300 Min. :2.000 Min. :1.000
- # 1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600
- # Median :5.800 Median :3.000 Median :4.350
- # Mean :5.843 Mean :3.057 Mean :3.758
- # 3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100
- # Max. :7.900 Max. :4.400 Max. :6.900
- # Petal.Width Species
- # Min. :0.100 setosa :50
- # 1st Qu.:0.300 versicolor:50
- # Median :1.300 virginica :50
- # Mean :1.199
- # 3rd Qu.:1.800
- # Max. :2.500
2.2 随机丢失一定量数据
随机在数据里产生 10% 的 缺失值。同时把
Species这个分类变量也去掉。- iris_mis <- missForest::prodNA(iris, noNA = 0.1) %>% select(-Species)
- summary(iris_mis)
- # Sepal.Length Sepal.Width Petal.Length
- # Min. :4.300 Min. :2.000 Min. :1.000
- # 1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.500
- # Median :5.800 Median :3.000 Median :4.300
- # Mean :5.856 Mean :3.049 Mean :3.707
- # 3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100
- # Max. :7.900 Max. :4.200 Max. :6.900
- # NA's :16 NA's :15 NA's :7
- # Petal.Width
- # Min. :0.100
- # 1st Qu.:0.300
- # Median :1.300
- # Mean :1.201
- # 3rd Qu.:1.800
- # Max. :2.500
- # NA's :13
2.3 可视化缺失数据
md.pattern(iris_mis)
表达的意思是Petal.Length一共7个missing 数据,其中两个和第二列的Petal.Weight在同样的坐标处丢失数据;剩下5个只有在自己的坐标处丢失数据。
2.4 进行补全
imputed_Data <- mice(iris_mis, m=5, maxit = 50, method = 'pmm', seed = 123)- m = 5 ,表示生成 5 组填补好的数据
- maxit = 50,每次产生填补数据的迭代次数,这里取 50 次
- method = ‘pmm’,使用1.2介绍的 Predictive Mean Matching 的方法(连续型数据采用之)
2.5 查看数据
由于我们这边生成了5组数据,所以可以一组一组查看
completeData <- mice::complete(imputed_Data,2)后面这个2就表示查看哪一组的数据
-
相关阅读:
剑指 Offer 10- II. 青蛙跳台阶问题
冰冰学习笔记:vector部分练习题分析
咖啡技术培训:9款网红咖啡制作配方合集,简单快速
一起学数据结构(9)——二叉树的链式存储及相关功能实现
根据您的数据量定制的ChatGPT,改变客户服务的方式
vue2知识点:vuex中四个map方法的使用
【网络】UDP和TCP套接字编程
C11线程池详解
金仓数据库 KingbaseES V8 GIS数据迁移方案(3. 基于ArcGIS平台的数据迁移到KES)
纹波类型及纹波抑制措施
- 原文地址:https://blog.csdn.net/qq_40206371/article/details/126020086
