-
机器学习笔记 - 使用机器学习进行鸟类物种分类
一、问题概述
科学家们已经确定,一种已知的鸟类应该分为 3 个不同且独立的物种。这些物种是该国特定地区的特有物种,必须尽可能精确地跟踪和估计它们的种群。
因此,一个非营利性的保护协会承担了这项任务。他们需要能够根据现场官员在野外观察到的特征记录他们遇到的物种。
使用某些遗传特征和位置数据,您能预测已观察到的鸟类种类吗?
这是一个初学者级别的练习比赛,你的目标是根据属性或位置预测鸟类的种类。
二、数据集
数据已方便地拆分为训练和测试数据集。在每次训练和测试中,您都会获得位置 1 到 3 的鸟类数据。
数据集下载地址
- 链接:https://pan.baidu.com/s/1aalzQNr0IQLQc3X4JTu9nQ
- 提取码:xvy0
下面来看看前五行
training_set.csvbill_depth bill_length wing_length location mass sex ID 14.3 48.2 210 loc_2 4600 0 284 14.4 48.4 203 loc_2 4625 0 101 18.4 NA 200 loc_3 3400 0 400 14.98211382 47.50487805 NA NA 4800 0 98 18.98211382 38.25930705 217.1869919 loc_3 5200 0 103 training_set和training_target可以根据‘id’列关联。
列的含义如下
- species : 动物种类 (A, B, C)
- bill_length : 喙长 (mm)
- bill_depth : 喙深 (mm)
- wing_length : 翼长 (mm)
- mass : 体重 (g)
- location : 岛型 (Location 1, 2, 3 )
- 性别 :动物性别(0:男性;1:女性;NA:未知)
三、编写代码
1、导入库
- import pandas as pd
- # plotting
- import matplotlib
- import matplotlib.pyplot as plt
- import seaborn as sns
- matplotlib.rcParams['figure.dpi'] = 100
- sns.set(rc={'figure.figsize':(11.7,8.27)})
- sns.set(style="whitegrid")
- %matplotlib inline
- # ml
- from sklearn.metrics import ConfusionMatrixDisplay, classification_report
- from sklearn.model_selection import train_test_split
- from sklearn.impute import SimpleImputer
- from sklearn.preprocessing import LabelEncoder
- from sklearn.tree import DecisionTreeClassifier
- from sklearn import tree
2、处理缺失值的方法
- def missing_vals(df):
- """prints out columns with perc of missing values"""
- missing = [
- (df.columns[idx], perc)
- for idx, perc in enumerate(df.isna().mean() * 100)
- if perc > 0
- ]
- if len(missing) == 0:
- return "no missing values"
- # sort desc by perc
- missing.sort(key=lambda x: x[1], reverse=True)
- print(f"There are a total of {len(missing)} variables with missing values\n")
- for tup in missing:
- print(str.ljust(f"{tup[0]:<20} => {round(tup[1], 3)}%", 1))
3、加载数据
首先,我们使用 read_csv 函数加载训练和测试数据。
我们还将 training_set.csv(包含特征)与 training_target.csv(包含目标变量)合并并形成训练数据。
- train = pd.read_csv("dataset/training_set/training_set.csv")
- labels = pd.read_csv("dataset/training_set/training_target.csv")
- # join target variable to training set
- train = train.merge(labels, on="ID")
- test = pd.read_csv("dataset/test_set/test_set.csv")

- target_cols = "species"
- num_cols = ["bill_depth", "bill_length", "wing_length", "mass"]
- cat_cols = ["location", "sex"]
- all_cols = num_cols + cat_cols + [target_cols]
- train = train[all_cols]
4、探索性数据分析Exploratory Data Analysis (EDA)
这是我们研究数据趋势和模式的地方,包括数字和分类。
train.info()使用 info 函数,我们可以看到行数和数据类型。
- <class 'pandas.core.frame.DataFrame'>
- Int64Index: 435 entries, 0 to 434
- Data columns (total 7 columns):
- # Column Non-Null Count Dtype
- --- ------ -------------- -----
- 0 bill_depth 434 non-null float64
- 1 bill_length 295 non-null float64
- 2 wing_length 298 non-null float64
- 3 mass 433 non-null float64
- 4 location 405 non-null object
- 5 sex 379 non-null float64
- 6 species 435 non-null object
- dtypes: float64(5), object(2)
- memory usage: 27.2+ KB
Numerical
让我们绘制数值变量的直方图。
train[num_cols].hist(figsize=(20, 14));
bill_depth 在 15 和 19 左右达到峰值
纸币长度在 39 和 47 左右达到峰值
翼长在 190 和 216 左右达到峰值
质量向右倾斜Categorical
- to_plot = cat_cols + [target_cols]
- fig, axes = plt.subplots(1, 3, figsize=(20, 7), dpi=100)
- for i, col_name in enumerate(train[to_plot].columns):
- sns.countplot(x = col_name, data = train, palette="Set1", ax=axes[i % 3])
- axes[i % 3].set_title(f"{col_name}", fontsize=13)
- plt.subplots_adjust(hspace=0.45)
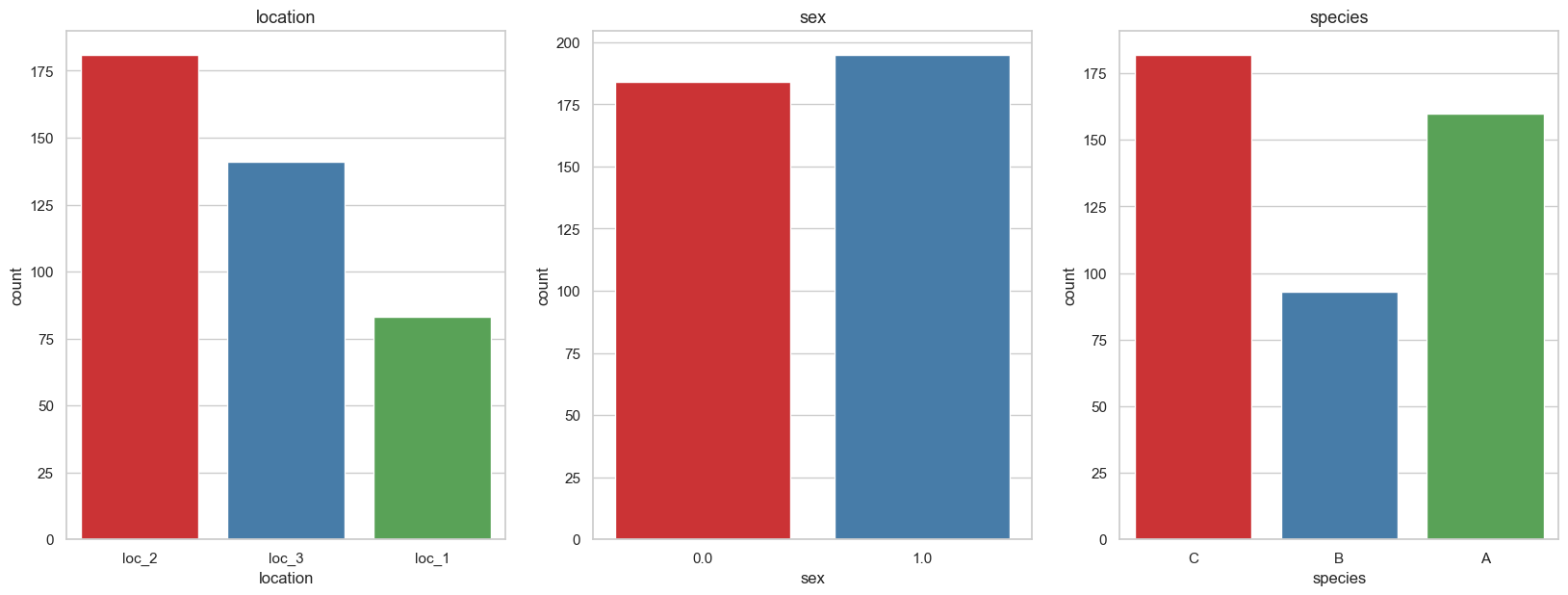
我们看到位置和物种似乎与它们各自的位置和物种相匹配(loc2 和物种 C、loc3 和物种 A)。 我们还看到雌性 (1) 鸟类比雄性鸟类略多。
train.species.value_counts()- C 182
- A 160
- B 93
- Name: species, dtype: int64
仔细观察,我们发现目标变量是不平衡的,其中 B 类比 C 低近 100 个类,比 A 低大约 70 个。
不平衡类是一个问题,因为它使模型偏向于更重视具有更多样本的类,即。 C 比 B 更经常被预测。
5、缺失数据
缺失值的百分比
missing_vals(train)- There are a total of 6 variables with missing values
- bill_length => 32.184%
- wing_length => 31.494%
- sex => 12.874%
- location => 6.897%
- mass => 0.46%
- bill_depth => 0.23%
通过我们的辅助函数,我们发现 bill_length 和wing_length 有超过 30% 的缺失值
热图Heatplot
- plt.figure(figsize=(10, 6))
- sns.heatmap(train.isnull(), yticklabels=False, cmap='viridis', cbar=False);
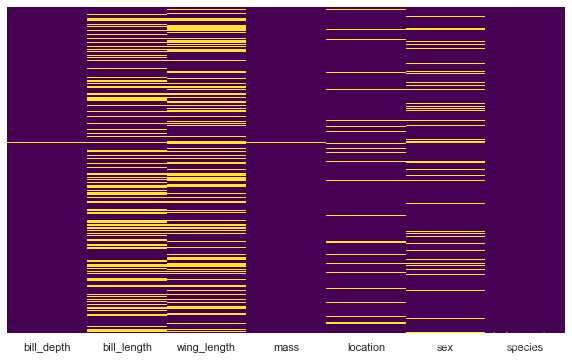
我们还可以绘制热图以可视化缺失值并查看是否有任何模式。
估算分类列
让我们先看看我们的分类变量中有多少缺失变量
train.sex.value_counts(dropna=False)- 1.0 195
- 0.0 184
- NaN 56
- Name: sex, dtype: int64
train.location.value_counts(dropna=False)- loc_2 181
- loc_3 141
- loc_1 83
- NaN 30
- Name: location, dtype: int64
让我们使用简单的 imputer 来处理它们,用最频繁的值替换它们。
- cat_imp = SimpleImputer(strategy="most_frequent")
- train[cat_cols] = cat_imp.fit_transform(train[cat_cols])
再次确认,已经没有缺失值了。如您所见,通过“最频繁”策略,缺失值被估算为 1.0,这是最频繁的。
train.sex.value_counts(dropna=False)- 1.0 251
- 0.0 184
- Name: sex, dtype: int64
估算数值列
让我们使用中值来估算我们的数值
- num_imp = SimpleImputer(strategy="median")
- train[num_cols] = num_imp.fit_transform(train[num_cols])
missing_vals(train)'no missing values'6、特征工程
train.species.value_counts()- C 182
- A 160
- B 93
- Name: species, dtype: int64
编码分类变量
使用标签编码器,我们可以将分类变量(和目标变量)编码为数值。 我们这样做是因为大多数 ML 模型不适用于字符串值。
- le = LabelEncoder()
- le.fit(train['species'])
- le_name_map = dict(zip(le.classes_, le.transform(le.classes_)))
- le_name_map
{'A': 0, 'B': 1, 'C': 2}我们可以先把编码器拟合到变量上,然后查看映射是什么样子的,这样以后我们就可以反转映射了
train['species'] = le.fit_transform(train['species'])对于其他具有字符串变量(非数字)的列,我们也进行相同的编码。
- for col in cat_cols:
- if train[col].dtype == "object":
- train[col] = le.fit_transform(train[col])
train.head()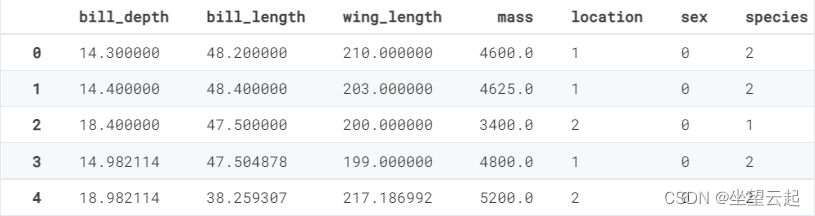
- # Convert cat_features to pd.Categorical dtype
- for col in cat_cols:
- train[col] = pd.Categorical(train[col])
我们还将分类特征转换为 pd.Categorical dtype
train.dtypes- bill_depth float64
- bill_length float64
- wing_length float64
- mass float64
- location category
- sex category
- species int64
- dtype: object
7、创建新特征
- train['b_depth_length_ratio'] = train['bill_depth'] / train['bill_length']
- train['b_length_depth_ratio'] = train['bill_length'] / train['bill_depth']
- train['w_length_mass_ratio'] = train['wing_length'] / train['mass']
在这里,我们创建一些具有除法的特征以形成变量的比率
train.head()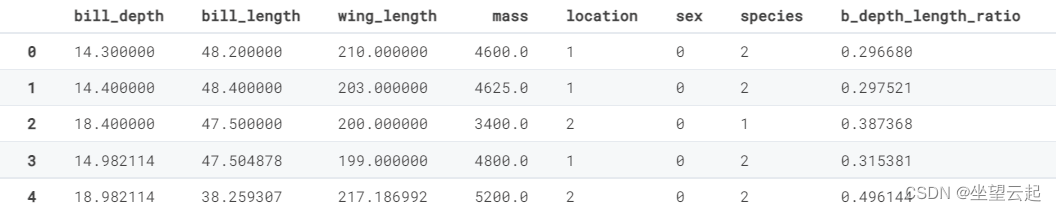
8、模型
训练测试拆分
现在是构建模型的时候了,我们首先将其拆分为 X(特征)和 y(目标变量),然后将其拆分为训练集和评估集。
训练是我们训练模型的地方,评估是我们在将模型拟合到测试集之前对其进行测试的地方。
- X, y = train.drop(["species"], axis=1), train[["species"]].values.flatten()
- X_train, X_eval, y_train, y_eval = train_test_split(
- X, y, test_size=0.25, random_state=0)
简单的决策树分类器
在这里,我们使用 max_depth = 2 的简单超参数拟合基线模型
dtree_model = DecisionTreeClassifier(max_depth = 2).fit(X_train, y_train)拟合数据后,我们可以使用它来进行预测
dtree_pred = dtree_model.predict(X_eval)9、模型性能
print(classification_report(dtree_pred, y_eval))- precision recall f1-score support
- 0 1.00 0.70 0.82 57
- 1 0.71 0.92 0.80 13
- 2 0.75 1.00 0.86 39
- accuracy 0.83 109
- macro avg 0.82 0.87 0.83 109
- weighted avg 0.88 0.83 0.83 109
分类报告向我们展示了分类器的有用指标。
例如,我们模型的 f1-score 为 0.83
10、混淆矩阵
我们还可以构建一个混淆矩阵来可视化我们的分类器在什么方面做得好/坏。
- # save the target variable classes
- class_names = le_name_map.keys()
- titles_options = [
- ("Confusion matrix, without normalization", None),
- ("Normalized confusion matrix", "true"),
- ]
- for title, normalize in titles_options:
- fig, ax = plt.subplots(figsize=(8, 8))
- disp = ConfusionMatrixDisplay.from_estimator(
- dtree_model,
- X_eval,
- y_eval,
- display_labels=class_names,
- cmap=plt.cm.Blues,
- normalize=normalize,
- ax = ax
- )
- disp.ax_.set_title(title)
- disp.ax_.grid(False)
- print(title)
- print(disp.confusion_matrix)
- Confusion matrix, without normalization
- [[40 0 0]
- [ 5 12 0]
- [12 1 39]]
- Normalized confusion matrix
- [[1. 0. 0. ]
- [0.29411765 0.70588235 0. ]
- [0.23076923 0.01923077 0.75 ]]
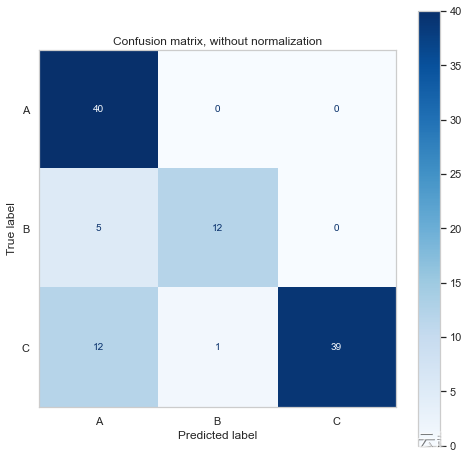
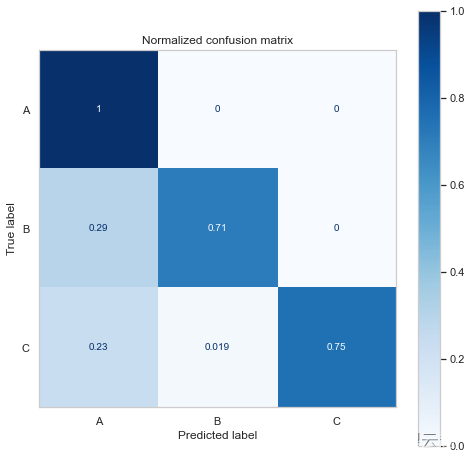
混淆矩阵向我们展示了它预测了更多的 A 类和 C 类,这并不奇怪,因为我们有更多的样本。
它还表明该模型在应该是 B/C 时预测了更多的 A 类。
11、特征重要性
- feature_imp = pd.DataFrame(sorted(zip(dtree_model.feature_importances_,X.columns)), columns=['Value','Feature'])
- plt.figure(figsize=(20, 15))
- sns.barplot(x="Value", y="Feature", data=feature_imp.sort_values(by="Value", ascending=False))
- plt.title('LightGBM Features')
- plt.tight_layout()
- # plt.savefig('lightgbm_fimp.png')
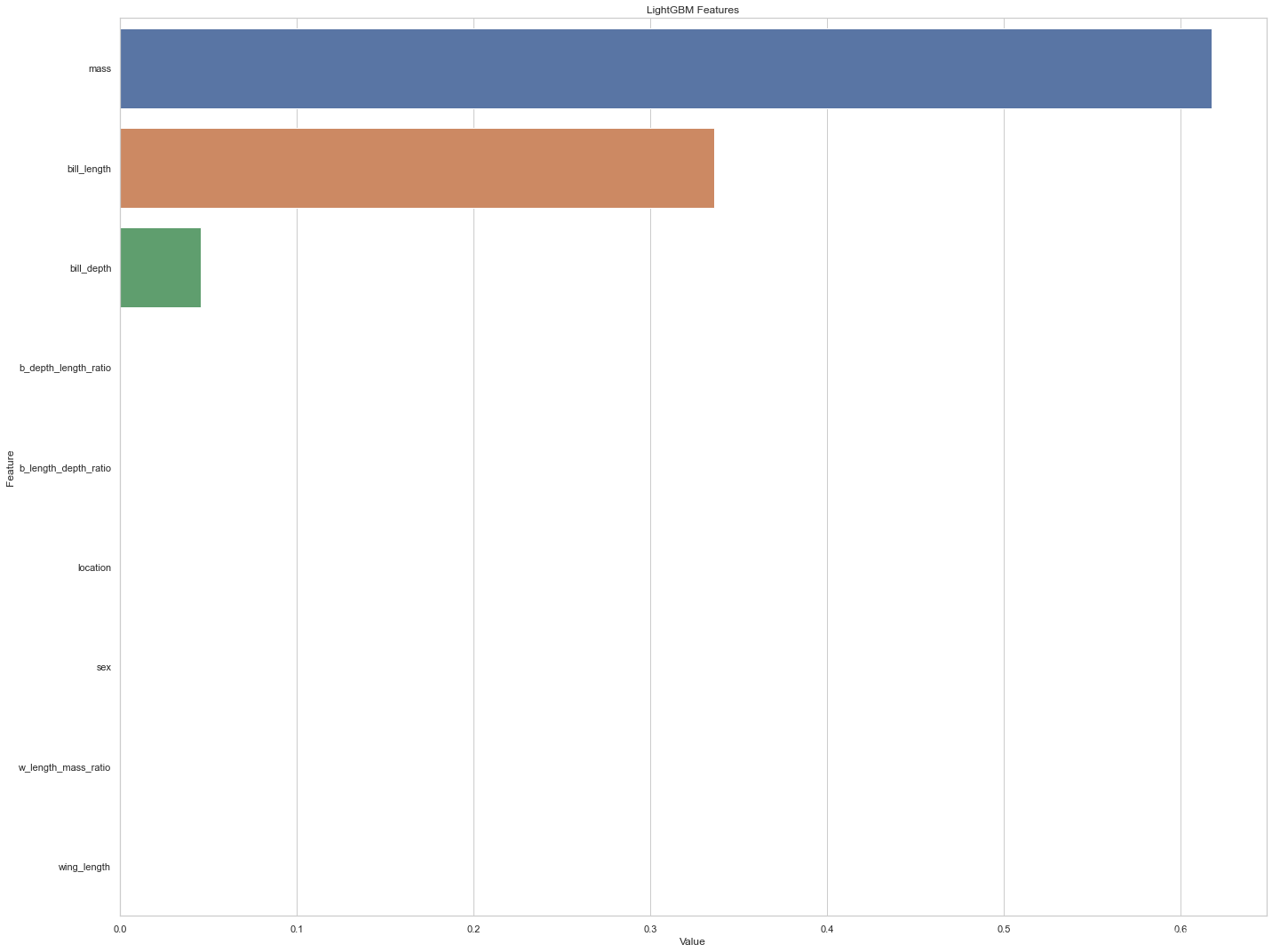
从特征重要性来看,似乎质量预测物种的能力最好,其次是喙长。 其他变量在分类器中的重要性似乎为零
- fig = plt.figure(figsize=(25,20))
- _ = tree.plot_tree(dtree_model,
- feature_names=X.columns,
- class_names=list(class_names),
- filled=True)
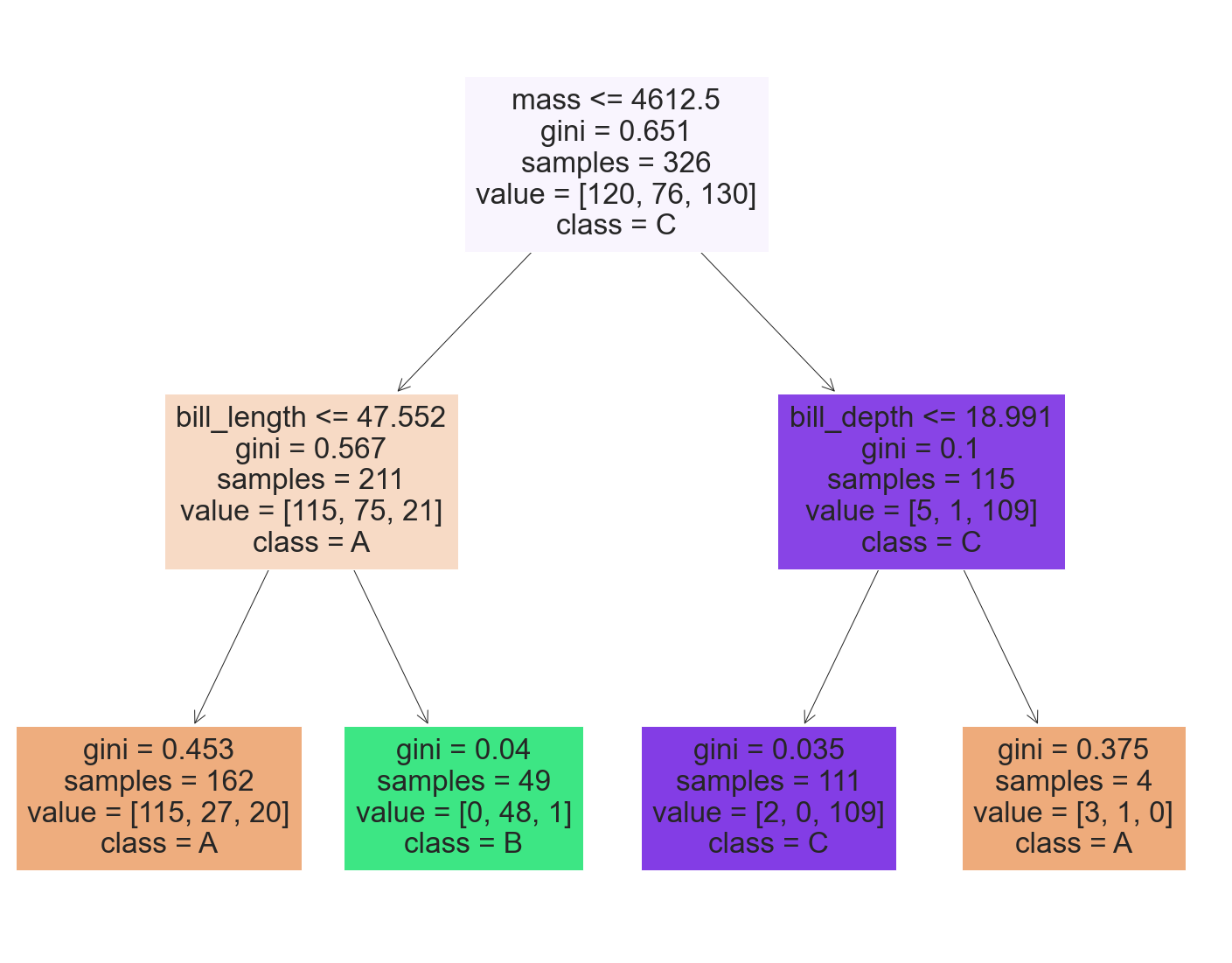
我们看到了在我们的决策树分类器的可视化中如何使用特征重要性。
在根节点中,如果质量低于 4600 左右,则检查 bill_length,否则检查 bill_depth,然后在叶处预测类别。
四、预测测试数据
现在是时候在我们将模型拟合到测试数据之前,对训练数据进行相同的特征预处理和工程了。
- le = LabelEncoder()
- cat_imp = SimpleImputer(strategy="most_frequent")
- num_imp = SimpleImputer(strategy="median")
- test[cat_cols] = cat_imp.fit_transform(test[cat_cols])
- test[num_cols] = num_imp.fit_transform(test[num_cols])
- for col in cat_cols:
- if test[col].dtype == "object":
- test[col] = le.fit_transform(test[col])
- # Convert cat_features to pd.Categorical dtype
- for col in cat_cols:
- test[col] = pd.Categorical(test[col])
- # save ID column
- test_id = test["ID"]
- all_cols.remove('species')
- test = test[all_cols]
- test['b_depth_length_ratio'] = test['bill_depth'] / test['bill_length']
- test['b_length_depth_ratio'] = test['bill_length'] / test['bill_depth']
- test['w_length_mass_ratio'] = test['wing_length'] / test['mass']
- test_preds = dtree_model.predict(test)
- submission_df = pd.concat([test_id, pd.DataFrame(test_preds, columns=['species'])], axis=1)
- submission_df.head()
ID species 0 2 2 1 5 0 2 7 0 3 8 0 4 9 0 请注意,物种值是数字,我们必须将其转换回字符串值。 使用之前带有 fit 的标签编码器,我们可以这样做。
le_name_map{'A': 0, 'B': 1, 'C': 2}- inv_map = {v: k for k, v in le_name_map.items()}
- inv_map
{0: 'A', 1: 'B', 2: 'C'}- submission_df['species'] = submission_df['species'].map(inv_map)
- submission_df.head()
ID species 0 2 C 1 5 A 2 7 A 3 8 A 4 9 A submission_df.to_csv('solution.csv', index=False)最后,我们将数据框写入 csv 文件。
-
相关阅读:
企业微信hook接口DLL调用(4.1.22.6009版本)
MySQL安装
centos7安装cdh6.3.2-附带安装包
crp Week1周报
在Qt中怎么操作MySQL数据库
高斯滤波算法及例程
【VeighNa】开始量化交易——第三章:构建价差套利
在Visual Studio/Qt Creator 中使用CMake安装和使用vcpkg包
【教学类】小2班学号字帖(A4横版2份)
最高频的五个面试题
- 原文地址:https://blog.csdn.net/bashendixie5/article/details/125635006