-
基因家族特征分析 - 染色体定位分析
1 染色体定位分析概念
染色体定位分析是将基因家族成员在染色体上的位置标注出来,观察基因家族成员在染色体上是否成簇分布。
2 分析工具
2.1 MG2C(MapGene2Chromosome)
MG2C是在线分析工具,网址如下:
http://mg2c.iask.in/mg2c_v2.0/- 1
2.1.1 输入文件
在页面上方有两个对话框,左边填入4列以制表符分隔的矩阵,从左到右,以此为:GeneID、起始位置、终止位置和染色体号,这些数据可由组装到染色体水平的注释文件得到。
2.1.2 输出结果
点击DRAW按钮,就可绘图,生成图片可以导出为.svg格式,后续再利用Ai进行修改。
2.1.3 参数修改
绘制的图片中,染色体id的字体、字号等细节通过页面左方的选项框进行修改。
2.2 TBtools
TBtools是功能强大的图形化操作的基于Windows、Mac操作系统开发的生信分析软件,下方是文献引用的GB/T 7714格式。
Chen C, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular plant, 2020, 13(8): 1194-1202.- 1
文献的DOI为https://doi.org/10.1016/j.molp.2020.06.009
选择菜单栏的Graphics,再选择下拉选项框中的Show Genes on Chromosome,选择Gene Location Visualize from GTF/GFF。
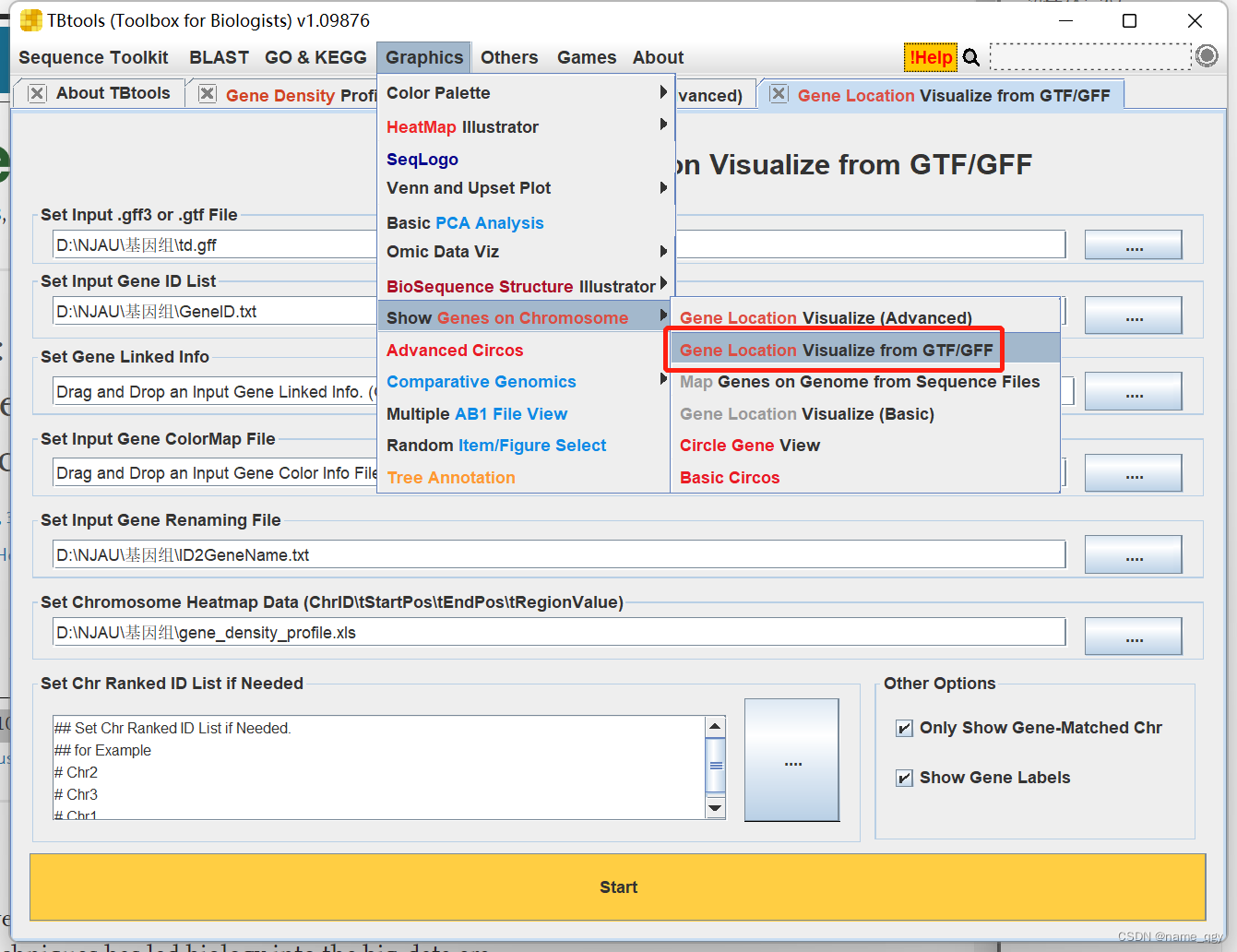
2.2.1 输入文件
- .gff注释文件;
- 想要展示的Gene,保存着基因ID的单列矩阵(基因ID是可以与gff文件第九列GeneID对应的);
- 重复基因对,保存着基因ID的两列以制表符分隔的矩阵(每一行的这两个基因序列是基本一致的,即同一个基因在染色体上出现了两次拷贝);
- Gene重命名,将gff注释文件中GeneID修改为自己想要的名字,制表符分隔的两列矩阵;
- 基因颜色,保存着制表符分隔的两列矩阵,第一列为Gene重命名,第二列是十进制的颜色代码;
- 染色体上基因密度的文件,通过TBtools的Sequence Toolkit菜单栏下的GFF3/GTF Manipulate选项栏下的Gene Density Profile功能,传入.gff文件,修改划分染色体的bin的大小即可生成。
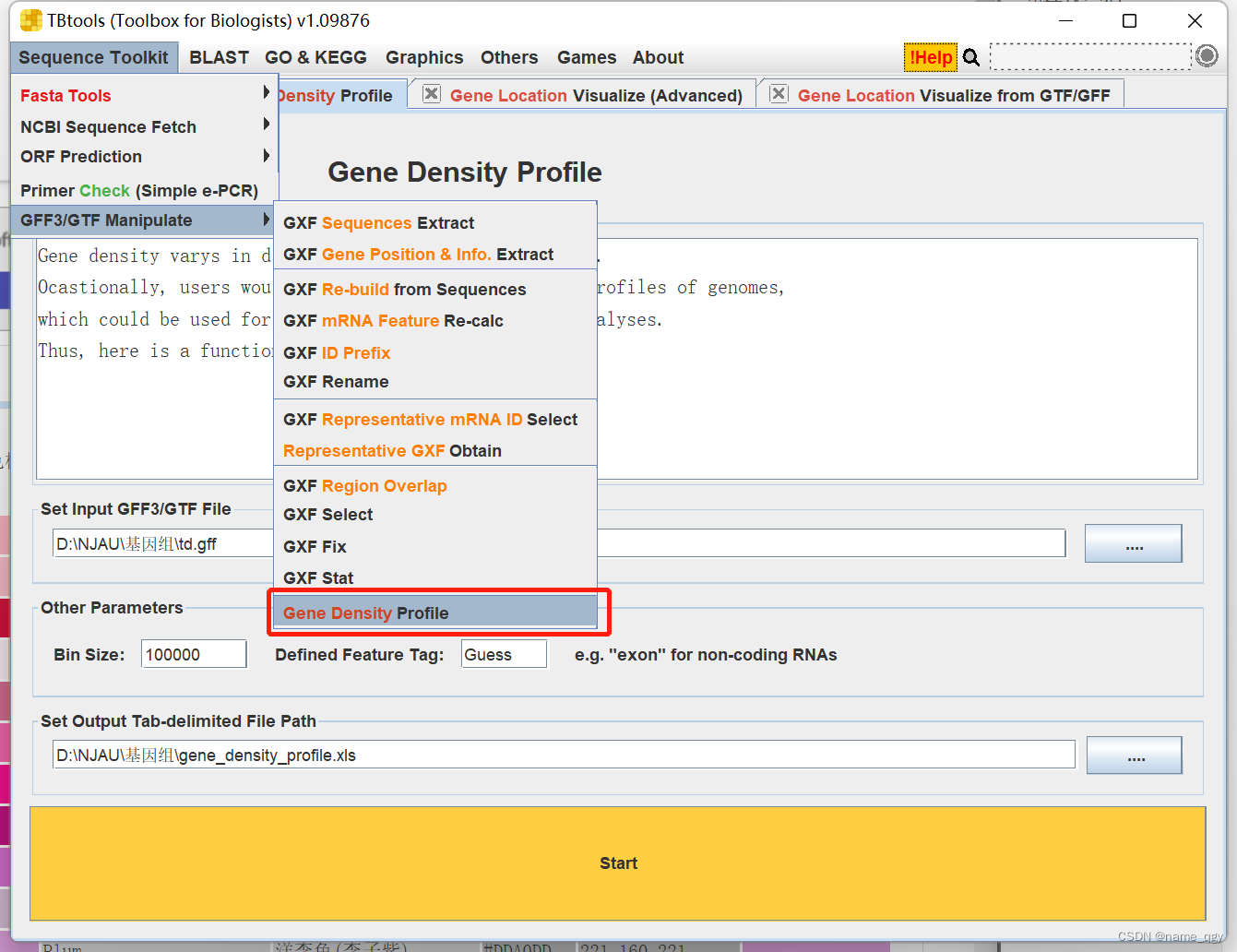
上述6个文件中,1和2是必须的,3-6是可选的。
2.2.2 输出结果
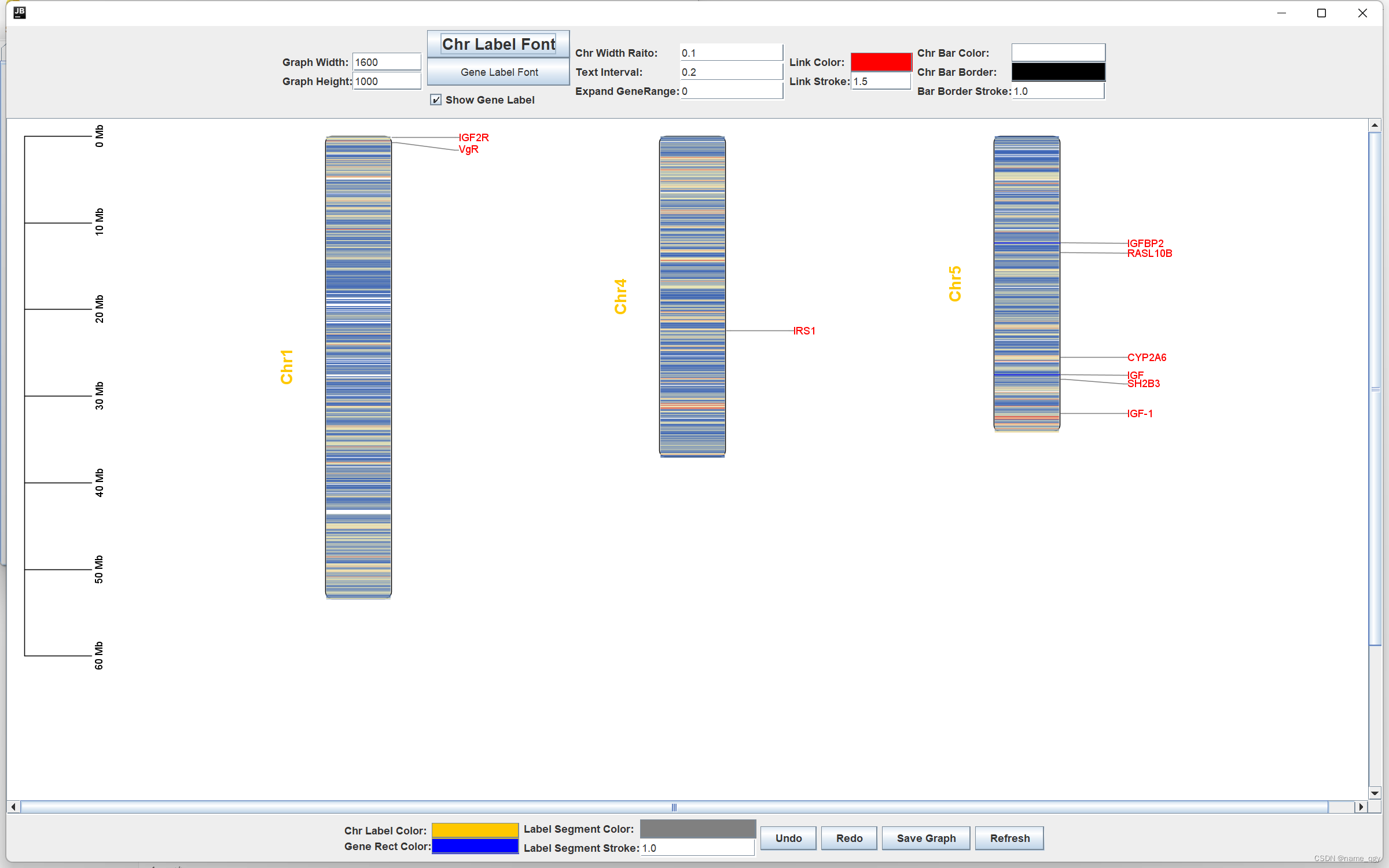
生成结果如上图所示,可以在该窗口下,修改染色体宽度、图片展示的比例等等,点击下方的Save Graph,根据自己的需要生成.png、.jpg、.svg等格式。
2.2.3 最终结果

再用Ai加个图例,标注不同的颜色对应的基因密度就很完美了。
-
相关阅读:
15. SAP ABAP OData 服务里 EntityType 和 EntitySet 的区别
C语言笔记第03章:数组
Docker 镜像全军覆没后,如何给Ubuntu手动安装 docker 服务
MySql ocp认证之MySql安装(一)
银行分布式存储基础设施创新实践分享
523. 连续的子数组和 (前缀和 + 同余性质+哈希)
Linux命令dmesg介绍和使用
Git 的基本概念和使用方式
golang学习笔记(defer基础知识)
K8s---Pod搭建LNMP
- 原文地址:https://blog.csdn.net/qq_50637636/article/details/125516436