-
分布式机器学习:同步并行SGD算法的实现与复杂度分析
1 分布式机器学习概述
大规模机器学习训练常面临计算量大、训练数据大(单机存不下)、模型规模大的问题,对此分布式机器学习是一个很好的解决方案。
1)对于计算量大的问题,分布式多机并行运算可以基本解决。不过需要与传统HPC中的共享内存式的多线程并行运算(如OpenMP)以及CPU-GPU计算架构做区分,这两种单机的计算模式我们一般称为 计算并行 )。
2)对于训练数据大的问题,需要将数据进行划分并分配到多个工作节点(Worker)上进行训练,这种技巧一般被称为 数据并行 。每个工作节点会根据局部数据训练出一个本地模型,并且会按照一定的规律和其他工作节点进行通信(通信内容主要是本地模型参数或者参数更新),以保证最终可以有效整合来自各个工作节点的训练结果并得到全局的机器学习模型。

如果是训练数据的样本量比较大,则需要对数据按照样本进行划分,我们称之为“数据样本划分”,按实现方法可分为“随机采样法”和“置乱切分法”。样本划分的合理性在于机器学习中的经验风险函数关于样本是可分的,我们将每个子集上的局部梯度平均,仍可得到整个经验风险函数的梯度。
如果训练数据的维度较高,还可对数据按照维度进行划分,我们称之为“数据维度划分”。它相较数据样本划分而言,与模型性质和优化方法的耦合度更高。如神经网络中各维度高度耦合,就难以采用此方式。不过,决策树对维度的处理相对独立可分,将数据按维度划分后,各节点可独立计算局部维度子集中具有最优信息增益的维度,然后进行汇总。此外,在线性模型中,模型参数与数据维度是一一对应的,故数据维度划分常与下面提到的模型并行相互结合。
3)对于模型规模大的问题,则需要对模型进行划分,并且分配到不同的工作节点上进行训练,这种技巧一般被称为 模型并行 。与数据并行不同,模型并行的框架下各个子模型之间的依赖关系非常强,因为某个子模型的输出可能是另外一个子模型的输入,如果不进行中间计算结果的通信,则无法完成整个模型训练。因此,一般而言,模型并行相比数据并行对通信的要求更高。

这里提一下数据样本划分中的几种划分方式。给定 个 维样本和 个工作节点,数据样本划分需要完成的任务是将 个样本以某种形式分配到 个工作节点上。
随机采样法中我们独立同分布地从 个样本中有放回随机采样,每抽取一个样本将其分配到一个工作节点上。这个过程等价于先随机采 个样本,然后均等地划分为 份。
随机采样法便于理论分析,但基于实现难度的考虑,在工程中更多采用的是基于置乱切分的划分方法。即现将 个样本随机置乱,再把数据均等地切分为 份,再分配到 个节点上进行训练。置乱切分相当于无放回采样,每个样本都会出现在某个工作节点上,每个工作节点的本地数据没有重复,故训练数据的信息量一般会更大。我们下面的划分方式都默认采取置乱切分的方法。
我们在后面的博客中会依次介绍针对数据并行和模型并行设计的各种分布式算法。本篇文章我们先看数据并行中最常用的同步并行SGD算法(也称SSGD)是如何在Spark平台上实现的。
2 同步并行SGD算法描述与实现
SSGD [1] 对应的伪代码可以表述如下:

其中,SSGD算法每次依据来自 个不同的工作节点上的样本的梯度来更新模型,设每个工作节点上的小批量大小为 ,则该算法等价于批量大小为
的小批量随机梯度下降法。
我们令 为逻辑回归问题的正则化经验风险。设 为权值(最后一维为偏置),样本总数为 , 为训练样本集。样本维度为 , (最后一维扩充), 。则 表示为:
这里
其梯度表示如下:
这里正则项的梯度 要分情况讨论:
(1) 不使用正则化
此时显然 。
(2) L2正则化( )
此时 。
(3) L1正则化( )
该函数不是在每个电都对 可导,只来采用函数的次梯度(subgradient)来进行梯度下降。 的一个比较好的次梯度估计是 。相比于标准的梯度下降,次梯度下降法不能保证每一轮迭代都使目标函数变小,所以其收敛速度较慢。
(4) Elastic net正则化( )
对 使用次梯度计算式, 使用其梯度计算式,得最终的梯度计算式为 。
我们约定计算第 个节点小批量 的经验风险的梯度 时不包含正则项的梯度,最终将 个节点聚合后再加上正则项梯度。
用PySpark对上述算法实现如下:
from sklearn.datasets import load_breast_cancer import numpy as np from pyspark.sql import SparkSession from operator import add from sklearn.model_selection import train_test_split from sklearn.metrics import accuracy_score n_slices = 3 # Number of Slices n_iterations = 300 # Number of iterations eta = 10 # iteration step_size, because gradient sum is divided by minibatch size, it shoulder be larger mini_batch_fraction = 0.1 # the fraction of mini batch sample lam = 0.001 # coefficient of regular term def logistic_f(x, w): return 1 / (np.exp(-x.dot(w)) + 1) def gradient(point: np.ndarray, w: np.ndarray): """ Compute linear regression gradient for a matrix of data points """ y = point[-1] # point label x = point[:-1] # point coordinate # For each point (x, y), compute gradient function, then sum these up # notice thet we need to compute minibatch size, so return(g, 1) return - (y - logistic_f(x, w)) * x def reg_gradient(w, reg_type="l2", alpha=0): """ gradient for reg_term """ assert(reg_type in ["none", "l2", "l1", "elastic_net"]) if reg_type == "none": return 0 elif reg_type == "l2": return w elif reg_type == "l1": return np.sign(w) else: return alpha * np.sign(w) + (1 - alpha) * w if __name__ == "__main__": X, y = load_breast_cancer(return_X_y=True) D = X.shape[1] X_train, X_test, y_train, y_test = train_test_split( X, y, test_size=0.3, random_state=0, shuffle=True) n_train, n_test = X_train.shape[0], X_test.shape[0] spark = SparkSession\ .builder\ .appName("SGD")\ .getOrCreate() matrix = np.concatenate( [X_train, np.ones((n_train, 1)), y_train.reshape(-1, 1)], axis=1) points = spark.sparkContext.parallelize(matrix, n_slices).cache() # Initialize w to a random value w = 2 * np.random.ranf(size=D + 1) - 1 print("Initial w: " + str(w)) for t in range(n_iterations): print("On iteration %d" % (t + 1)) w_br = spark.sparkContext.broadcast(w) (g, mini_batch_size) = points.sample(False, mini_batch_fraction, 42 + t)\ .map(lambda point: gradient(point, w_br.value))\ .treeAggregate( (0.0, 0),\ seqOp=lambda res, g: (res[0] + g, res[1] + 1),\ combOp=lambda res_1, res_2: (res_1[0] + res_2[0], res_1[1] + res_2[1]) ) w -= eta * g/mini_batch_size + lam * reg_gradient(w, "l2") y_pred = logistic_f(np.concatenate( [X_test, np.ones((n_test, 1))], axis=1), w) pred_label = np.where(y_pred < 0.5, 0, 1) acc = accuracy_score(y_test, pred_label) print("iterations: %d, accuracy: %f" % (t, acc)) print("Final w: %s " % w) print("Final acc: %f" % acc) spark.stop()我们尝试以 正则化、0.001的正则系数运行。初始权重如下:
Initial w: [ 0.09802896 0.92943671 -0.04964225 0.63915174 -0.61839489 0.86300117 -0.04102299 -0.01428918 0.84966149 0.50712175 0.10373804 -0.00943291 0.47526645 -0.19537069 -0.17958274 0.67767599 0.24612002 0.55646197 -0.76646105 0.86061735 0.48894574 0.87838804 0.05519216 -0.14911865 0.78695568 0.26498925 0.5789493 -0.20118555 -0.79919906 -0.79261251 -0.77243226]
最终的模型权重与在测试集上的准确率结果如下:
Final w: [ 2.22381079e+03 4.00830646e+03 1.34874211e+04 1.38842558e+04 2.19902064e+01 5.08904164e+00 -1.79005399e+01 -8.85669497e+00 4.28643902e+01 1.74744234e+01 2.24167323e+00 2.89804554e+02 -1.05612399e+01 -5.93151080e+03 1.60754311e+00 2.92290287e+00 2.46318238e+00 1.51092034e+00 4.23645852e+00 1.38371670e+00 2.20694252e+03 5.18743708e+03 1.32612364e+04 -1.39388946e+04 3.03078787e+01 4.41094696e+00 -2.24172172e+01 -5.27976054e+00 6.10623037e+01 1.83347648e+01 2.78974813e+02] Final acc: 0.912281
代码中有两个关键点,一个是
points.sample(False, mini_batch_fraction, 42 + t)。函数sample负责返回当前RDD的一个随机采样子集(包含所有分区),其原型为:RDD.sample(withReplacement: bool, fraction: float, seed: Optional[int] = None) → pyspark.rdd.RDD[T]
参数
withReplacement的值为True表示采样是有放回(with Replacement, 即replace when sampled out),为False则表示无放回 (without Replacement)。如果是有放回,参数fraction表示每个样本的期望被采次数,fraction必须要满足 ;如果是无放回,参数fraction表示每个样本被采的概率,fraction必须要满足位于 区间内。还有一个关键点是
.treeAggregate( (0.0, 0),\ seqOp=lambda res, g: (res[0] + g, res[1] + 1),\ combOp=lambda res_1, res_2: (res_1[0] + res_2[0], res_1[1] + res_2[1]) )该函数负责对RDD中的元素进行树形聚合,它在数据量很大时比
reduce更高效。该函数的原型为RDD.treeAggregate(zeroValue: U, seqOp: Callable[[U, T], U], combOp: Callable[[U, U], U], depth: int = 2) → U[source]
其中
zeroValue为聚合结果的初始值,seqOp函数用于定义单分区(partition)做聚合操作的方法,该方法第一个参数为聚合结果,第二个参数为分区中的数据变量,返回更新后的聚合结果。combOp定义对分区之间做聚合的方法,该方法第一个参数为第二个参数都为聚合结果,返回累加后的聚合结果。depth为聚合树的深度。我们这里
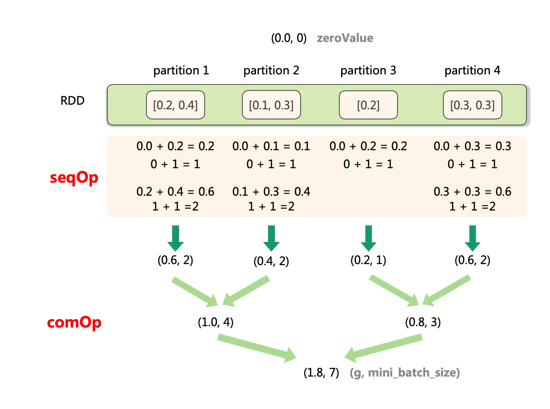
treeAggregate想要聚合得到一个元组(g, mini_batch_size),g为所有节点样本的随机梯度和,mini_batch_size为所有节点所采的小批量样本之和,故我们将聚合结果的初始值zeroVlue初始化为(0,0, 0)。具体的聚合过程描述如下:-
对每个partition:
a. 初始化聚合结果为
(0.0, 0)。b. 对当前partition的序列元素,依次执行聚合操作
seqOp。c. 得到当前partition的聚合结果
(partition_sum, partition_count)。 -
对所有partition:
a. 按照树行模式合并各partition的聚合结果,合并方法为
combOp。b. 得到合并结果
(total_sum, total_count)。
形象化地表示该聚合过程如下图所示:
3 算法收敛性及复杂度分析
3.1 收敛性和计算复杂度
假设目标函数 是 -强凸函数,并且 光滑,如果随机梯度的二阶矩有上界,即 ,当步长 时,对于给定的迭代步数 ,SGD具有 的次线性收敛速率:
而这意味着SGD的迭代次数复杂度为 ,也即 轮迭代后会取得 的精度。
尽管梯度的计算可以被分摊到个计算节点上,然而梯度下降的迭代是串行的。每轮迭代中,Spark会执行同步屏障(synchronization barrier)来确保在各worker开始下一轮迭代前 已被更新完毕。如果存在掉队者(stragglers),其它worker就会空闲(idle)等待,直到下一轮迭代。故相比梯度的计算,其迭代计算的“深度”(depth)是其计算瓶颈。
3.2 通信复杂度
map过程显然是并行的,并不需要通信。broadcast过程需要一对多通信,并且reduce过程需要多对一通信(都按照树形结构)。故对于每轮迭代,总通信时间按
增长。
这里 为除去driver节点的运算节点个数, 是节点之间的通信延迟。 是节点之间的通信带宽。
是每轮通信中节点间传输的信息大小。故消息能够够以
的通信轮数在所有节点间传递。
-
-
相关阅读:
wordpress全站开发指南-面向开发者及深度用户(全中文实操)--wordpress是什么
【代码精读】ATF的异常向量表
爬取任意百度贴吧评论(可直接Copy)
【算法 - 动态规划】找零钱问题Ⅰ
MyBatis执行SQL语句的两种方式
速锐得解码特斯拉Model Y整车网关电路及CAN通信协议DBC控制策略
猜名次(附完整代码)
java语法复习:注解
Vue中组件化编码使用、实现组件之间的参数传递(实战练习二)
尝试FreeBSD下安装ollama
- 原文地址:https://blog.csdn.net/JavaMonsterr/article/details/125506024
