-
深度剖析monai(一) Data和Transforms部分
前言
最近没什么事,研究了一下
monai,它是一个优秀的基于pytorch的医学深度学习框架,包括了Tansformers(负责数据的读取和数据增强)、Loss functions(包含常见的损失函数)、Network architectures(实现了常用的医学图像分割model)、Metrics(验证时的评估函数)、Optimizer(优化器)、Data(Dataset和DataLoader)等几个常用的深度学习组件。通过这些组件,我们可以定义好自己的model后,方便地进行训练。在这篇文章里咱们就想先来说一下monai的Transformers数据增强组件和Data组件。简单使用
首先我们回忆一下,大家平时在写训练逻辑时肯定是先定义
Dataset类,Dataset类可以通过调用自身的__getitem__方法返回数据,此时数据的维度为[C, H, W, D],C代表通道数,H,W,D分别代表高、宽、深(三维时才有深度这个维度)。然后通过DataLoader类多次调用Dataset类的__getitem__生成多个样本,将其组合起来,此时返回的数据维度为[B, C, H, W, D], B代表batch_size大小。
monai加载数据时也是按照这个思路来写的,先定义Dataset,再使用DataLoader。在定义Dataset的时候,我们可以向其传入一系列monai自定义的数据增强方法,比如数据的读取、数据的随机旋转、裁剪、翻转、切分patch,归一化、标准化、转为tentor等,这些数据增强操作统一被写到了monai.Transformers模块中。和Pytorch一样,这些数据增强操作统一可以由monai.transforms.Compose类包裹起来,这样数据就可以自动流式处理了,减少了代码量。
下面我们来看一个简单的例子,是直接调用的不带字典的数据增强方法,但是这种方式不能用dataloader包装。from monai import transforms, data # 定义数据集列表 data_list = ["F:/9.4Data/ski10/image/image-001.nii.gz", "F:/9.4Data/ski10/image/image-002.nii.gz", "F:/9.4Data/ski10/image/image-003.nii.gz", "F:/9.4Data/ski10/image/image-004.nii.gz" ] # 定义数据增强操作 train_transform = transforms.Compose([ transforms.LoadImage(), # 加载图像,底层会根据文件名来选择对应的数据读取器,nii结尾的文件默认用ITK读取数据 transforms.AddChannel(), # 增加通道,monai所有Transforms方法默认的输入格式都是[C, W, H, ...],第一维一定是通道维 transforms.ToTensor() # 将numpy转为tensor,注意和pytorch不一样的是,此操作并不包含归一化步骤 ])- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
其实好多数据增强操作都是在
image和label上同时进行的,比如裁剪和旋转。和pytorch数据增强方法torchvision.transform不同的是,monai中每一个数据增强方法类都对应一个字典增强类,以d结尾。这样的字典增强类以一个字典对象作为输入,如{"image": "", "label": ""},构造时可以通过keys参数指定在image或label上进行操作,在这个类内部通过__call__()方法进行相应的数据增强操作,具体可查看源码。最终的输出也是一个字典,该字典所包含的key和传入的key值一致。下面是一个简单的例子:from monai import transforms, data data_list = [{"image": "F:/9.4Data/ski10/image/image-001.nii.gz", "label": "F:/9.4Data/ski10/label/labels-001.nii.gz"}, {"image": "F:/9.4Data/ski10/image/image-002.nii.gz", "label": "F:/9.4Data/ski10/label/labels-002.nii.gz"}, {"image": "F:/9.4Data/ski10/image/image-003.nii.gz", "label": "F:/9.4Data/ski10/label/labels-003.nii.gz"}, {"image": "F:/9.4Data/ski10/image/image-004.nii.gz", "label": "F:/9.4Data/ski10/label/labels-004.nii.gz"} ] train_transformd = transforms.Compose([ # 加载图像,会默认根据文件后缀选择相应的读取类 transforms.LoadImaged(keys=["image", "label"]), # 增加通道维度 transforms.AddChanneld(keys=["image", "label"]), # 根据前景裁剪,会把前景部分裁剪出来 transforms.CropForegroundd(keys=["image", "label"], source_key="label", margin=5), # 转化为tensor,这里没有做归一化,只是单纯地转为tensor的float transforms.ToTensord(keys=["image", "label"]) ])- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
其他数据增强方法
见官方文档(后面有时间补充)
这里说一个特殊的数据增强方法transforms.RandCropByPosNegLabeld。
功能:主要是在原图上按照正负样本比例随机裁剪出指定个固定大小的patch块,适应于正负样本不平衡的情况,通过此操作可以平衡样本,也可以切出固定大小的patch块送入网络中进行训练。
其功能倒是没什么可说的,主要是前面我们说过所有的数据增强类输入输出都是一个字典对象,每个字典对象代表一个训练对象,而这个类因为可以切出好几个patch,所以它输出的是一个包含多个字典对象的列表,如下图所示。
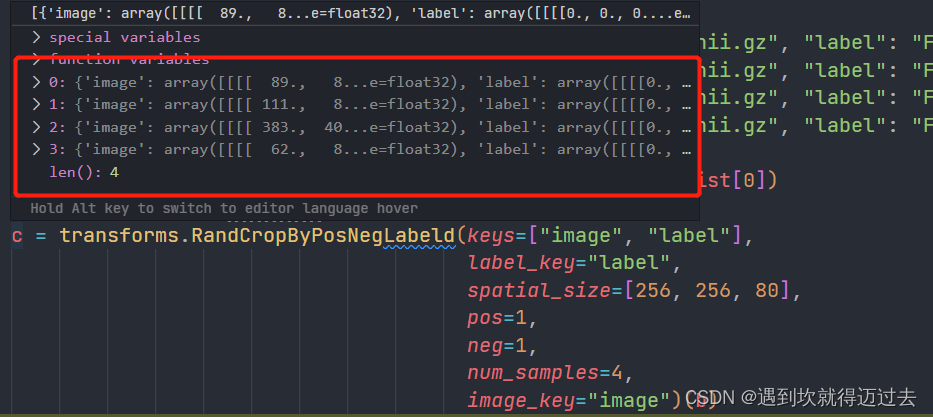
那么,问题来了,此数据增强类输出的多个字典是如何输入下一个数据增强类的呢(数据增强器的输入都应该是字典啊,而不是列表)?
在看了Compose类的源码之后,我发现Compose在把上一个数据增强类输出的结果送入下一个数据增强类的时候,会做一个判断:如果是列表,则循环进行输入;如果是字典,则直接输入。
这样循环输入后相当于多了一个batch_size维度,后期也印证了我这个想法,当我dataloader中的batch_size=2, 而transforms.RandCropByPosNegLabeld类中num_samples=4时每一个迭代其batch_size=2*4=8from monai import transforms, data data_list = [{"image": "F:/9.4Data/ski10/image/image-001.nii.gz", "label": "F:/9.4Data/ski10/label/labels-001.nii.gz"}, {"image": "F:/9.4Data/ski10/image/image-002.nii.gz", "label": "F:/9.4Data/ski10/label/labels-002.nii.gz"}, {"image": "F:/9.4Data/ski10/image/image-003.nii.gz", "label": "F:/9.4Data/ski10/label/labels-003.nii.gz"}, {"image": "F:/9.4Data/ski10/image/image-004.nii.gz", "label": "F:/9.4Data/ski10/label/labels-004.nii.gz"} ] train_transformd = transforms.Compose([ # 加载图像,会默认根据文件后缀选择相应的读取类 transforms.LoadImaged(keys=["image", "label"]), # 增加通道维度 transforms.AddChanneld(keys=["image", "label"]), # 根据前景裁剪,会把前景部分裁剪出来 transforms.CropForegroundd(keys=["image", "label"], source_key="label", margin=5), # 按比例裁剪背景和前景, 如果num_samples不为1,则会将指定值个的裁剪后的样本,放入list中返回,最后dataloader会拼起来 # 比如这里num_samples=4, dataloader的batch_size=2,那么最终每次迭代会返回4*2=8个样本,即bacth_size=8 # spatial_size超过原本数据大小后会报错 # 自定义归一化数据 Uniformd(keys=["image"]), transforms.RandCropByPosNegLabeld(keys=["image", "label"], label_key="label", spatial_size=[256, 256, 80], pos=1, neg=1, num_samples=4, image_key="image"), # 使用插值算法放缩到固定尺寸, size_mode='all'时表示不会保留原有的长宽比 # transforms.Resized(keys=["image", "label"], spatial_size=[256, 256, 100], size_mode="all", mode=["area", "nearest"]), # 归一化放缩像素值,比如放缩到0-1;这里并不适用ski10数据集,因为si10数据集中每个样本的取值范围不一样,所以我们自定义了 # transforms.ScaleIntensityRanged(keys=["image", "label"], # a_min=0, a_max=5000, # b_min=0, b_max=1), # 转化为tensor,这里没有做归一化,只是单纯地转为tensor的float transforms.ToTensord(keys=["image", "label"]) ]) train_dataset = data.Dataset(data=data_list, transform=train_transformd) train_dataLoader = data.DataLoader(dataset=train_dataset, batch_size=2, shuffle=True, num_workers=2) print('训练数据集数量', len(train_dataset)) for batch_data in train_dataLoader: image, label = batch_data["image"], batch_data["label"] print('image shape:', image.shape, 'label shape:', label.shape, 'max:', torch.max(image), 'min:', torch.min(image))- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
自定义数据读取器
在上面例子中,我们读取数据
nii是使用transforms.LoadImaged(keys=["image", "label"])方法根据文件名读取图像的,可是我们有没有想过内部到底是如何读取数据的呢?
原来这个类有一个reader参数,这是一个读取数据的类,内部就是通过调用Reader类来根据文件名读取图像的。那么我们有定义过Reader类吗,答案是没有,官方已经写好了,读取nii或nii.gz会调用ITKReader类,读取png、jpeg会使用PILReader。
如果我们想要定义自己的数据读取器,应该怎么做呢?
答案是继承data.ImageReader类,实现get_data,read,verify_suffix方法即可(具体返回值可看官方文档),这里我在ITKReader的基础上,自定义了一个归一化类,它可以计算最大值和最小值,从而将体素值归一化到[0, 1], 代码如下,使用的时候直接作为参数传入即可:from monai import transforms, data # 自定义读取器的get_data方法,注意读取器处理的对象是一个nii文件,他并不知道是image还是label,是在loadImage中调用的 class MyReader(data.ITKReader): def __init__(self, channel_dim: Optional[int] = None, series_name: str = "", reverse_indexing: bool = False, series_meta: bool = False, **kwargs): super().__init__(channel_dim, series_name, reverse_indexing, series_meta, **kwargs) def get_data(self, img): image, meta = super().get_data(img) image = np.array(image) # 只对image进行归一化操作 if np.max(image) != 4: max_value, min_value = np.max(image), np.min(image) # 根据最大最小值进行归一化放缩到0-1 image = (image - min_value) / (max_value - min_value) # print(np.max(image), np.min(image)) return image, meta # 使用自定义读取器类 # 加载图像,会默认根据文件后缀选择相应的读取类 transforms.LoadImaged(keys=["image", "label"], reader=MyReader)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
自定义数据增强操作
还是回到刚刚那个问题,我想要根据每个nii文件的最大值和最小值进行归一化,除了在读取数据时提前操作,还有别的办法吗?
当然有!直接定义一个自己的归一化数据增强类Uniformd岂不是更方便。
那应该如何定义呢?
官方并没有说,不过我看源码,首先是要继承’MapTransform, InvertibleTransform’两个类,然后实现__call__(数据增强正向调用时用)和inverse方法(增强后的数据返回原始数据,好像几乎用不到)即可。
强调一下,因为monai中每一个字典增强类都对应一个不带字典的同样功能的数据增强类,所以官方内部实现时是直接实例化了一个,然后在内部调用。
而我自己定义的为了简单,是直接写了个函数来完成对于操作。
代码如下:class Uniformd(MapTransform, InvertibleTransform): """ 归一化值 """ def __init__( self, keys, dtype: Optional[torch.dtype] = None, device: Optional[torch.device] = None, wrap_sequence: bool = True, allow_missing_keys: bool = False, ) -> None: super().__init__(keys, allow_missing_keys) def __call__(self, data): d = dict(data) for key in self.key_iterator(d): self.push_transform(d, key) d[key] = self.uniform(d[key]) return d def uniform(self, data): max_value, min_value = np.max(data), np.min(data) # 根据最大最小值进行归一化放缩到0-1 data = (data - min_value) / (max_value - min_value) return data def inverse(self, data): d = deepcopy(dict(data)) for key in self.key_iterator(d): # Create inverse transform # inverse_transform = ToNumpy() # Apply inverse d[key] = self.uniform(d[key]) # Remove the applied transform self.pop_transform(d, key) return d- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
之后就可以像官方数据增强类一样初始化调用使用了
# 自定义归一化数据 Uniformd(keys=["image"])。总结
折腾了两天,终于解决了自己的诸多疑惑,看来最好的学习资料还是源码和官网,大家善加利用 !
monai官网链接 -
相关阅读:
可变参数函数
GO语言学习笔记(与Java的比较学习)(三)
QSPI介绍
Springcloud之OAuth2
【测开求职】面试题:计算机网络 精简版整理
基于LSM树的存储机制简述
产品帮助中心必备的6大常规功能,你知道几个?
Maven面试题
七牛云创建存储空间并绑定自定义域名-https协议
修正两个shapefile之间的数字化错误
- 原文地址:https://blog.csdn.net/qq_43705697/article/details/125429634