-
基于卷积神经网络ResUnet 多模态融合实验、BraTS 3d数据集(nii.gz)4模态融合分割
1、前言
之前介绍了unet对BraTS 3d数据集的2d图片分割,实现思路如下:
1、对BraTS 3d数据集进行切片,沿着某个模态的横断面切割
2、划分数据集、包括训练集、验证集等等
3、网络训练
4、评估模型性能等等
具体的可以参考本文:基于Unet的BraTS 3d 脑肿瘤医学图像分割,从nii.gz文件中切分出2D图片数据_brats2020unet分割-CSDN博客
看了评论和私信,不少人问可不可以将四个模态融合进去,而非单独某个模态分割。
因为多模态本人没有接触过,不过要是仅仅把四个模态融合还是很简单的
一般CNN网络的输入是3通道的RGB,而我们想要把BraTS 3d四个模态融合,其实就是变成
了4个通道的输入而已
这里不用纠结4通道的数据是啥,反正是四种形态,每个形态都是h*w的灰度图像而已事实上,CNN的隐藏层都把数据卷积成几百个维度了,都是语义信息罢了
2、数据集介绍
BRATS 脑肿瘤分割数据(brain tumor segmentation challenge,BraTS Chanllenge)
标签如下:

这里原始数据集要很多G,这里为了方便仅用十个样本来实验
数据是nii格式,维度如下:数据、gt的维度

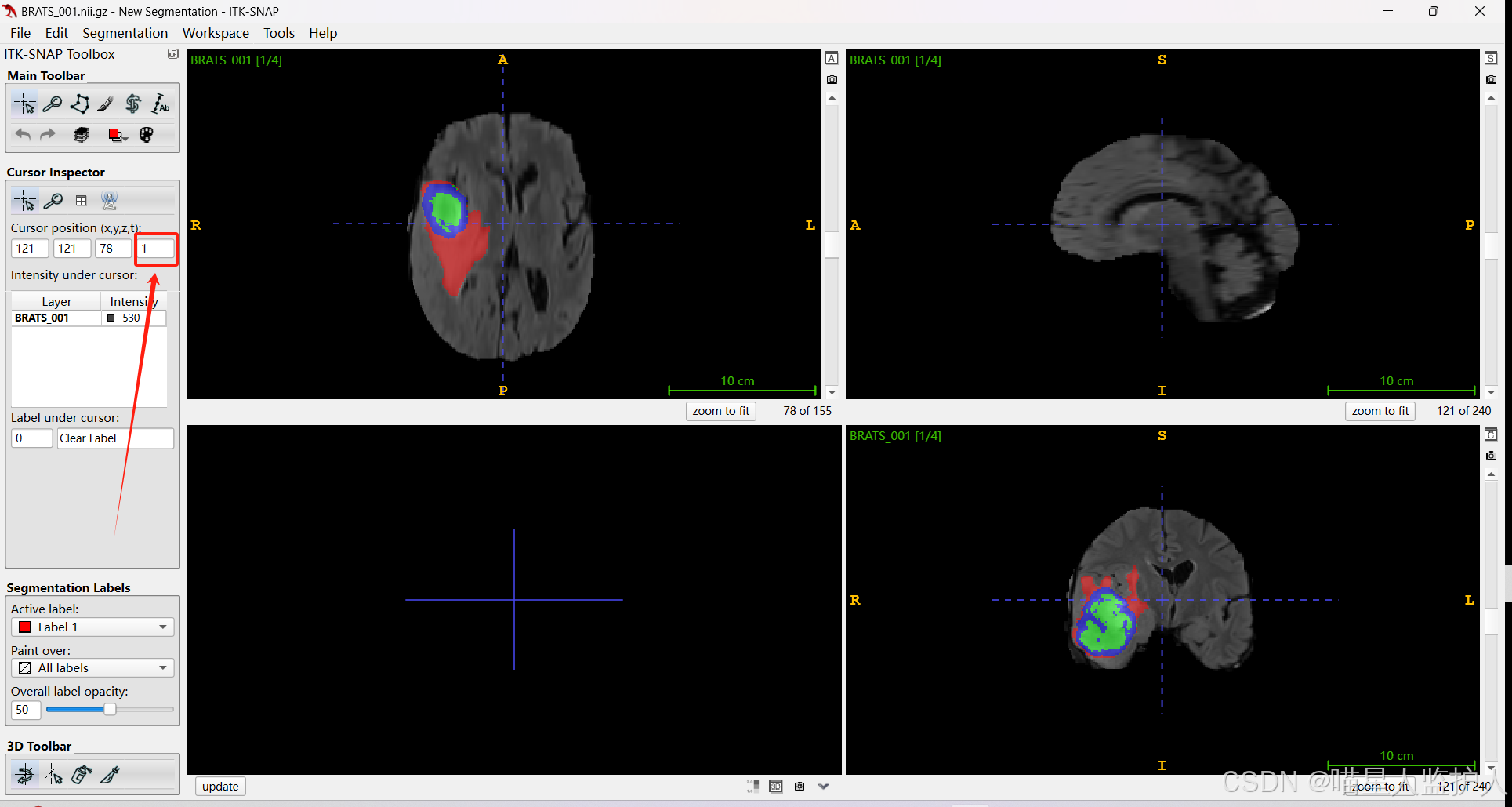
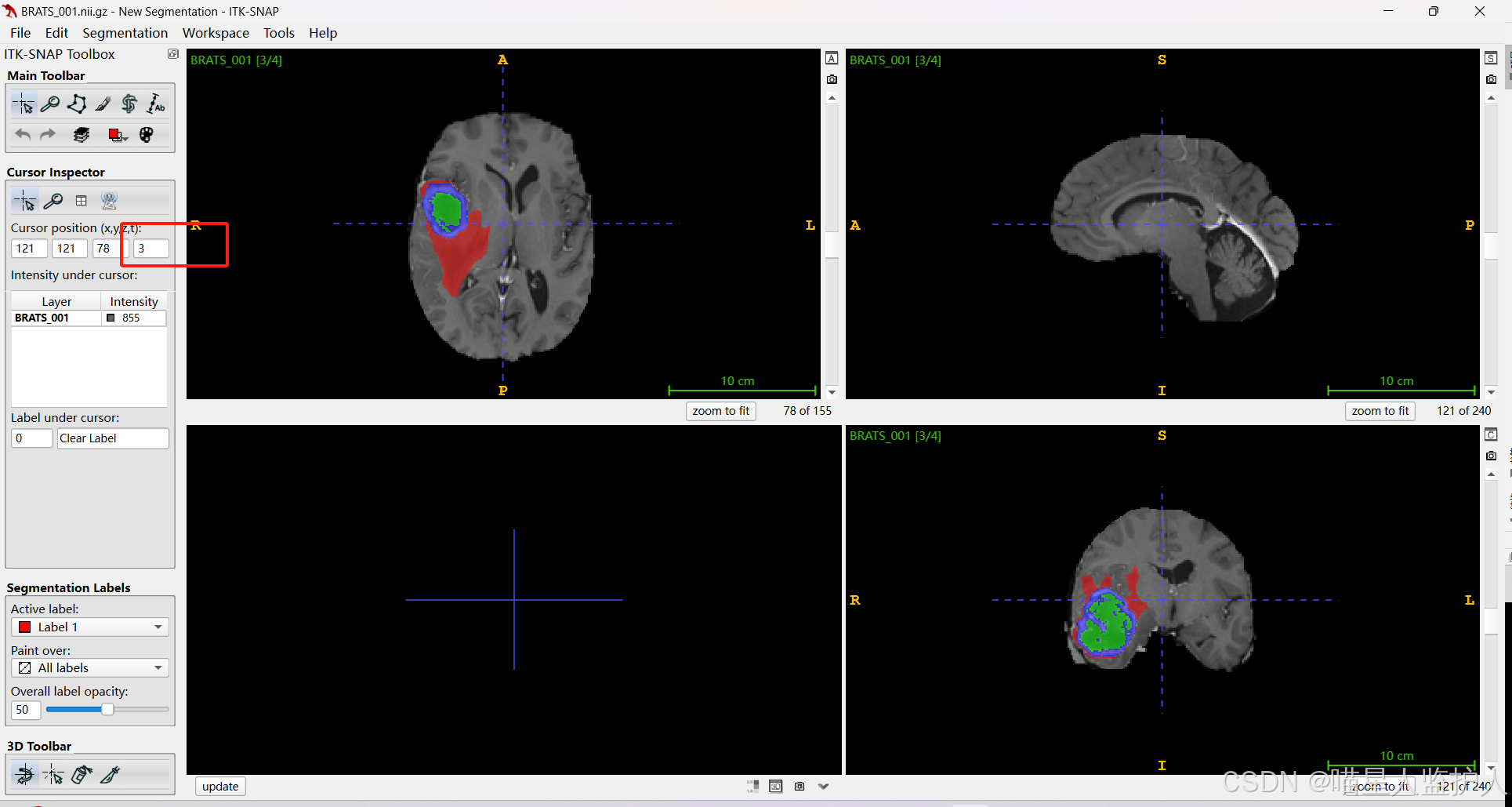
可以用ITK-SNAP打开,箭头所指的就是不同模态,可以更换
3、数据集转换
因为数据的维度是这种的
data的维度是4,其中分别为4个模态、x、y、z方向的size
gt 是没有模态的,因为病变区域是不变的

需要注意的是,我们实验的是多模态的信息融合,而非3D的语义分割,请记住这一点,防止下面会混淆
事实上,语义分割的2D领域很多,而3D貌似没有2d分割实用
虽然3d分割要比2d分割效果好,毕竟3d能把握数据的空间体素信息。类似于卷积相比于线性的图像识别,卷积感受野是图像的一个patch,而线性分类仅仅把握像素点。毕竟一般图像来看,像素点的灰度值往往与周围邻域的区域有关
但是3d分割消耗的资源远超2d分割,而且3d的数据采集也很麻烦。等等原因,一般来说我们接触的都是2d图像的分割,基本上sam模型也是以2d图像分割为主
所以,我们想要把BRATS数据集的 (4, 155, 240, 240) 维度先分片,这里还是沿着x轴,也就是155这个方向,所以会变成 (4, 240, 240)的shape
一般的图像维度是(240,240,3)------->3 RGB
这里的4是四种模态,也可以理解成四个channel,每一个都是240*240的灰度图像
本来写了一个dataset直接把nii.gz文件切片后输入网络,不过考虑到对gpu资源可能消耗过大,这里就先生成数据。但是jpg、png图像都是3通道的,不能保存4通道的数据,所以这里用npy保存
3.1 nii.gz 转成 npy 文件
这里需要把一开始的nii.gz的BRATS数据集摆放成这样:

生成的npy如下,这里做了归一化的操作

3.2 npy 文件可视化
可以用下面代码对 npy 可视化
- import numpy as np
- import matplotlib.pyplot as plt
- im_path = 'ResUnet/data_npy/train/images/50.npy'
- mk_path = 'ResUnet/data_npy/train/labels/50.npy'
- image = np.load(im_path)
- mask = np.load(mk_path)
- print('image:',image.shape)
- print('mask:',mask.shape,np.unique(mask))
- plt.figure(figsize=(12,8))
- plt.subplot(2,2,1)
- plt.imshow(image[0],cmap='gray')
- plt.title('FLAIR')
- plt.subplot(2,2,2)
- plt.imshow(image[1],cmap='gray')
- plt.title('T1w')
- plt.subplot(2,2,3)
- plt.imshow(image[2],cmap='gray')
- plt.title('t1gd')
- plt.subplot(2,2,4)
- plt.imshow(image[3],cmap='gray')
- plt.title('T2w')
- plt.show()
- plt.imshow(mask)
- plt.show()
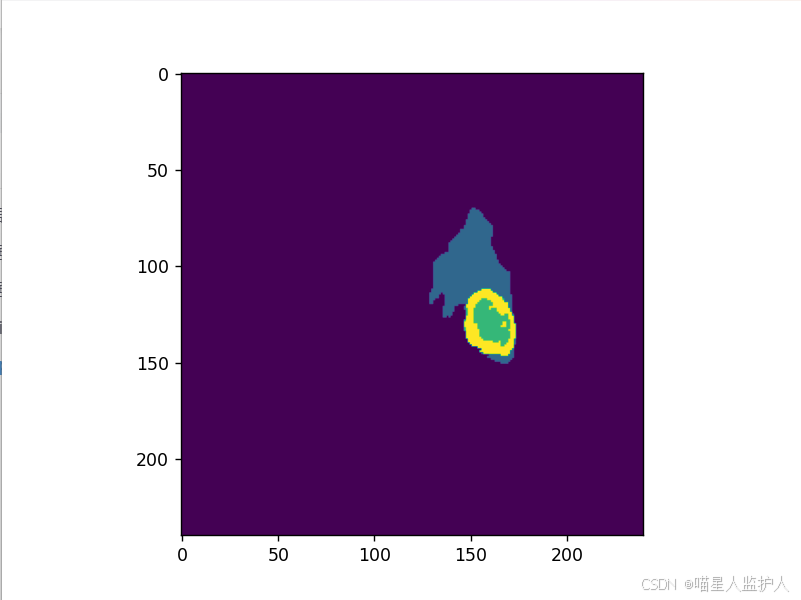
如下:

可以看到image的维度是3维的
数据

4、ResUnet 多模态融合分割
这里采用的的ResUnet网络
需要注意的是,这里的网络可以任意更换,只要网络的输入维度和输出维度改成4即可
- # 实例化模型
- model = UResnet(num_classes=args.nc,input_channels=args.input)
- model.to(device)

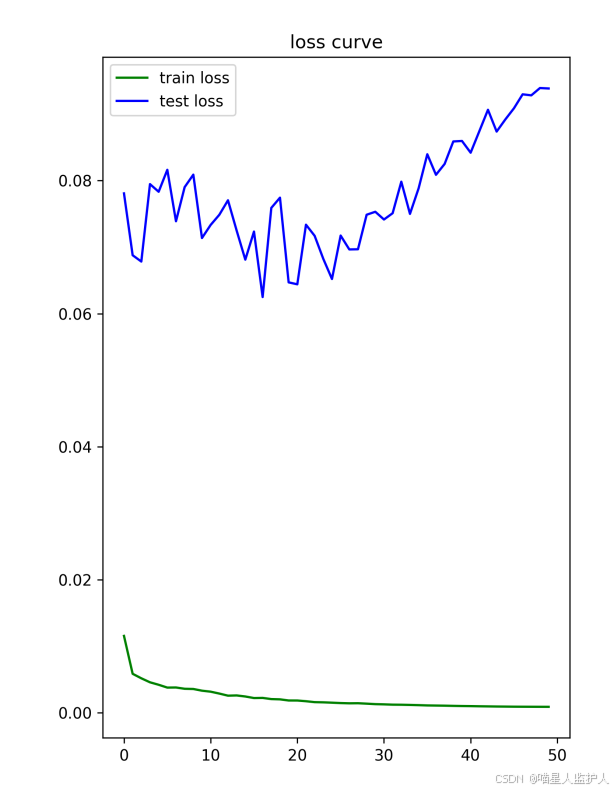
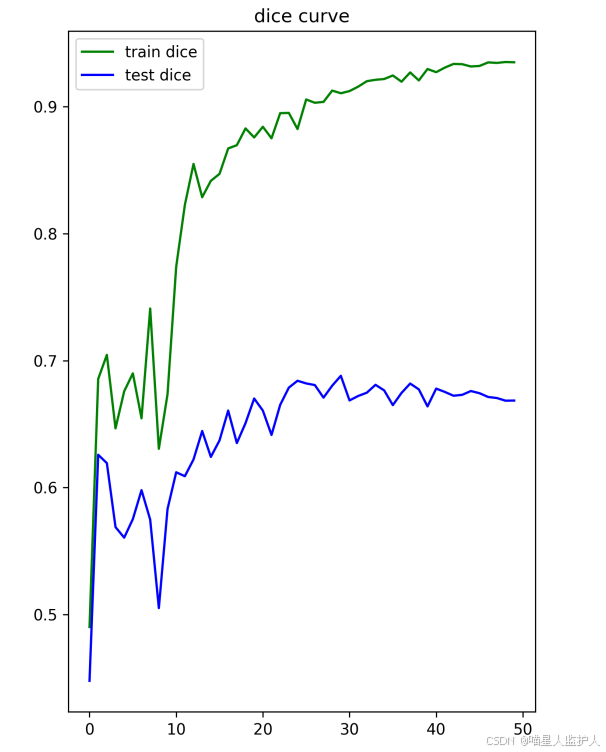
这里训练了50个epoch,训练集的dice有0.94左右,测试集的dice有0.67左右

看样子,确实融合分割的效果好,不过这里只用了十个数据集,导致数据不足,所以过拟合了
5、推理
推理的话,这里输入是nii.gz文件,生成的也是nii.gz文件
实现逻辑是把nii文件沿着横断面逐层切片,然后推理完把结果融合成nii文件,输出
推理的结果:
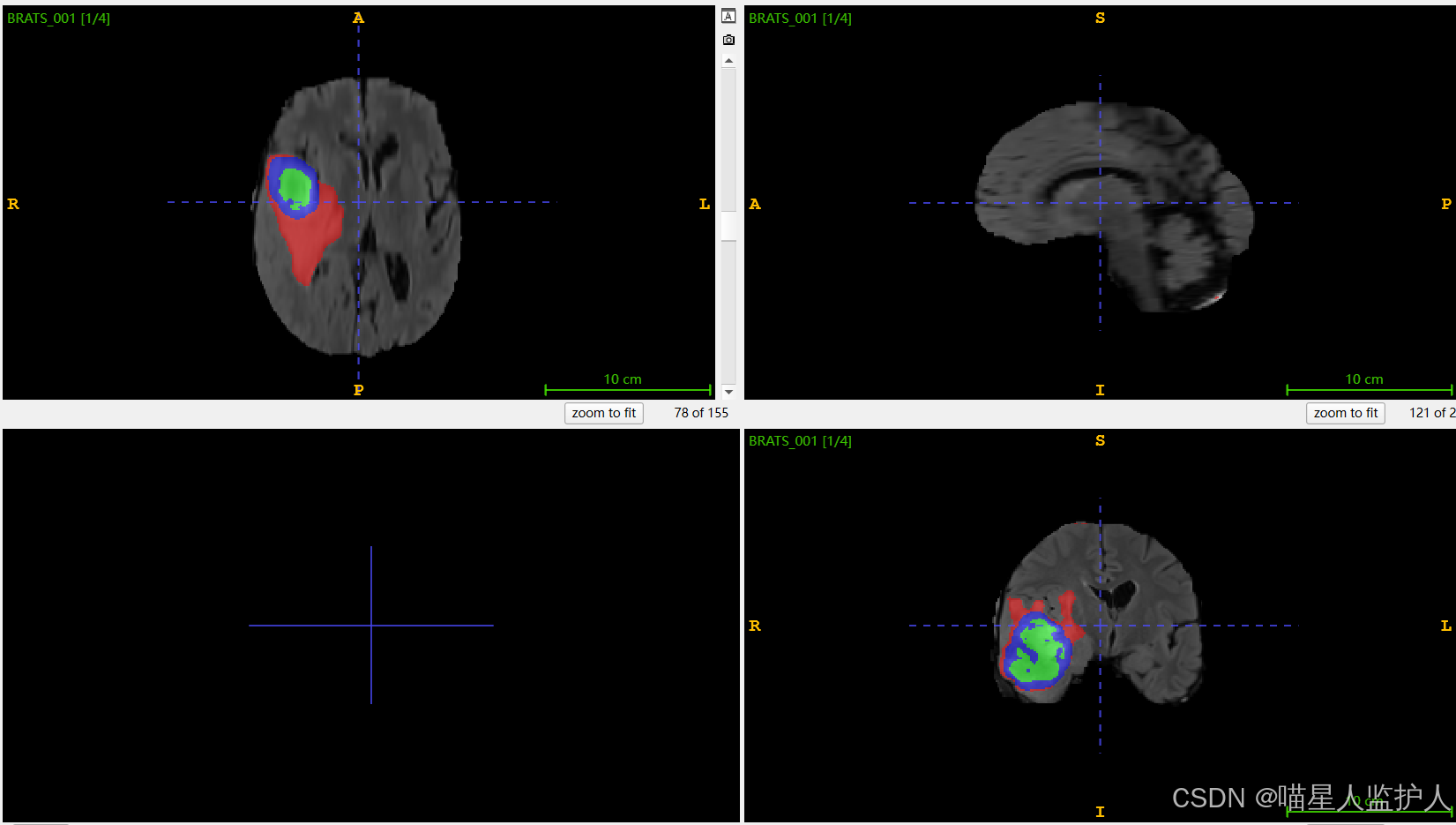
真实标签:

6、下载、其他
实验下载:ResUnet网络对BRATS脑肿瘤多模态融合的语义分割项目实现资源-CSDN文库
ResUnet网络毕竟很久了,换成SwinUnet、TransUnet、DenseUnet之类的效果估计会好点
或者对数据进行增广、扩充数据集等等
像这种尺度的融合很简单,不管是灰度图、RGB图像还是4通道的模态融合,其实就是数据的为敌就是(x,h,w)罢了。几通道就是x变成几罢了,然后在dataset里面把加载数据的代码改一下,网络的输入部分其实就是改个数字。
从这次实验,我也有很多启发,sam分割不考虑大模型其实就是加了个bbox的维度,后面可以尝试尝试把unet和sam进行融合做医学图像语义分割。
-
相关阅读:
mysql基础
MyBatisPlus-标准数据层CRUD功能制作
byte数据与Int和bit转换类
python代码实现论文〖文献引用顺序〗修改校对
Chatgpt版本的opencv安装教程
C++ 运算符重载
GEE4:FCMASK算法进行去云处理
pc与android设备进行通信
Zotero详细功能补充!熟练使用!【进阶版,持续更新】
VMware安装Kali操作系统(全网最详细不接受任何反驳)
- 原文地址:https://blog.csdn.net/qq_44886601/article/details/140987576