-
NBT:快准全!geNomad——宏病毒组鉴定新工具

期刊:nature biotechnology
影响因子:46.9
发表时间:2023年9月
在测序数据中识别可移动遗传元件对了解其多样性、生态学、生物技术应用和对公共健康的影响至关重要。本研究开发了geNomad——可同时识别和注释测序数据中的质粒和病毒序列。geNomad使用227897个标记蛋白图谱的数据集来提供病毒基因组的功能基因注释和分类匹配。geNomad还使用条件随机场模型高精度检测整合到宿主基因组中的前病毒。在线使用网址:https://portal.nersc.gov/genomad.

图1 geNomad网页主页面
1、鉴定和注释质粒和病毒的框架
geNomad采用混合方法进行质粒和病毒鉴定,结合了无比对分类器(序列分支)和基于基因的分类器(标记分支),通过利用两种分类器的优势来提高分类性能。
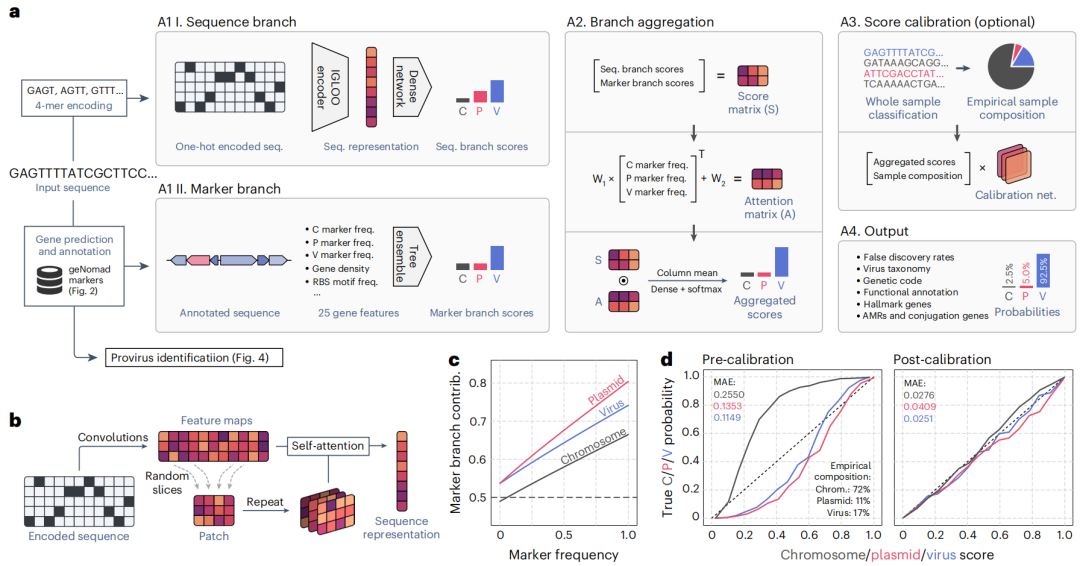
图2 鉴定和注释质粒和病毒的框架
2、生成蛋白质图谱数据集,用于序列分类和蛋白质注释
geNomad使用染色体、质粒或病毒特异性的227,897个蛋白质谱的标记物集对基因进行分类,并为处理的序列提供功能信息。
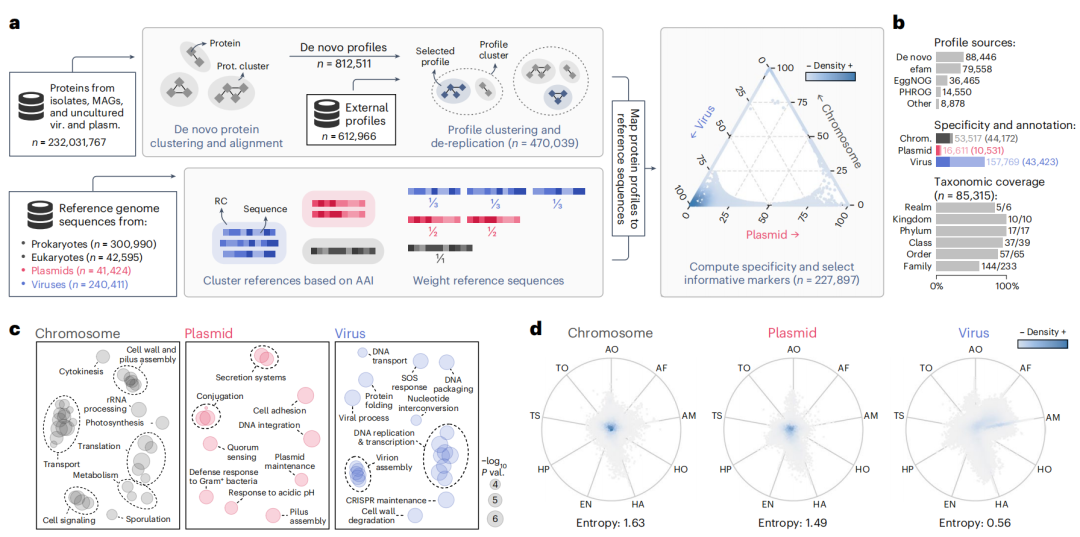
图3 蛋白质图谱数据集
3、geNomad可准确识别质粒和病毒
与其他工具相比,geNomad在质粒和病毒分类任务中,在所有序列长度范围内均表现出较好的总体分类性能,短序列中尤其明显。
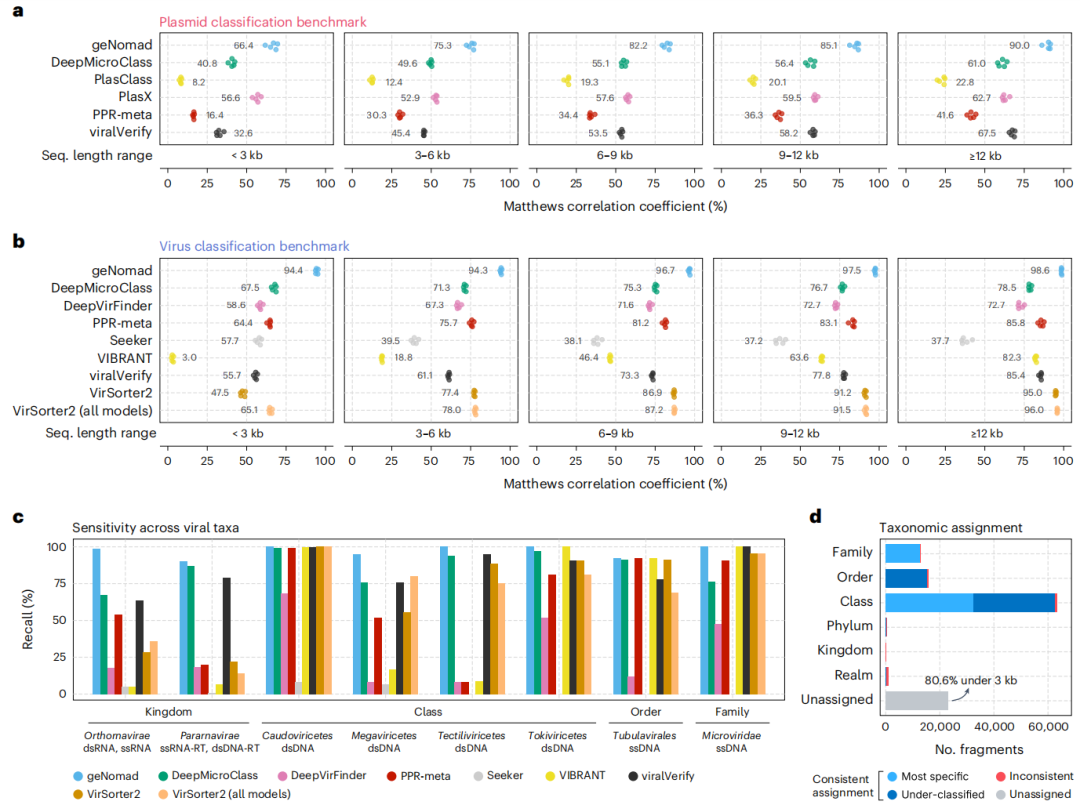
图4 基因组准确地识别病毒和质粒
4、敏感、精确的鉴定前噬菌体
其他流行工具(Phigaro39、VIBRANT和VirSorter2)进行了比较,geNomad性能更优,划分的前噬菌体更完整,污染水平较低。
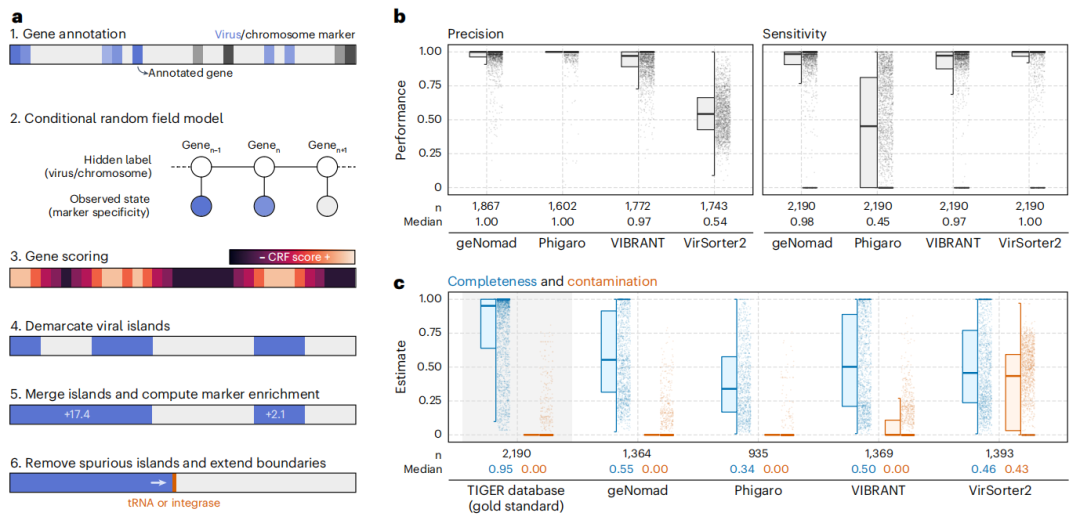
图5 使用标记信息来划分原噬菌体的边界
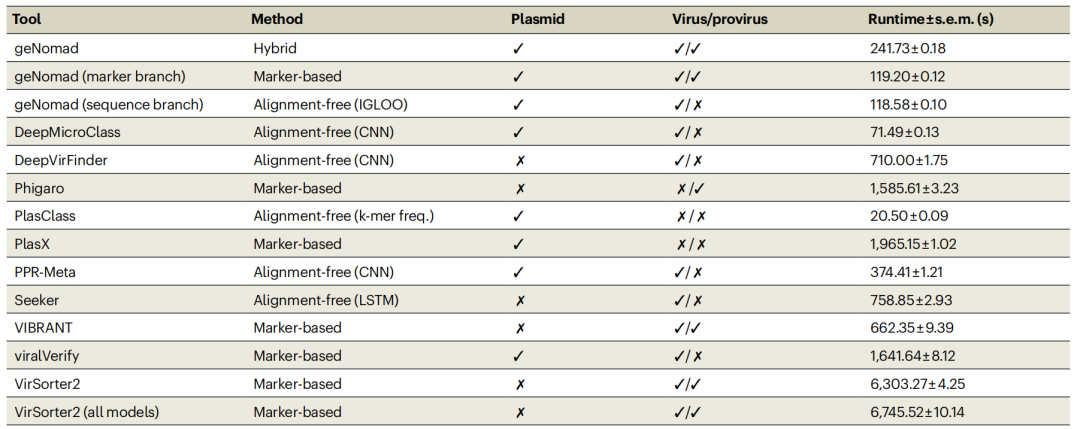
5、geNomad运行速度快,可对大型数据集进行分析
其他流行工具相比,geNomad运行速度快,并且geNomad的标记和序列分支可以独立运行,可以将运行时间减少一半。geNomad最近被用于处理超过2.7万亿个碱基对的测序数据,从而发现了数百万种包含在IMG/VR和IMG/PR数据库中的病毒和质粒。
表1 质粒和病毒鉴定工具的分类方法和平均运行时间
6、geNomad可以发现RNA病毒和巨型病毒
开发者使用病毒的最新知识训练geNomad,大大提高了geNomad对RNA病毒和巨型病毒的鉴定能力。
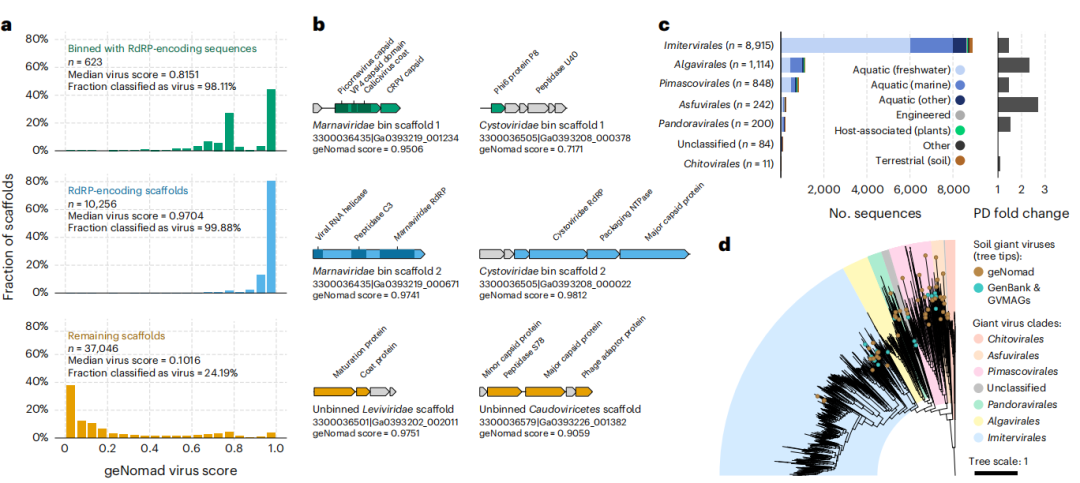
图6 环境测序数据中发现RNA病毒和巨型病毒
结论
geNomad结合基因信息和深度神经网络信息来识别质粒和病毒序列,使用了超过200,000个标记蛋白谱的数据集对病毒基因组的功能基因进行注释和分类分配。使用条件随模型,高精度地检测整合到宿主基因组中的前噬菌体。在基准测试中,各种性能大大优于其他工具。
凌恩生物宏病毒分析流程已更新使用geNomad,用于宏病毒数据中前噬菌体的鉴定。更多测序项目了解,请关注凌恩生物公众号,紧跟CNS步伐,用最新的流程助力您的科研!
参考文献
Identification of mobile genetic elements with geNomad.nature biotechnology,2023.
-
相关阅读:
【liunx】进程的状态
spdx-sbom-generator使用记录
JS高级(四):正则表达式、常见的特殊字符、案例、预定义类、正则替换
App移动端测试(6)—— 应用程序(apk)包管理与activity
qt creater11 翻译国际化教程教程:
ESP8266-Arduino编程实例-BME680环境传感器驱动
PHP写用户注册、登录和密码重置功能
Effective Objective-C 学习(三)
【Mysql】第3篇--数据库约束
pytorch无法使用cuda
- 原文地址:https://blog.csdn.net/SHANGHAILINGEN/article/details/133942282