-
基于R的linkET包qcorrplot可视化Mantel test相关性网络热图分析correlation heatmap
写在前面
需求是对瘤胃宏基因组结果鉴定到的差异菌株与表观指标、瘤胃代谢组、血清代谢组、牛奶代谢组中有差异的部分进行关联分析,效果图如下:

数据准备
逗号分隔的csv格式文件,两个表格,一个是每个样本对应的表观指标数据,另一个是每个样本对应的菌群丰度,我这里用的是genus水平
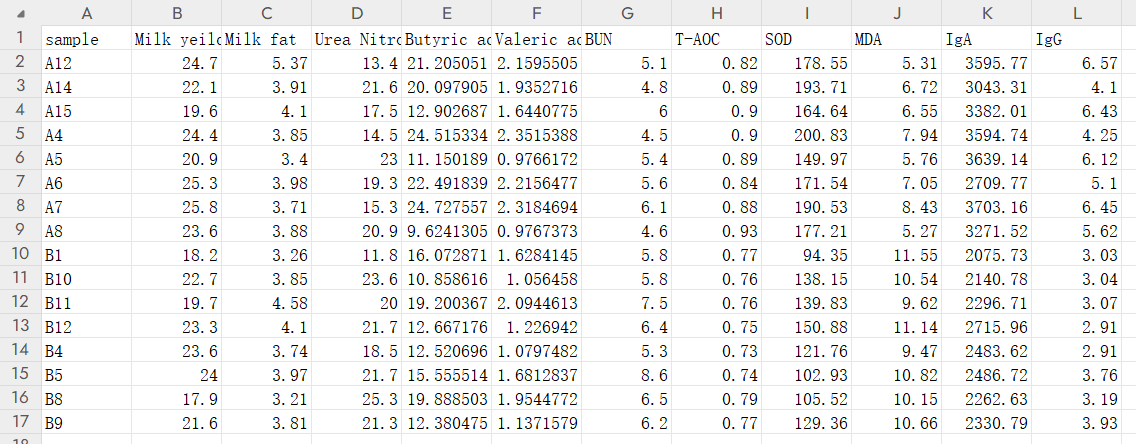
- 需要关联的表观数据
rumen.csv
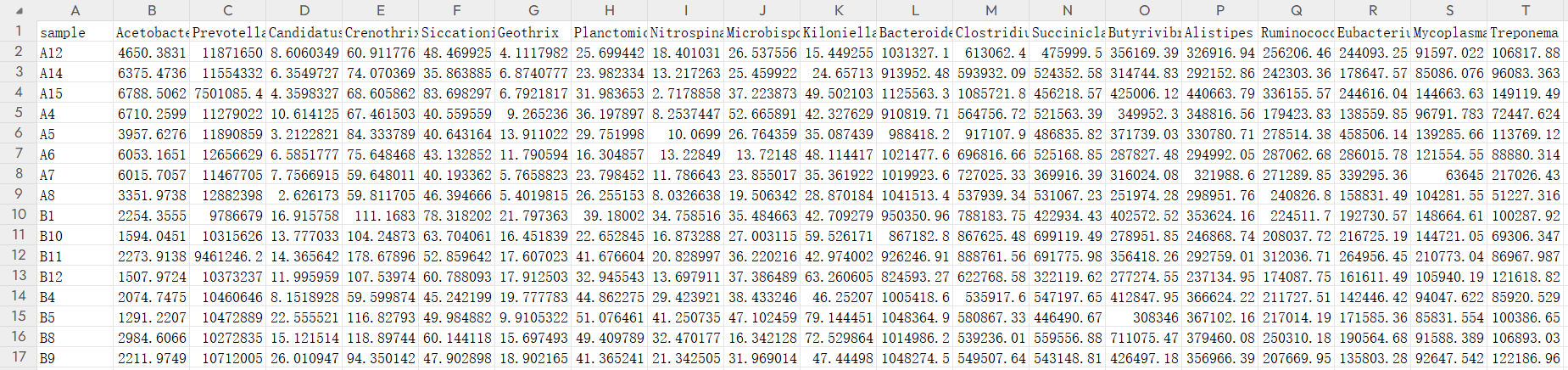
- 不同样本的菌群丰度
genus.csv
R包linkET可视化
- 装包
install.pakages("linkET") library(linkET)- 1
- 2
如果报错R版本有问题装不上(我的4.3.1版本R出现了这个报错)请尝试:
install.packages("devtools") devtools::install_github("Hy4m/linkET", force = TRUE) packageVersion("linkET")- 1
- 2
- 3
- 读取数据
library(ggplot2) rumen <- read.csv("rumen.csv",sep=",",row.name=1,stringsAsFactors = FALSE,check.names = FALSE) genus <- read.csv("genus.csv",sep=",",row.name=1,stringsAsFactors = FALSE,check.names = FALSE) #如果报错row.names重复错误请检查数据格式是否为csv- 1
- 2
- 3
- 4
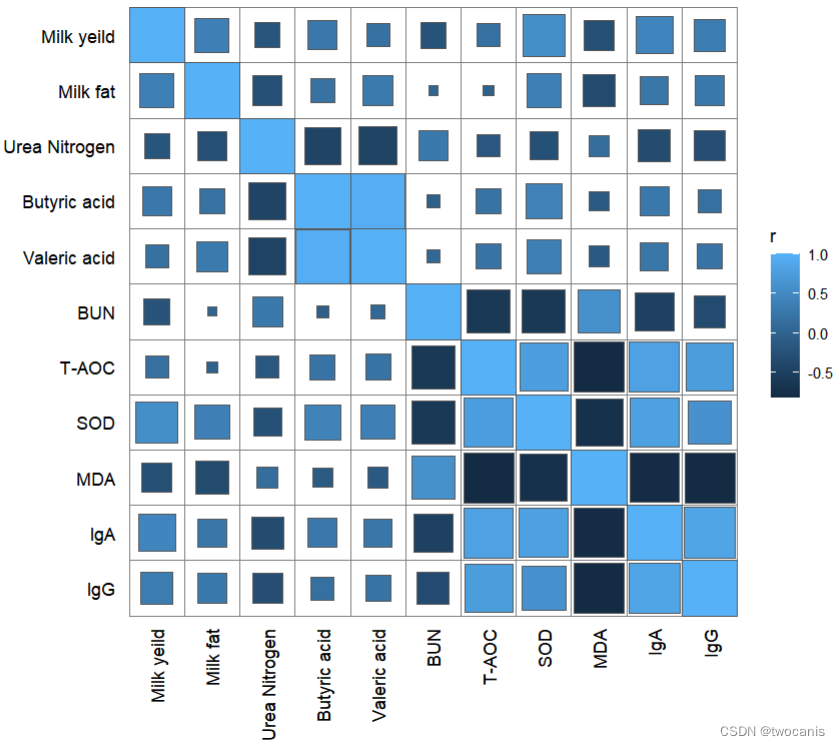
rumen.csv组内相关系数
matrix_data(list(rumen = rumen)) %>% as_md_tbl() correlate(rumen) %>% as_matrix_data() correlate(rumen) %>% as_md_tbl() correlate(rumen) %>% as_md_tbl() %>% qcorrplot() + geom_square() #如果对“%>%”功能报错,装具有此功能的包即可,比如dplyr library(vegan) correlate(rumen, genus, method = "spearman") %>% qcorrplot() + geom_square() + geom_mark(sep = '\n',size = 3, sig_level = c(0.05, 0.01, 0.001), sig_thres = 0.05, color = 'white') + #添加显著性和相关性值 scale_fill_gradientn(colours = RColorBrewer::brewer.pal(11, "RdBu"))- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
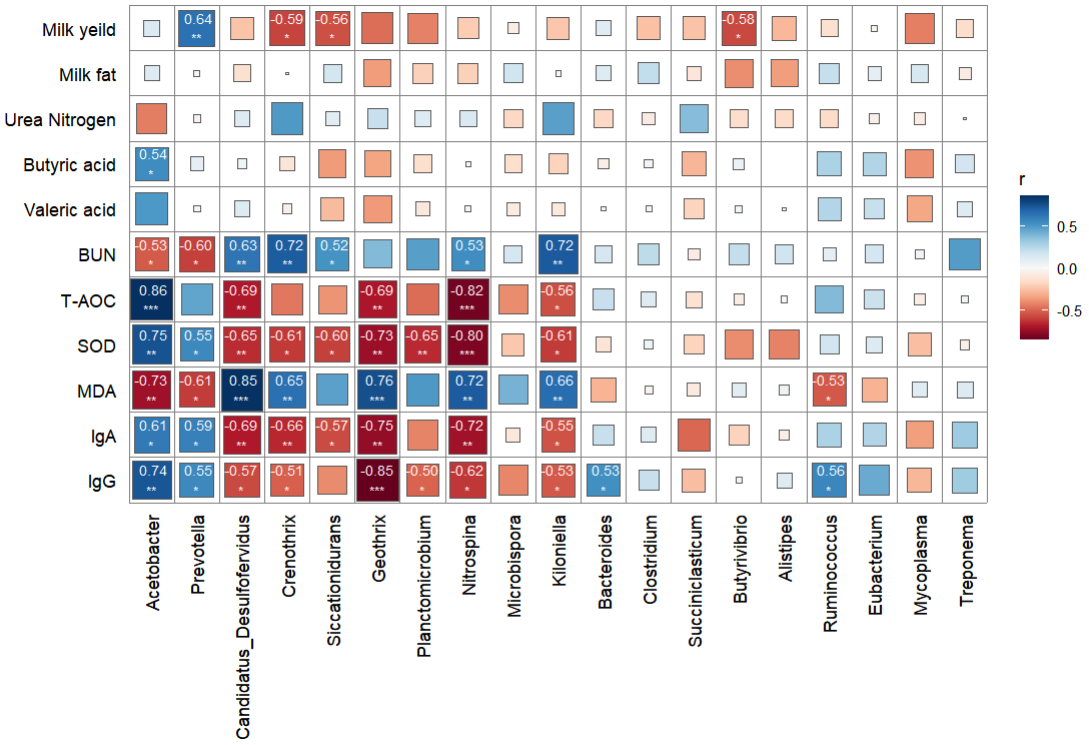
- 两个表格进行关联生成相关性矩阵图,带显著性标记
library(vegan) correlate(rumen, genus, method = "spearman") %>% qcorrplot() + geom_square() + geom_mark(sep = '\n',size = 3, sig_level = c(0.05, 0.01, 0.001), sig_thres = 0.05, color = 'white') + #添加显著性和相关性值 scale_fill_gradientn(colours = RColorBrewer::brewer.pal(11, "RdBu"))- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 加工可视化
library(dplyr) mantel <- mantel_test(rumen, genus, spec_select = list(Milk_yeild=1,Milk_fat=2,Urea_Nitrogen=3,Butyric_acid=4,Valeric_acid=5,BUN=6, T_AOC=7,SOD=8,MDA=9,IgA=10,IgG=11))%>% mutate(rd = cut(r, breaks = c(-Inf, 0.5, Inf), labels = c("< 0.5", ">= 0.5")), pd = cut(p, breaks = c(-Inf, 0.01, 0.05, Inf), labels = c("< 0.01", "0.01 - 0.05", ">= 0.05"))) qcorrplot(correlate(genus), type = "lower", diag = FALSE) + geom_square() +geom_mark(sep = '\n',size = 1.8, sig_level = c(0.05, 0.01, 0.001), sig_thres = 0.05,color="white") + geom_couple(aes(colour = pd, size = rd), data = mantel, curvature = nice_curvature()) + scale_fill_gradientn(colours = RColorBrewer::brewer.pal(11, "RdBu")) + scale_size_manual(values = c(0.5, 1, 2)) + scale_colour_manual(values = color_pal(3)) + guides(size = guide_legend(title = "Mantel's r", override.aes = list(color = "black"), order = 2), colour = guide_legend(title = "Mantel's p", override.aes = list(size = 3), order = 1), fill = guide_colorbar(title = "Pearson's r", order = 3))- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
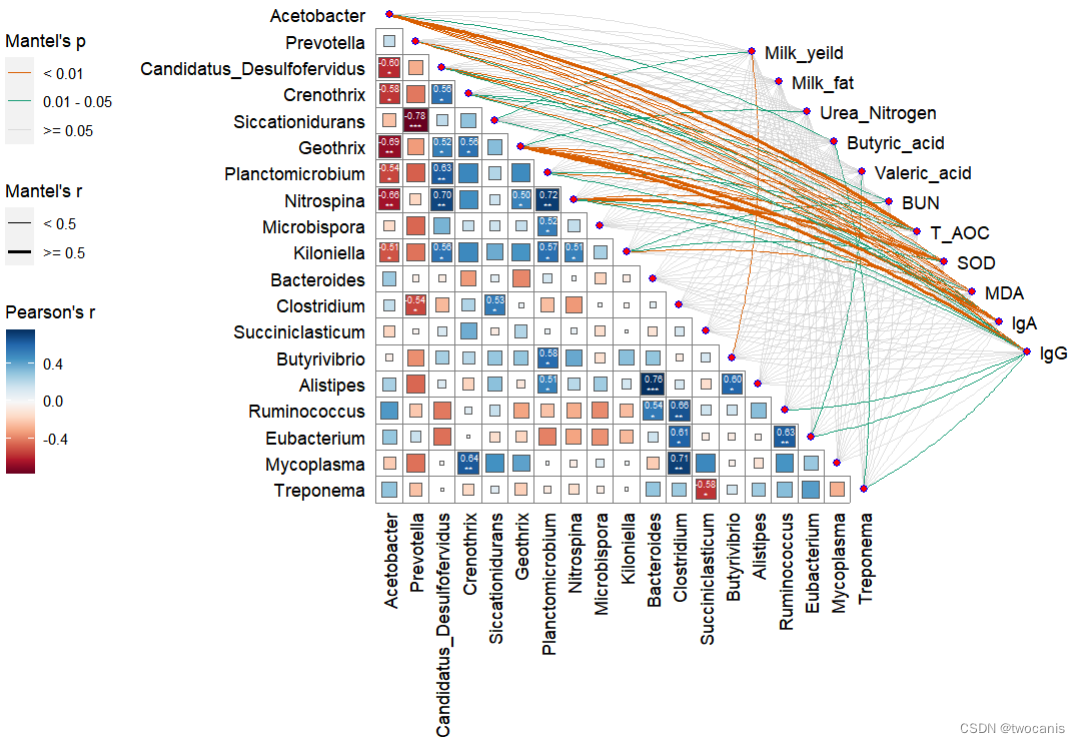
- 不显著的灰色连接线部分也可以去掉让画面更干净。其余细节去AI加工即可。
- 需要关联的表观数据
-
相关阅读:
Java 线程安全 与 锁
Golang 串口通信
详细介绍如何使用Ipopt非线性求解器求解带约束的最优化问题
安装typescript环境并开启VSCode自动监视编译ts文件为js文件
基于MobileNet的轻量级卷积神经网络实现玉米螟虫不同阶段识别分析
数据中台实战(11)-数据中台的数据安全解决方案
【目标检测】50、YOLOX | 回归 anchor-free 的 YOLO 依然能打!
鸿蒙Harmony应用开发—ArkTS声明式开发(容器组件:Refresh)
关于“兆易创新杯”中国研究生电子设计竞赛的一点个人小经验
微信小程序连接数据库与WXS的使用
- 原文地址:https://blog.csdn.net/twocanis/article/details/133313555
