-
用于人类复杂疾病成药性评估的R包:DREAM包
已获批药物开发新适应症时,需要对所研究的疾病进行彻底的成药性评估。先前成药性主要通过三种不同的方法进行评估,第一种方法基于公共存数据,关注不同治疗条件下药物的作用机制(MOA);第二种是基于网络,关注疾病背后的受损分子相互作用;第三种基于结构,关注药物及其分子靶标的内在结构特性。但是,这些方法均存在一些局限性。因此,引入R包DREAM,将机械和化学中心方法集成到统一的计算工作流程中。DREAM致力于对感兴趣的病理状况进行成药性评估,利用强大的药物再利用和药物组合预测功能。
功能与模块介绍
DREAM包提供多种功能,(i)鉴定疾病相关基因和推断共表达网络,(ii)识别候选药物靶点,(iii)药物MOA的推断和评估,(iv)评估药物 - 药物相似性,以及(v)确定候选药物以重新利用和联合治疗疾病等。
DREAM包由三个模块组成。模块1用于输入基因表达数据并检索与疾病相关的基因。Wilcoxon和F-test用来评估疾病与健康之间基因表达差异并测试基因表达稳定性。此外可以构建或使用预先构建的基因共表达网络来执行在下一个模块中实现的操作。模块2用于评估所研究疾病的成药性以及疾病网络上药物的拓扑特性(例如疾病网络上被研究药物的作用区域及其药物靶标之间的距离),来研究药物间的协同作用。模块3用于药物选择,识别适合再利用和组合的药物。选择是通过多目标遗传算法(GA)进行。
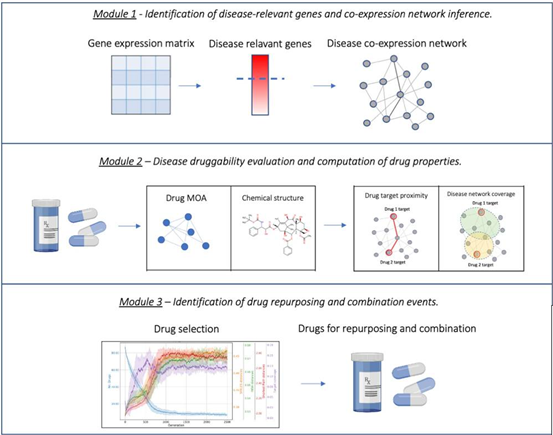
实例分析
在这里,通过对特应性皮炎的案例研究来展示DREAM的功能。
第一步,确定在特应性病变中相对于未受影响的皮肤表达改变的基因,利用这些基因构建代表特应性病变的共表达网络。
第二步,通过生成从LINCS21存储库收集的扰动谱的共表达网络来表征在FIBRNPC细胞系中测试的1000种药物的MOA。
第三步,通过PubChem(https://pubchem.ncbi.nlm.nih.gov/)收集其结构表示来推断所考虑药物的化学结构/子结构。
最后,通过DREAM的功能整合这三层信息,对特应性皮炎病变的成药性进行建模。
药物组合预测鉴定了20种药物组成的2种组合,17种药物组成的3种组合和4种药物组成的4种组合治疗特应性皮炎。

下期我们将继续分享DREAM包的R实现。
References:
Federico A, Fratello M, Pavel A, Möbus L, Del Giudice G, Serra A, Greco D. DREAM: an R package for druggability evaluation of human complex diseases. Bioinformatics. 2023 Jul 1;39(7):btad442. doi: 10.1093/bioinformatics/btad442IF: 5.8 Q1 . PMID: 37471593IF: 5.8 Q1 ; PMCID: PMC10374489IF: 5.8 Q1 .
搜索公众号“单细胞学会”,获取更多信息。
-
相关阅读:
空数组循环会不会执行
STM8应用笔记5.8位定时器应用之一
springboot留守儿童爱心网站springboot35
sed续与awk(粗糙版)
【云开发】给我 10 分钟,带你上手一个 AWS serverless web server
【深度学习实验】前馈神经网络(六):自动求导
《八月算法》——位运算
Log4j的原理及应用详解(五)
IBM LinuxONE Community Cloud 免费试用申请教程
【HarmonyOS】【JAVA UI】自定义通知的实现
- 原文地址:https://blog.csdn.net/qq_42458954/article/details/132883433