-
医学图像处理——DeepDrr工具CT生成DRR
一、DeepDrr工具介绍
它可以将CT数据模拟X射线投影生成DRR数据。
DeepDRR将3D和2D的材料分解和散射估计的机器学习模型分别与投影、衰减和噪声注入的分析模型相结合,以实现所需的性能,其实现的流程图如下:
 1. DeepDRR工具Github链接
1. DeepDRR工具Github链接
2. DeepDRR工具文档说明二、DeepDRR工具使用
由于实验室的1080ti不支持cuda11,为了节约调试时间就租用了RTX 2080 Ti * 1卡,租用平台AutoDL,以下是平台的配置详情。
按照1. DeepDRR工具Github链接配置环境就行,在这个平台直接跑README.md文件内容。# 注意把该命令 conda env create -f environment.yaml # 换成 conda env create -f environment.yml # 下面是文档生成命令不用管 sphinx-apidoc -f -o docs/source deepdrr- 1
- 2
- 3
- 4
- 5
- 6
后续就是提示缺啥包就按啥包。
三、测试DeepDRR工具
3.1 测试数据集
它提供了一个最简单的使用例子,从NifTi.nii.gz(nii文件是volume file格式,常见的一种医学图像,可以由MRI软件生成,它可以看成2维,也可以看成三维)文件中加载一个CT数据,并模拟一个X射线投射生成一张DRR。但是我们没有上述CT数据。
3.1.1 阿里云数据
1. 阿里云数据,找了几个数据集,都是PNG格式的,后续使用.png转.nii.gz,其效果不好。
// 多幅png图片转.nii.gz文件 import numpy as np import nibabel as nib from glob import glob from torch.utils.data import Dataset from torchvision import transforms from PIL import Image class DataSet(Dataset): # 方便读数据 def __init__(self, label_path): self.label_path = label_path self.transform = transforms.Compose([transforms.ToTensor()],) def __len__(self): return len(self.label_path) def __getitem__(self, idx): label = self.label_path[idx] label = Image.open(label) label = self.transform(label) return label label_path = r'C:\Users\tanhui\Downloads\CP\679\3041\*' label_path = glob(label_path) dataset = DataSet(label_path) label = dataset[0] z, x, y = label.shape z = len(dataset) # 通道数 allImage = np.zeros([x, y, z], dtype='uint8') # print(x, y, z) for i in range(z): label = dataset[i] singleImage = label.numpy() allImage[:, :, i] = singleImage # print(allImage.shape) new_image = nib.Nifti1Image(allImage, np.eye(4)) nib.save(new_image, 'data2.nii.gz')- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
3.1.2 LIDC-IDRI肺结节dicom数据集
2. LIDC-IDRI肺结节dicom数据集,这个数据集可能要科学上网才下得快且要安装它站里面的一个下载工具(数据集太大,工具帮助我们选择性下载),附上一个简单的下载教程。
// 多张dicm格式文件转.nii.gz文件 #coding=utf-8 import SimpleITK as sitk def dcm2nii(dcms_path, nii_path): # 1.构建dicom序列文件阅读器,并执行(即将dicom序列文件“打包整合”) reader = sitk.ImageSeriesReader() dicom_names = reader.GetGDCMSeriesFileNames(dcms_path) reader.SetFileNames(dicom_names) image2 = reader.Execute() # 2.将整合后的数据转为array,并获取dicom文件基本信息 image_array = sitk.GetArrayFromImage(image2) # z, y, x origin = image2.GetOrigin() # x, y, z spacing = image2.GetSpacing() # x, y, z direction = image2.GetDirection() # x, y, z # 3.将array转为img,并保存为.nii.gz image3 = sitk.GetImageFromArray(image_array) image3.SetSpacing(spacing) image3.SetDirection(direction) image3.SetOrigin(origin) sitk.WriteImage(image3, nii_path) if __name__ == '__main__': dcms_path = r'D:\data\manifest-1668773888310\LIDC-IDRI\LIDC-IDRI-0002\01-01-2000-NA-NA-98329\3000522.000000-NA-04919' # dicom序列文件所在路径 nii_path = r'.\test.nii.gz' # 所需.nii.gz文件保存路径 dcm2nii(dcms_path, nii_path)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
3.1.3 LIDC-IDRI肺结节dicom数据集
使用MRIcroGL软件查看合成的CT数据。

3.2 测试代码
使用下面测试代码,将上面合成的test.nii.gz CT数据生产一张DR。
from deepdrr import geo, Volume, MobileCArm from deepdrr.projector import Projector # separate import for CUDA init carm = MobileCArm() ct = Volume.from_nifti('/path/to/ct_image.nii.gz') # Initialize the Projector object (allocates GPU memory) with Projector(ct, carm=carm) as projector: # Orient and position the patient model in world space. ct.orient_patient(head_first=True, supine=True) ct.place_center(carm.isocenter_in_world) # Move the C-arm to the desired pose. carm.move_to(alpha=30, beta=10, degrees=True) # Run projection image = projector()- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
效果如下:
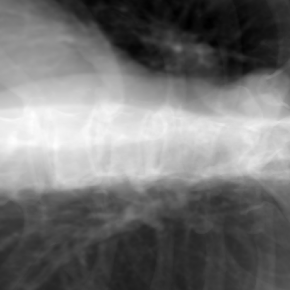
四、总结
简单的将DeepDRR工具运行了一下,初测了它的很小一部分功能,后续期待能基于该工具做出更好的应用。
-
相关阅读:
自上而下 or 自下而上?企业部署RPA的2种策略
linux下使用qt+mpv调用GPU硬件解码
旁路openwrt启用ipv6
how2heap2.31学习(2)
网络-fetch
Linux——程序地址空间
《c++程序设计》谭浩强课后习题答案 第一张
测试用例的优化与整理:确保软件质量的关键步骤
【Leetcode】200. 岛屿数量
百度松果 美丽的三角形(求杨辉三角n行m列的数奇偶性)
- 原文地址:https://blog.csdn.net/weixin_44911552/article/details/127942035