-
机器学习实战—降维
一.简介
在实际的机器学习案例中,我们的实例可能会涉及成千上万甚至数百万个特征,这样会导致我们模型的整个的训练过程会极其的缓慢,这个问题通常称为维度的诅咒。而我们可以通过数据降维去减少我们应用到实际训练的特征,此时担忧来了,特征的减少会不会影响模型的精确度。实际是,数据降维确实会丢失一些信息,它虽然能够加速训练,但是会轻微降低系统性能,同时它也会让流水线更加的复杂,维护难度大大上升;不过在有些情况下,降低数据的维度可能会过滤掉一些不必要的噪声和细节,从而导致性能更加的好(因为不同的特征对模型的影响程度是不同的,甚至有些特征完全对我们的训练目标不会造成影响)。另一方面,在我们的世界中我们可能能想象的最高维度就是三维空间,就像我们在《星际穿越》的结尾看到主角库珀进入的五维空间也只是人类的一个想象,所以我们太习惯三维空间,所以当我们试图去想象一个高维空间时,是很难想象的。所以降低维度可以帮助我们更好的去理解空间中数据在空间中的分布。
二.降低维度的主要方法
2.1 简介
在学习机器学习中真正的降维算法之前,我们需要了解两种减少维度的方法:投影和流形学习
2.2 投影
投影的原理还是比较好理解的,打个比方如果我们用一束光射向一个篮球,最后在墙面上程序的影子就是一个圆形,这样我们就实现了将三维的球降维成了二维的圆。

如上图我们将位于三维空间(实例有三个特征x,y,z)的实例映射到了二维,此时在二维平面上实例点通过降低维度抛弃了特征y,只有两个特征x,y。这样通过投影就实现了降维。
投影的缺点:
通过上面我们知道了投影的原理,但是我们设想一下这种情况,如果数据在三维空间中的分布是扭曲的,这时候投影就不能很好的处理降维工作了。设想数据的分布类似瑞士卷,这时候直接进行投影的话会让瑞士卷的不同层数据挤压到一起,此时我们需要在二维平面上展开瑞士卷,这时候就涉及到更复杂的技术。

2.3 流形学习
在理解流行学习之前,我们需要理解一下什么叫做流形,流形是局部具有欧几里得空间性质的空间,在数学中用于描述几何形体。通俗理解就是如果我们将上面的瑞士卷展开,这时瑞士卷就变成二维了,但我们可以通过让瑞士卷卷起来去在三维空间中去表示它,而卷起来的瑞士卷就是我们所说的流形。所以我们称上面的瑞士卷就是一个2D流形,2D流形就是可以在更高维度的空间中弯曲和扭曲的2D形状,更一般而言d维流形是n维空间(其中d
现在我们给出流形学习的定义,例如我们可以假设某些高维数据,例如图像,本身是由某些低维分布产生的。那么,我们就可以通过流形学习的手段,将数据从高维降低到低维,从而更好地把握数据的本质。例如下图我们取 x 1 x_1 x1=5作为我们的决策边界,然后展开流行就得到了图2的2D效果(可以看出有很好的分类效果),这就是通过流形学习降低维度的原理。

三.PCA降维
3.1 简介
PCA(主成分分析)就是我们上面介绍的投影的一种,是迄今为止最流行的降维算法,它会识别最靠近数据的超平面,然后将数据投影到其上。
3.2 保留差异性
在我们使用PCA时,我们要去选择最合适的超平面去让数据投影到其上,而选取超平面的原则就是尽量让数据投影到超平面上时能保留最大的差异性。下面举一个二维降到一维的例子来讲解什么是差异性:

上图假设PCA取了两个超平面l1和l2,可以发现l1上最后投影的数据差异性是很大的,而l2很多数据投影后被压缩到了一起,差异性过小,所以PCA会选择l1作为其超平面。那么在实际应用中我们该如何确定这个超平面呢?思路就是比较原始数据集与其轴上的投影之间的均方距离,使这个均方距离最小的轴是最合理的选择,这就是PCA背后简单的思想。
3.3 主要成分
通过上面的分析我们总结一下PCA的原理。PCA的主要思想是将n维特征映射到d维上(如3.2中的n是2,的维是1),这d维是全新的正交特征也被称为主成分,是在原有n维特征的基础上重新构造出来的d维特征。PCA的构造主成分的流程如下:
- 第一个主成分:选择原始数据中差异性最大的超平面(如上面使用均方距离来判断)
- 第二个主成分:选取与第一个主成分正交的超平面,然后选择选择使原始数据差异性最大的超平面(如3.2中只有l2与l1是正交的)
- 第三个主成分:选取与第一个与第二主成分正交的超平面,如何同选取使得原始数据差异性最大的超平面作为第三个主成分
- 以此类推,迭代上面过程直到找出n个主成分(虽然我们找到了与原始数据维度相同数量的主成分数量,但我们会发现大部分方差都包含在前面d个坐标轴中,后面的坐标轴所含的方差几乎为0。于是,我们可以忽略余下的坐标轴,只保留前面d个含有绝大部分方差的坐标轴。事实上,这相当于只保留包含绝大部分方差的维度特征,而忽略包含方差几乎为0的特征维度,这样就实现对数据特征的降维处理)
那么训练集的主成分是如何找的,PCA使用的是一种称为奇异值分解(SVD矩阵论知识)的标准矩阵分解技术,来获取主成分矩阵的:
V = ( C 1 , C 2 , C 3 , . . . . . , C n ) V=V=⎝ ⎛C1,C2,C3,.....,Cn⎠ ⎞⎛⎝⎜C1,C2,C3,.....,Cn⎞⎠⎟ 在Python代码中可以使用Numpy中的svd()函数来获得所有的主主成分:
from sklearn.datasets import load_iris import numpy as np iris=load_iris() x=iris.data xcenter=x-x.mean(axis=0)#求每个特征的均方差 U,S,Vt=np.linalg.svd(xcenter) Vt.T- 1
- 2
- 3
- 4
- 5
- 6
- 7
结果分析:
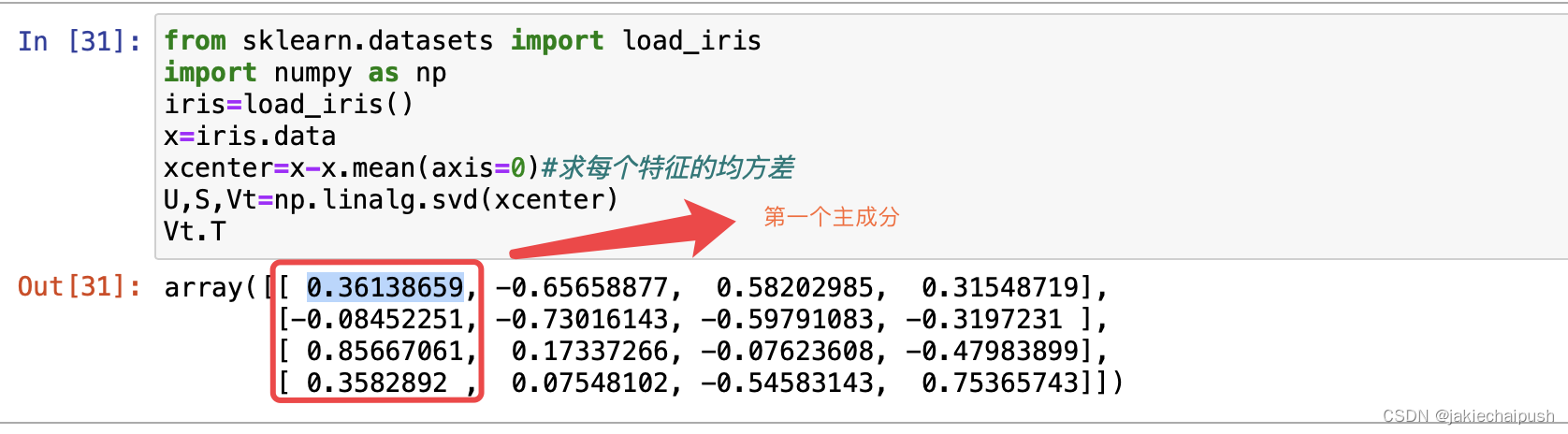
鸢尾花数据有四个特征,所以其数据都分布在四维空间中,所以这里产生来4个主成分(1列为一个坐标)
3.4 向下投影到d维度
上面我们已经知道列主成分的求法,那么我们就可以将数据集映射到前d个主成分定义的超平面上了,要将训练集投影到超平面上并得到维度为d的简化数据集,计算方法上训练集矩阵X于矩阵 W d W_d Wd的矩阵相乘,矩阵 W d W_d Wd定义为包含V的前d项矩阵,计算公式如下:
X d − p r o j = X W d X_{d-proj}=XW_d Xd−proj=XWd
python代码如下:
W2=Vt.T[:,:2] #这里的2就是选取V的前两列,即将4维的鸢尾花数据映射到2维上 X2D=xcenter.dot(W2)- 1
- 2

3.5 在Scikit-learn使用PCA的接口
from sklearn.decomposition import PCA pca=PCA(n_components=2)#降为2维 X2D2=pca.fit_transform(x)- 1
- 2
- 3

3.6 可解释方差比
可解释方差比表示沿每个成分的数据集方差的比率,它们的和<=1, 可解释方差之和比越接近1,特征数越多,越接近原数据。在scikit-learn可以通过explained_variance_ratio_变量来获取。
pca.explained_variance_ratio_- 1

可解释方差比可以帮助我们降维时选择合适的维度,只要将n_component设置为我们想要的可解释方差比即可,scikit-learn会帮助我们自动去选择合适的维度。
pca=PCA(n_components=0.95) X_reduce=pca.fit_transform(x) X_reduce- 1
- 2
- 3

总的来说,pca会将高维数据映射到低维,不仅让我们可以直观的观察数据加快模型训练过程,还可以一定程度上对大数据集进行了一定的压缩(因为有特征数量变少了)
3.7 随机PCA
随机PCA是另一种PCA算法,它可以比传统使用SVD方法的PCA更快的找到主成分(这就意味着当n和d差距比较大时,随机PCA可以更快的帮我们找到d个主成分)。在scikit-learn我们只需要将svd_solver设置为"randomized"即可:
rnd_pca=PCA(n_components=2,svd_solver="randomized") x_reduce=rnd_pca.fit_transform(x)- 1
- 2
其实在实际使用中,我们可以将svd_solver设置为auto,这样Scikit-learn会自动帮助我们在不同的情况下选择合适的pca算法。
3.8 增量PCA(IPCA)
我们都知道电脑的内存是有限的,就拿我的电脑内存是16个G来说,如果我要用我电脑去在32G的数据上跑PCA算法,如果我使用前面介绍的PCA算法,我的内存就会爆炸(因为前面的PCA算法都需要一次性将整个训练集放入内存,这远不是我电脑内存能够承受的),所以就出现了增量PCA算法,它会将训练集划分为多个小批量,并一次将一个小批量送入(IPCA)算法,因为内存和存储之间交换数据是很花费时间的,所以IPCA效率不如前面介绍的PCA算法。使用方法如下:
from sklearn.decomposition import IncrementalPCA n_batcher=6 #将鸢尾花数据分为6个批量 inc_pca=IncrementalPCA(n_components=2) for X_batch in np.array_split(x,6): inc_pca.partial_fit(X_batch) X_reduce=inc_pca.transform(x)- 1
- 2
- 3
- 4
- 5
- 6
四.内核PCA
4.1 简介
我在支持向量机这篇博客中介绍了一个多项式核技术,它是一种数据技术,可以将实例隐式的映射到一个高维的空间,从而可以使用支持向量机进行非线性分类和回归。在前面我们都是选择线性的超平面来映射数据集。然而有时候,数据不是线性的,不能直接进行PCA降维。这里就需要用到和支持向量机一样的核函数的思想,先把数据集从n维映射到线性可分的高维N>n,然后再从N维降维到一个低维度n’, 这里的维度之间满足n’
4.2 scikiit-learn接口使用
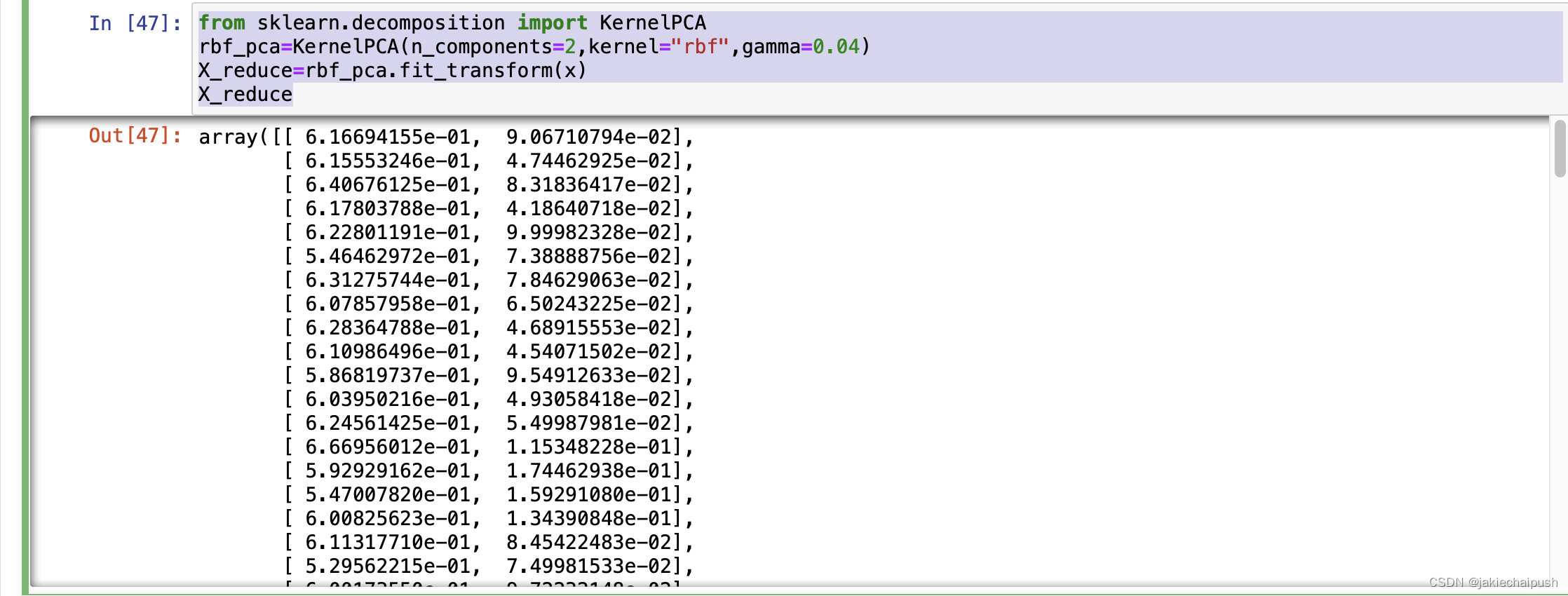
下面代码使用Scikit-learn的KernelPCA类以及用RBF内核执行KPCA
from sklearn.decomposition import KernelPCA rbf_pca=KernelPCA(n_components=2,kernel="rbf",gamma=0.04) X_reduce=rbf_pca.fit_transform(x) X_reduce- 1
- 2
- 3
- 4
五.LLE降维
5.1 简介
局部线性嵌入(LLE)是另一种强大的非线性降维(KPCA也是非线性降维技术)技术,它是一种流形学习技术,LLE在流形建模方面是做的非常好的,不像PCA那样依赖于投影。LLE的工作原理是首先测量每个训练实例如何与其最近的邻居线性关系,然后寻找最好地保留这些局部关系的训练集低维表示。回忆一下流形学习的原理:我们可以假设某些高维数据,例如图像,本身是由某些低维分布产生的。那么,我们就可以通过流形学习的手段,将数据从高维降低到低维,从而更好地把握数据的本质,而在LLE中寻找的线性关系就是所谓低维在高维的局部体现,而我们可以使用这种关系将高维映射到低维。
5.2 Scikit-learn中使用
scikit-learn中使用LocallyLinearEmbedding类来进行降维(这种降维就是我们2.3中介绍的展开瑞士卷的过程)
from sklearn.manifold import LocallyLinearEmbedding lle=LocallyLinearEmbedding(n_components=2,n_neighbors=10)#n_neighbors就是我们要求线性关系的邻居实例的数量 X_reduce=lle.fit_transform(x) X_reduce- 1
- 2
- 3
- 4
5.3 LLE的工作原理
下面将一步步介绍LEE的工作原理
- 首先LLE对每个训练实例 x i x_i xi,算法会识别出最近的邻居,这个和knn算法找邻居的原理是一样的,所以可以使用knn算法来找到邻居
- ,LLE会尝试将 x i x_i xi重构为这些邻居的线性函数,即找到权重系数 w i , j ,使得 x i w_{i,j},使得x_i wi,j,使得xi与 ∑ j = 1 m w i , j x i \sum_{j=1}^m w_{i,j}x_i ∑j=1mwi,jxi之间的距离尽可能小(线性回归拟合问题),W是权重矩阵
W ^ = a r g m i n ∑ i = 1 m ( x i − ∑ j = 1 m w i , j x j ) 2 \hat W=argmin\sum_{i=1}^m(x_i-\sum_{j=1}^m w_{i,j}x_j)^2 W^=argmini=1∑m(xi−j=1∑mwi,jxj)2
满足 { w i , j , x j 不属于 x i 的邻居 ∑ j = 1 m w i , j = 1 , 其中 i = 1 , 2 , 3 , . . . . , m 满足满足{wi,j,∑j=1mwi,j=1,xj不属于xi的邻居其中i=1,2,3,....,m{wi,j,∑mj=1wi,j=1,xj不属于xi的邻居其中i=1,2,3,....,m - 什么求出权重矩阵后我们已经在高维空间找到了实例的局部线性关系,现在就需要将训练实例映射到低维空间,同时尽可能保留这些局部关系到低维。如何保证这个约束条件,我们可以让d维度空间中
x
i
x_i
xi的图像与关系
∑
j
=
1
m
w
i
,
j
x
i
\sum_{j=1}^m w_{i,j}x_i
∑j=1mwi,jxi的距离同样保持最小,注意这里的
∑
j
=
1
m
w
i
,
j
x
i
\sum_{j=1}^m w_{i,j}x_i
∑j=1mwi,jxi是从高维映射到低维的,因为局部线性关系在降维中是保持不变的,所以这里在低维度空间中我们同样希望降维后的实例依然与
∑
j
=
1
m
w
i
,
j
x
i
\sum_{j=1}^m w_{i,j}x_i
∑j=1mwi,jxi保持最小的距离。
Z ^ = a r g m i n ∑ i = 1 m ( z i − ∑ j = 1 m w i , j z j ) 2 \hat Z=argmin\sum_{i=1}^m(z_i-\sum_{j=1}^m w_{i,j}z_j)^2 Z^=argmini=1∑m(zi−j=1∑mwi,jzj)2
-
相关阅读:
掌握苏宁API,一键获取商品详情,解锁无尽商业可能
第20节-PhotoShop基础课程-橡皮檫工具
QT pyside2 线程嵌套子线程 实现开始运行和停止运行
【C语言速查表】数据类型、转义与格式控制、ascii码
c++ 并发与多线程(12)线程安全的单例模式-1
这些软件测试面试题如果都会,月薪最少20k+【内附面试宝典】
Taurus.MVC WebAPI 入门开发教程6:全局控制器DefaultController与全局事件。
C++运算符重载
mybatis判断是否存在参数
过拟合问题(机器学习)
- 原文地址:https://blog.csdn.net/qq_43456605/article/details/127748692