-
R包WGCNA---转录组WGCNA共表达网络构建(基本概念)
R包WGCNA---转录组WGCNA共表达网络构建(基本概念)
1. WGCNA简介
基于基因表达量相关性计算的网络在生物信息学中广泛应用,加权的基因共表达网络用来描述多个样本中基因表达的相关模式。此时,基于
权重的相关性网络分析(WGCNA)应运而生,WGCNA可以挖掘高度相关基因的表达模块(module)或基因簇(cluster),并使用特征基因(eigengene)或关键基因(hub gene)来简要代表这类基因簇,相关的模块之间和模块与外部表型值可以计算成员测量值(module membership measures),作为后续分析的选择指标。这种相关性网络的方法可以用于基因扫描鉴定生物标记和治疗靶点,已经成为系统生物学中重要的分析方法。有学者开发了相关的
R包WGCNA,该R包文章于2008年发表在BMC bioinformatics上面,题目为,目前网页显示引用量为12800+,是多基因模块化聚类,、共表达网络分析的重要工具。此外,了解该方法的实施细节,WGCNA不仅可以用于基因表达型数据,也可以作为一种数据挖掘工具,拓宽研究应用。
本文主要是依据WGCNA官网和优秀的博主文章,介绍WGCNA在基因表达数据中的原理及应用介绍。WGCNA: an R package for weighted correlation network analysis- 1
 目前在google scholar中引用次数为12820次。
目前在google scholar中引用次数为12820次。该文章共有两个作者,
第一作者在UCLA做数据分析(https://peterlangfelder.com/about-the-author/),下图所示;
通讯作者是人类遗传与统计系的教授(https://www.biostat.ucla.edu/people/horvath):

2. WGCNA分析原理
(1)R包WGCNA的主要功能
1. network construction; 2. module detection; 3. module and gene selection; 4. calculations of topological properties; 5. data simulation; 6. visualization; 7. interfacing with external software packages.- 1
- 2
- 3
- 4
- 5
- 6
- 7
(2)WGCNA的基本概念和工作流程
主要参考下面的官方文档和生信宝典的教程文章:
https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/Tutorials/index.html https://cloud.tencent.com/developer/article/1936465- 1
- 2
Step1:基本概念的理解(英文表格来自WGCNA英文文章)
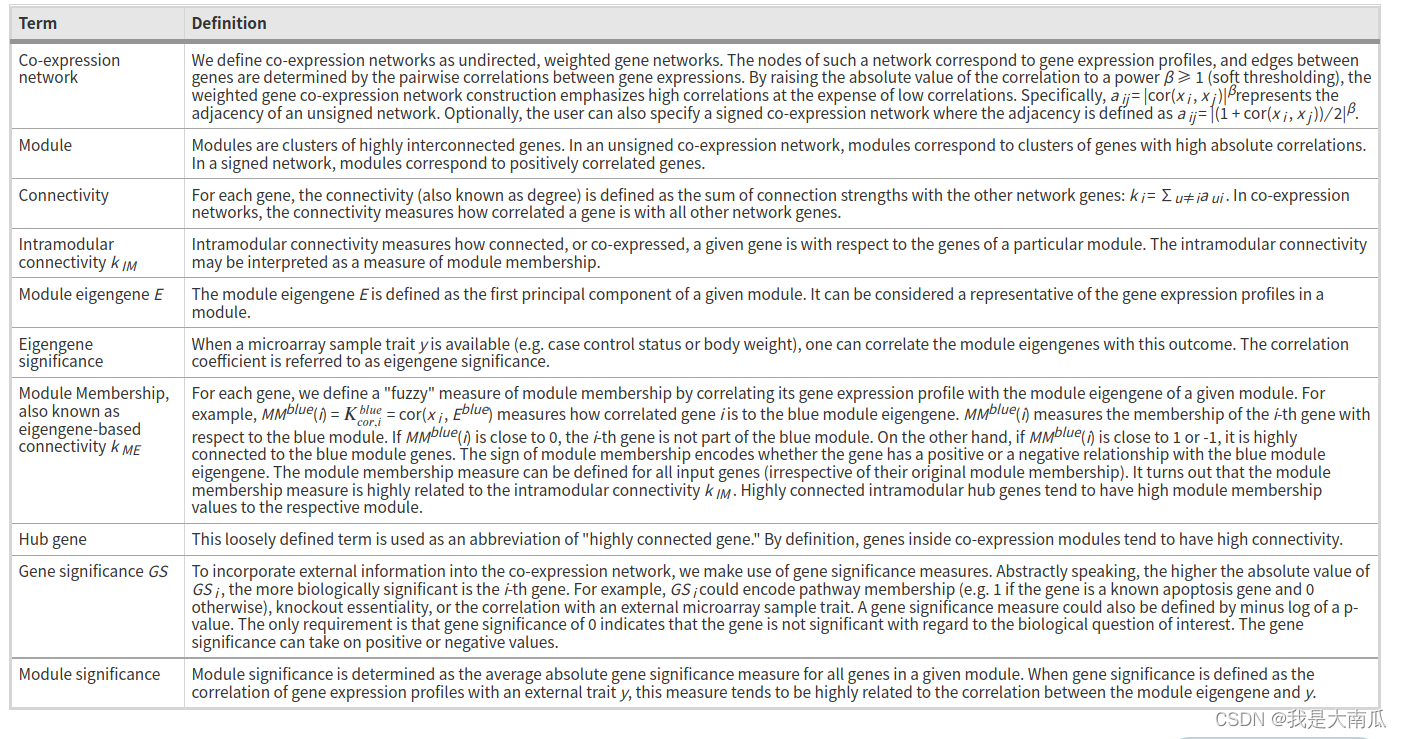
中文翻译参考生信宝典的教程文档,非常详实,下面为其内容截图:

Step2:工作流程如下
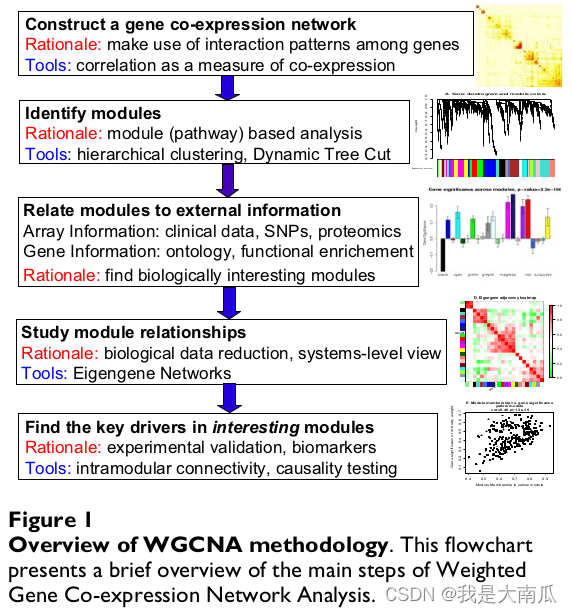
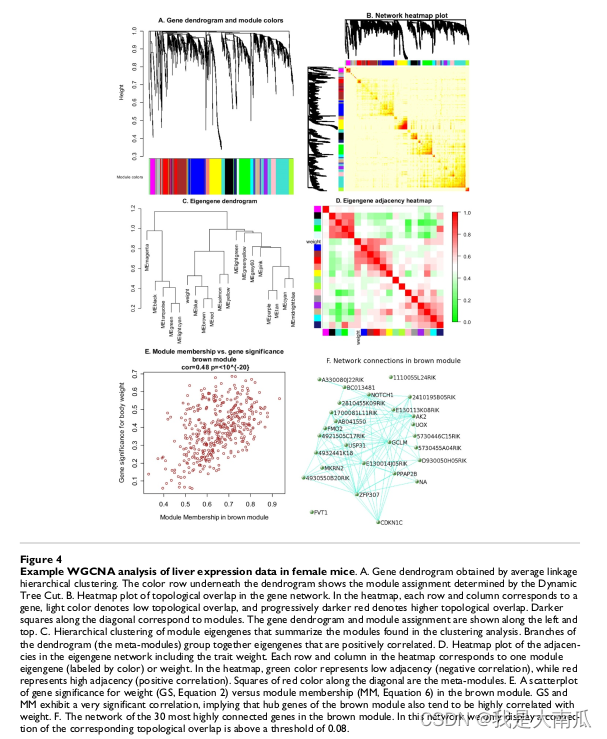
主要的模块构建,模块选择和网络绘制(具体实操代码参看官方文档或其他博客):
(3)WGCNA分析的常见问题及注意事项
https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/faq.html- 1
WGCNA中的FAQ问题(主要内容截图):



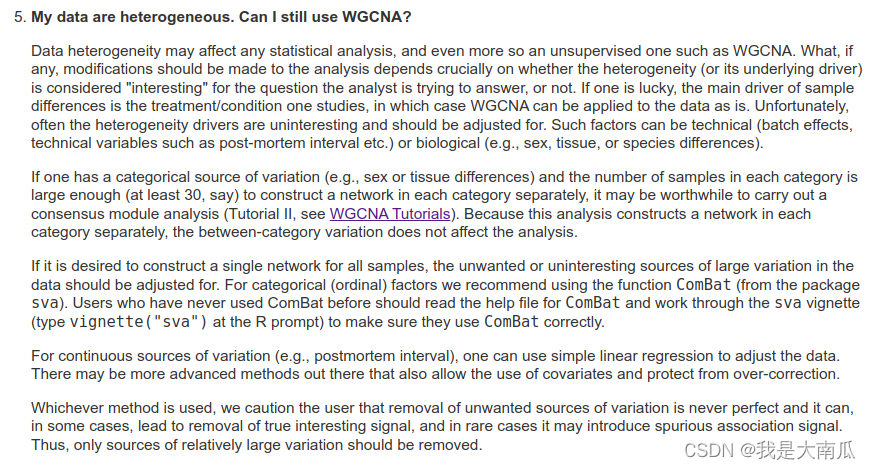
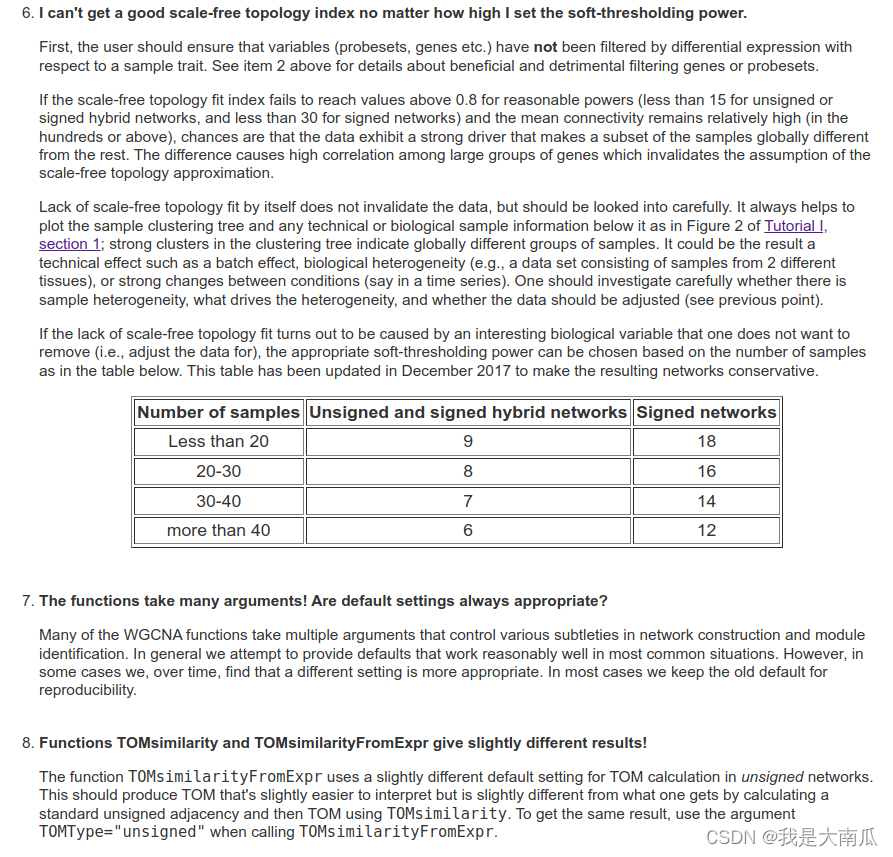
文章中也提到WGCNA使用的一些局限:
(1)WGCNA已经假定输入数据已经是处理妥当的,已经是标准化的数据(microarray data)
使用一些Bioconductor的R包完成标准化过程,要结合生物学意义进行标准化,即使数学上没问题也不行。
(2)WGCNA工具是存在偏差或失效的
当存在技术问题,组织污染,实验设计不当等原因都会导致结果分析偏差。
(3)WGCNA中的多种共表达模块检测方法并未进行比较
WGCNA中多种方法没有比较,比如默认的层级聚类在几套数据中表现较好,可以与其他方法比较作为标准。
(4)WGCNA中鉴定的网络相关的两个nodes是没有方向的
鉴定的网络中有连线的两个nodes,只是相关,没有调控方向。参考:
https://cloud.tencent.com/developer/article/1936465 (生信宝典文章)
https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/ (官方网站)
https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/faq.html (常见问题)
https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/Tutorials/index.html (官方教程) -
相关阅读:
Go语言函数进阶:值传递、引用传递、函数式编程
安装RPM包或源码包
Vulnhub Me-and-My-Girlfriend
【CTFHUB】SSRF绕过方法之靶场实践(二)
无胁科技-TVD每日漏洞情报-2022-8-12
天线设计该如何入门
CH9101国产USB转异步串口芯片兼容替代PL2303GC/PL2303HXD/FT230X/FT232RQ/CY7C65213
基于Vivado和Ego1的密码锁设计
基于SSM框架的ACG动漫周边交易平台设计与实现
你的大型设计文件还在用QQ传输?过期了怎么办?Share Creators快速的大文件传输解决方案
- 原文地址:https://blog.csdn.net/cfc424/article/details/127507286