-
单细胞测序原理
一、单细胞技术与传统技术的差异性
以单细胞分辨率解析其他组学掩盖掉的细胞异质性难题
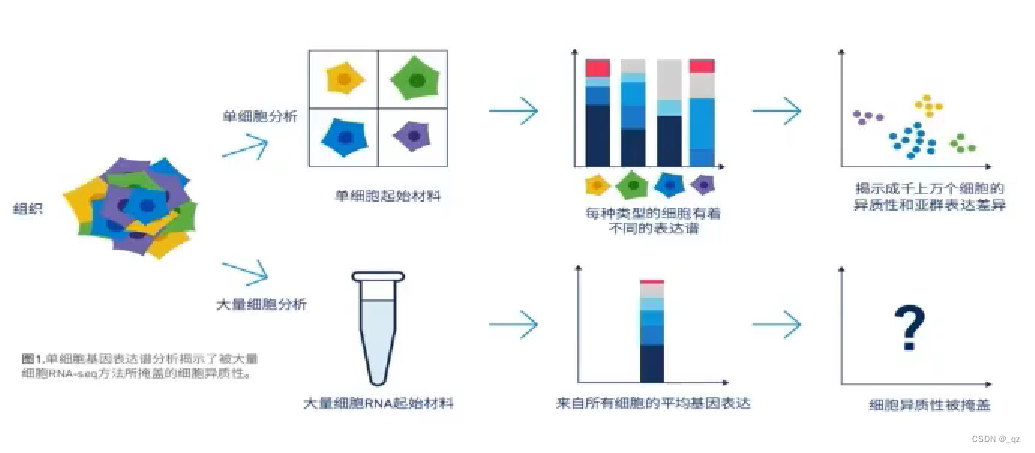
包括细胞类型识别,细胞状态分析,基因特征分析,细胞亚型分析,肿瘤谱系推断,特异性基因鉴定空间转录组

空间转录组同时提供组成异质性和结构异质性信息,主要有空间基因分布表达,marker基因鉴定,不同区域分子空间互作,亚群或组织区域功能分析
二、单细胞测序技术原理
基于标签(barcode)的单细胞识别,给每个细胞加上独一无二的DNA序列,这样在测序的时候,就把携带相同barcode的序列视为来自同一个细胞了。这种策略,可以通过一次建库,测得数百上千个单细胞的信息
每一个油滴中会落入一个细胞以及一个凝胶微珠,那么在每一个凝胶微珠中上长满了不同的Cell Barcode和UMI Barcode连接形成的序列,再加上一端PolyT的抓手,构成我们的捕获凝胶微珠。而这个凝胶微珠抓手就会使用oligo dT抓住mRNA构建文库
10X的基本技术原理:一个油滴=一个单细胞=一个凝胶微珠=一个RNA-Seq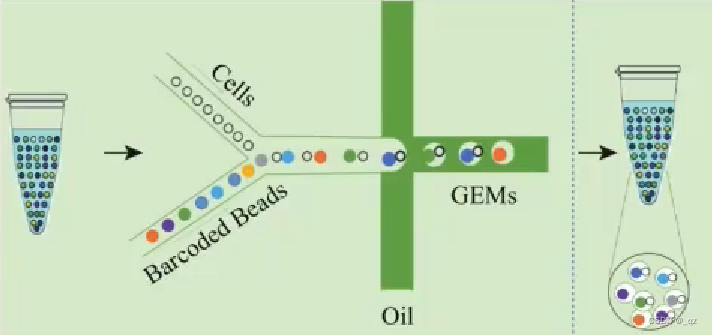
- 含Barcode 的凝胶珠(Gel Beads),细胞和酶的混合物,油三者混合,形成GEMS(油包水的微体系)
- GEMS形成后,细胞裂解,凝胶珠自动溶解释放大量的 Barcode 序列,随后mRNA 逆转录产生带有 Barcode 和 UMI 信息的 cDNA ,基于凝胶珠上的 Cell Barcode和UMI可以将来自同一细胞的mRNA分选出来
- UMI(unique molecular identifiers):UMIs 是由 4-10 个随机核苷酸组成的序列,在 mRNA 反转录后,进入到文库中,每一个 mRNA,随机连上一个 UMI,因此可以计数不同的 UMI,最终计数 mRNA 的数量。
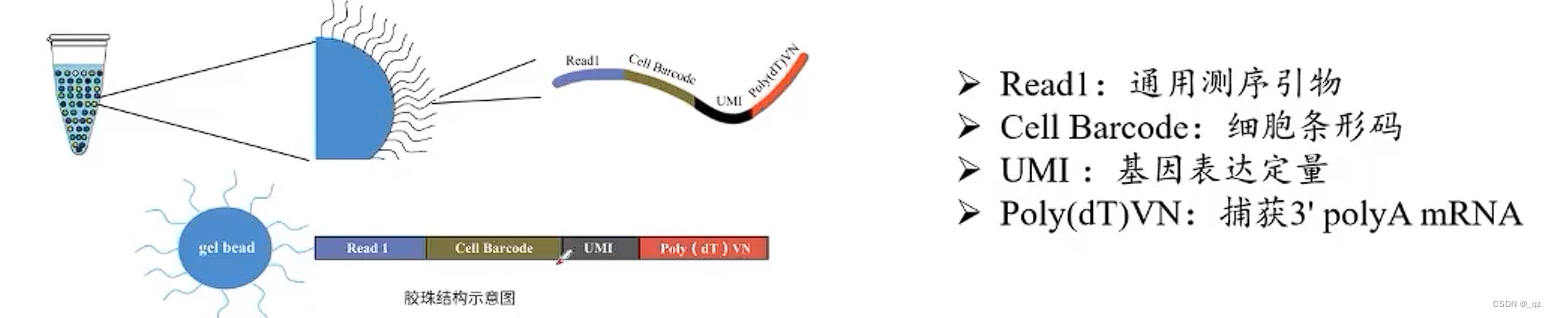
问题: 单细胞测序并非是单独细胞一个个的测序,也是将 cDNA 提取出来之后混合在一起测序,那么怎样区分某个RNA是来自哪个细胞的?混合完之后怎样还原到原来细胞中去?
方法: 通过Ploy(dT)VN 这段序列去反转录,然后捕获 mRNA ,反转录结束之后每个RNA分子都带有cell Barcode的标签,同一个细胞里面的cell Barcode是一样的
三、空间转录组技术原理
借助spatial barcode 技术将RNA分子还原到原来的空间位置
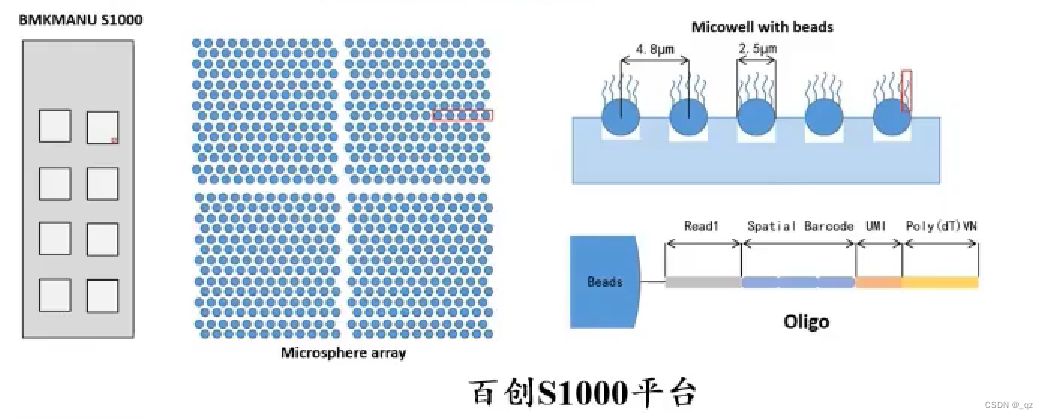
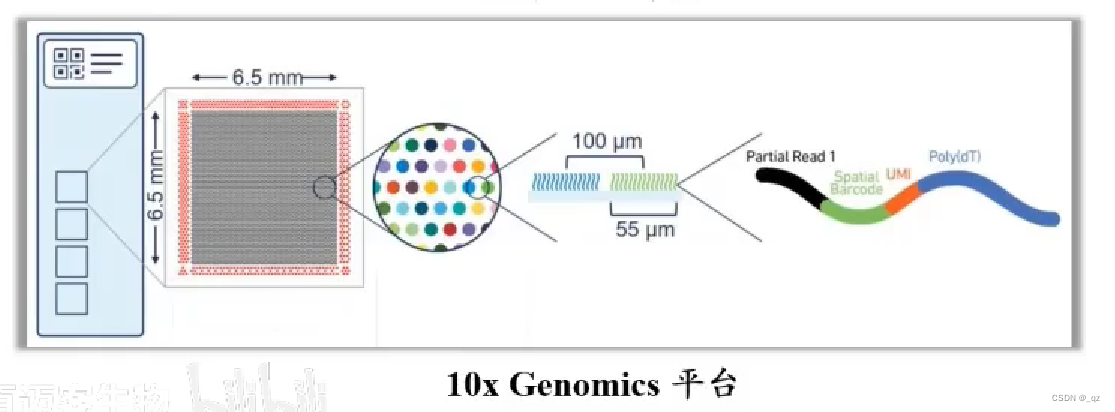
- 基于微孔微珠上 spatial barcode 特定的序列实现空间定位,检测组织切片上捕获到的3`mRNA空间基因表达
- 每个spot 包含spatial barcode
- 组织切片的细胞会释放mRNA,迁移到每个spot的mRNA会被标记上spatial barcode序列
- 根据数据中的spatial barcode信息对数据进行分析,以确定哪些数据来自哪个位置,从而实现空间基因表达的可视化
REF:
https://www.cnblogs.com/emanlee/p/14933354.html
https://blog.csdn.net/weixin_46021869/article/details/115733190 -
相关阅读:
只需3个步骤,轻松解决程序员在Java中生成、扫描二维码难题
C--小Why的商品归位-- 牛客小白月赛77
CAD(计算机辅助设计)软件的开发框架
平衡树之B树
docker day04
Smallest number(dfs全排列)
Python第三方库 - Flask(python web框架)
【报表实战篇】格间计算性能提升方案
标签页的使用
Fast Unsupervised Projection for Large-Scale Data
- 原文地址:https://blog.csdn.net/m0_45210226/article/details/127708821