-
lapply apply详解sapply mapply 列表变成数据框 数据框变成列表 list和dataframe
- a_list=rna_msc_match
- do.call(cbind, lapply(lapply(a_list, unlist), `length<-`, max(lengths(a_list))))
- # cat months dog
- my_list=a_list
- # 假设你的列表名为my_list,包含相同列名的数据框
- # 例如:my_list <- list(df1, df2, df3, ...)
- # 其中 df1, df2, df3 是数据框
- # Step 1: 合并数据框
- combined_df <- do.call(rbind, my_list)
- # Step 2: 添加列表名
- combined_df$List_Name <- unlist(lapply(seq_along(my_list), function(i) rep(names(my_list)[i], nrow(my_list[[i]]))))
- a
- combined_df
lapply
曾经有一位大神讲过,区分R语言是否进阶的标准是,能否用好apply。这个大神叫Jimmy。
其实,我们在之前用apply去批量做过生存分析,当时为了提升速度,还是用了R语言的并行策略,parApply
lapply(X, FUN, ...)
输入的内容是X中的一个元素,(遍历X中的每个元素,X通常是超过两个元素的向量)
输出的结果为list
apply家族中还有一个更加神奇运用更广泛的成员,他的名字叫lapply,也就是list + apply,顾名思义,他返回的数据是列表
我们看看他的三个小应用:
第一,批量操作。
加入我想读入一下四个CSV格式的文件,
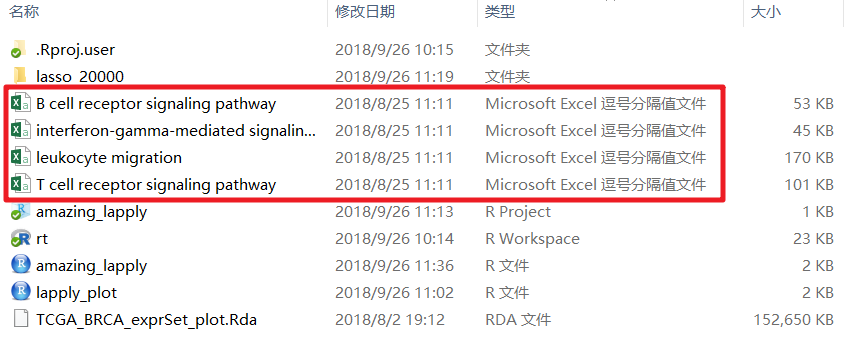
我们可以一个一个的读:
-
- fcsv1 = read.csv("B cell receptor signaling pathway.csv")
- fcsv2 = read.csv("interferon-gamma-mediated signaling pathway.csv")
- fcsv3 = read.csv("leukocyte migration.csv")
- fcsv4 = read.csv("T cell receptor signaling pathway.csv")

如果有1000个文本呢,理论上对于一个会编程的人,重复的事情超过三次,他就受不了开始编写批量操作的脚本了,哪怕这个事情最终只要重复四次就可以完成,
这不是作秀,这是态度,关乎程序员的尊严。
如果用上lapply呢? 首先我们把需要读取的文件名称提取出来
-
files = list.files(pattern="*.csv") -
files
文件名称是这个样子的
-
- > files
- [1] "B cell receptor signaling pathway.csv"
- [2] "interferon-gamma-mediated signaling pathway.csv"
- [3] "leukocyte migration.csv"
- [4] "T cell receptor signaling pathway.csv"
现在我们批量读取,x 是需要批量处理的因素,FUN表示施加的功能,这里是函数
lapply(X, FUN, ...)
fcsv <-lapply(files,read.csv)读取进去后,这四个文件已列表的形式存在于fcsv中,我们还可以对他命名
names(fcsv) <- files一般情况下,批量读取数据框后,还需要把他们合并,如果每一个文件的抬头一样,我们需要把文件按照行合并 这时候会得到一个大的数据框
要实现这个操作有四种方法: 分别是ldply,do.call,dplyr包中的bind_rows,data.table包中的rbindlist,
实际上最常用的是前面两个, 我最开始使用的是do.call, 这些方法也是变化无穷的
-
exp_df1 <- plyr::ldply(fcsv, data.frame) -
exp_df2 = data.frame(do.call(rbind,fcsv)) -
exp_df3 = dplyr::bind_rows(fcsv) -
exp_df4 = data.table::rbindlist(fcsv)

最终都能实现功能,其中ldply会把文件名称作为单独的一列,所以多一列 ,而其他方法把文件名加上系数作为行名。
实际上lapply只是实现批量操作,配套的函数,function才是他神奇的点睛之笔,我们可以用内置函数,比如,read.csv 也可以用自己定义的函数,这个在第三条的时候再说。
第二,批量读取Rdata数据。
把数据储存成Rdata是我R语言路上一个豁然开朗的瞬间,也是伟人Jimmy教给我的。
从此我告别了把数据在R语言里面写来写去的日子,所有中间数据保存成Rdata,一直到出图为止。
假如我的工作目录中有这个文件,导入是这个样子的,用load函数就行
load("Lasso_df_20000_01.Rdata")那么如何批量读取Rdata呢,这里面有个小坑。
-
files = list.files(pattern="*.Rdata") -
files
有6个文件需要读取
-
> files -
[1] "Lasso_df_20000_01.Rdata" "Lasso_df_20000_02.Rdata" -
[3] "Lasso_df_20000_03.Rdata" "Lasso_df_20000_04.Rdata" -
[5] "Lasso_df_20000_05.Rdata" "Lasso_df_20000_06.Rdata"
按照之前的思路,用lapply
fload1 = lapply(files,load)可以读取,但是读取到的是文件名称,不是内容,这是因为load会把文件存到临时地点,lapply会把他破坏掉,
下面的方法的两种方法可以把数据导入进来,任意选取一种,就是告诉他读取到全局环境变量中。
- fload2 = lapply(files,load,.GlobalEnv)
- fload3 = lapply(files,load,environment())

数据被读取进来,但是没有被存入list中,这时候我们需要用get函数获取到内容
fload = lapply(files, function(x) get(load(x)))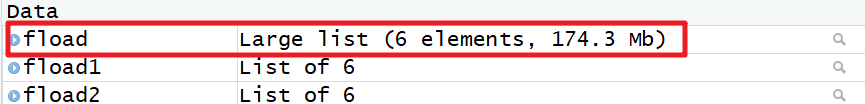
这就成功了,数据加起来有174Mb啊,同样的,也有四种方法可以把他们合并
-
df1 = plyr::ldply(fload, data.frame) -
df2 = data.frame(do.call(rbind,fload )) -
df3 = dplyr::bind_rows(fload ) -
df4 = data.table::rbindlist(fload)
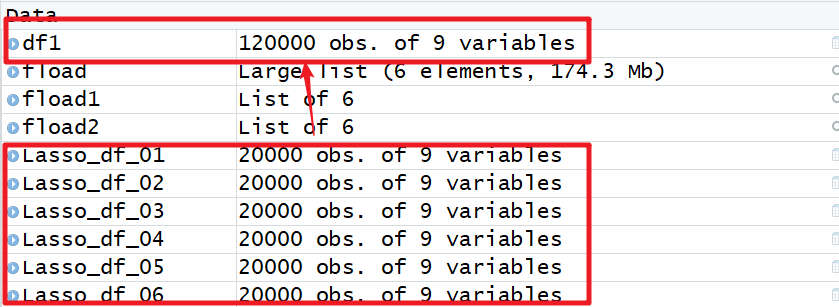
这样就很方便了。
第三,批量作图。
只要lapply后面的函数不一样,就可以实现很多批量操作,
我们以批量作图这种直观的方式阐述以下如何自定义函数
本次还是使用以前的那个肿瘤表达数据,这个数据被用过至少3次
我们加载数据,加载R包,定义要作图的基因
-
load(file = "TCGA_BRCA_exprSet_plot.Rda") -
library(ggstatsplot) -
genes <- c("BRCA1","ESR1","TP53","ERBB2")
现在我们定义一个作图的函数,输入基因就出图
-
- tnplot <- function(gene,exprSet){
- require(ggstatsplot)
- ggbetweenstats(data = exprSet,
- x = sample,
- y = gene)
- }
我们输入一个基因测试一下,发现可以
tnplot("BRCA1",exprSet)
现在我们批量操作,还是用lapply,只不过当函数有多个参数的时候,第一个以外的写在函数后面,以逗号分隔,三个四个都可以
在本例中,expreSet这个数据集就是第二个参数,读取完毕后,所有的作图数据存在p1中。
-
p1 <- lapply(genes, tnplot,exprSet)
批量作图展示
-
library(cowplot) -
plot_grid(plotlist=p1,nrow =2,labels = LETTERS[1:4])

是不是很赞,当然,lapply实现的是批量,具体有哪些惊艳的操作,取决于我们如何写出使用的function。
而函数是R语言进阶的另外一道坎,我也给自己丢下一个必填的坑。
Until next time,it‘s goodbye!

-
相关阅读:
操作系统4小时速成:处理机调度,调度方法,调度准则,典型的调度算法,响应比
Flutter 教程之 如何在app中启用 Google 支付
【LeetCode热题100】--240.搜索二维矩阵II
HTTP 协商缓存 Last-Modified,If-Modified-Since
Python基础之dict和set的使用
【环境搭建】linux docker安装nexus3
[100天算法】-键值映射(day 42)
希亦CG声波清洗机:眼镜党福利,家庭必备清洗机
Jtti:windows虚拟机如何设定永久静态路由
插画、插图网站,免费(商用)
- 原文地址:https://blog.csdn.net/qq_52813185/article/details/127589464