-
R语言和医学统计学(9):多重检验
本文首发于公众号:医学和生信笔记,完美观看体验请至公众号查看本文。
医学和生信笔记,专注R语言在临床医学中的使用,R语言数据分析和可视化。
前言
这是R语言和医学统计学的第9篇内容。
主要是用R语言复现课本中的例子。我使用的课本是孙振球主编的《医学统计学》第4版,封面如下:

多个样本均数间的多重比较
使用课本例4-2的数据。
首先是构造数据,本次数据自己从书上摘录。。
trt<-c(rep("group1",30),rep("group2",30),rep("group3",30),rep("group4",30)) weight<-c(3.53,4.59,4.34,2.66,3.59,3.13,3.30,4.04,3.53,3.56,3.85,4.07,1.37, 3.93,2.33,2.98,4.00,3.55,2.64,2.56,3.50,3.25,2.96,4.30,3.52,3.93, 4.19,2.96,4.16,2.59,2.42,3.36,4.32,2.34,2.68,2.95,2.36,2.56,2.52, 2.27,2.98,3.72,2.65,2.22,2.90,1.98,2.63,2.86,2.93,2.17,2.72,1.56, 3.11,1.81,1.77,2.80,3.57,2.97,4.02,2.31,2.86,2.28,2.39,2.28,2.48, 2.28,3.48,2.42,2.41,2.66,3.29,2.70,2.66,3.68,2.65,2.66,2.32,2.61, 3.64,2.58,3.65,3.21,2.23,2.32,2.68,3.04,2.81,3.02,1.97,1.68,0.89, 1.06,1.08,1.27,1.63,1.89,1.31,2.51,1.88,1.41,3.19,1.92,0.94,2.11, 2.81,1.98,1.74,2.16,3.37,2.97,1.69,1.19,2.17,2.28,1.72,2.47,1.02, 2.52,2.10,3.71) data1<-data.frame(trt,weight) data1$trt <- factor(data1$trt) str(data1) ## 'data.frame': 120 obs. of 2 variables: ## $ trt : Factor w/ 4 levels "group1","group2",..: 1 1 1 1 1 1 1 1 1 1 ... ## $ weight: num 3.53 4.59 4.34 2.66 3.59 3.13 3.3 4.04 3.53 3.56 ...- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
数据一共两列,第一列是分组(一共四组),第二列是低密度脂蛋白测量值
进行完全随机设计资料的方差分析:
fit <- aov(weight ~ trt, data = data1) summary(fit) ## Df Sum Sq Mean Sq F value Pr(>F) ## trt 3 32.16 10.719 24.88 1.67e-12 *** ## Residuals 116 49.97 0.431 ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1- 1
- 2
- 3
- 4
- 5
- 6
- 7
LSD-t检验
library(agricolae) res <- LSD.test(fit, "trt", p.adj = "none") # LSD-t检验 res ## $statistics ## MSerror Df Mean CV t.value LSD ## 0.4307502 116 2.7025 24.2855 1.980626 0.3356368 ## ## $parameters ## test p.ajusted name.t ntr alpha ## Fisher-LSD none trt 4 0.05 ## ## $means ## weight std r LCL UCL Min Max Q25 Q50 Q75 ## group1 3.430333 0.7151247 30 3.193002 3.667664 1.37 4.59 2.9650 3.530 3.9825 ## group2 2.715333 0.6381586 30 2.478002 2.952664 1.56 4.32 2.3175 2.665 2.9650 ## group3 2.698000 0.4971671 30 2.460669 2.935331 1.68 3.68 2.3375 2.655 2.9800 ## group4 1.966333 0.7464421 30 1.729002 2.203664 0.89 3.71 1.3350 1.905 2.4225 ## ## $comparison ## NULL ## ## $groups ## weight groups ## group1 3.430333 a ## group2 2.715333 b ## group3 2.698000 b ## group4 1.966333 c ## ## attr(,"class") ## [1] "group"- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
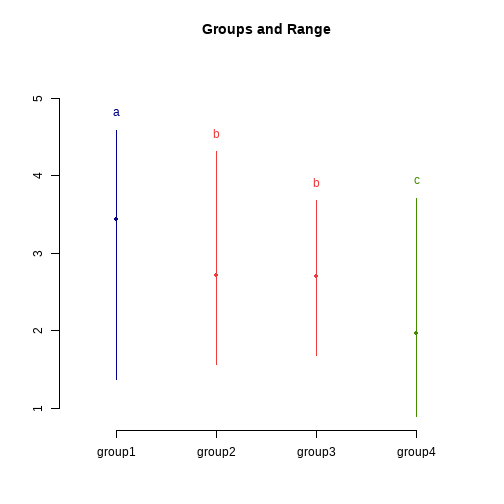
只看
$groups下面的输出,这个结果解读和SPSS差不多,后面标相同字母的是没有差异的。所有
group2和group3是没差别的,和另外两组有差别。还可以可视化结果:
plot(res)- 1
TukeyHSD
这里介绍一种
TukeyHSD方法:TukeyHSD(fit) ### 每个组之间进行比较,多重比较 ## Tukey multiple comparisons of means ## 95% family-wise confidence level ## ## Fit: aov(formula = weight ~ trt, data = data1) ## ## $trt ## diff lwr upr p adj ## group2-group1 -0.71500000 -1.1567253 -0.2732747 0.0002825 ## group3-group1 -0.73233333 -1.1740587 -0.2906080 0.0001909 ## group4-group1 -1.46400000 -1.9057253 -1.0222747 0.0000000 ## group3-group2 -0.01733333 -0.4590587 0.4243920 0.9996147 ## group4-group2 -0.74900000 -1.1907253 -0.3072747 0.0001302 ## group4-group3 -0.73166667 -1.1733920 -0.2899413 0.0001938- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
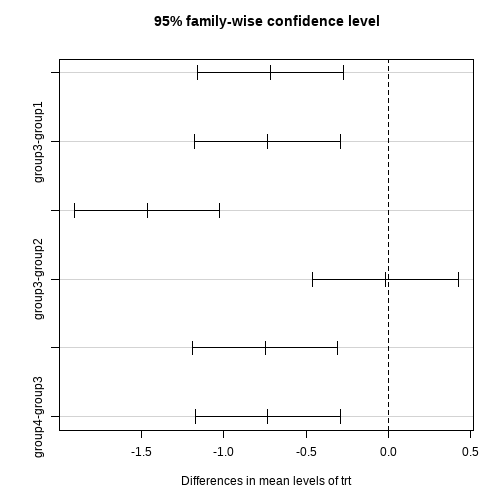
这个结果更直观,可以直接看到每个组之间的比较,后面给出了P值。
可视化结果:
plot(TukeyHSD(fit))- 1
Dunnett-t检验
使用
Dunnett-t检验进行多重比较:library(multcomp) ## 载入需要的程辑包:mvtnorm ## 载入需要的程辑包:survival ## 载入需要的程辑包:TH.data ## 载入需要的程辑包:MASS ## ## 载入程辑包:'TH.data' ## The following object is masked from 'package:MASS': ## ## geyser res <- glht(fit, linfct = mcp(trt = "Dunnett")) summary(res) ## ## Simultaneous Tests for General Linear Hypotheses ## ## Multiple Comparisons of Means: Dunnett Contrasts ## ## ## Fit: aov(formula = weight ~ trt, data = data1) ## ## Linear Hypotheses: ## Estimate Std. Error t value Pr(>|t|) ## group2 - group1 == 0 -0.7150 0.1695 -4.219 0.000148 *** ## group3 - group1 == 0 -0.7323 0.1695 -4.322 0.000115 *** ## group4 - group1 == 0 -1.4640 0.1695 -8.639 < 1e-04 *** ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## (Adjusted p values reported -- single-step method)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
结果也是非常明显,所有组和安慰剂组相比都有意义。
可视化结果:
plot(res)- 1

SNK-q检验
还是使用
agricolae包。library(agricolae) res <- SNK.test(fit, "trt") res ## $statistics ## MSerror Df Mean CV ## 0.4307502 116 2.7025 24.2855 ## ## $parameters ## test name.t ntr alpha ## SNK trt 4 0.05 ## ## $snk ## Table CriticalRange ## 2 2.801028 0.3356368 ## 3 3.357590 0.4023275 ## 4 3.686381 0.4417253 ## ## $means ## weight std r Min Max Q25 Q50 Q75 ## group1 3.430333 0.7151247 30 1.37 4.59 2.9650 3.530 3.9825 ## group2 2.715333 0.6381586 30 1.56 4.32 2.3175 2.665 2.9650 ## group3 2.698000 0.4971671 30 1.68 3.68 2.3375 2.655 2.9800 ## group4 1.966333 0.7464421 30 0.89 3.71 1.3350 1.905 2.4225 ## ## $comparison ## NULL ## ## $groups ## weight groups ## group1 3.430333 a ## group2 2.715333 b ## group3 2.698000 b ## group4 1.966333 c ## ## attr(,"class") ## [1] "group"- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
这个结果和LSD检验的结果解读一样,就不再赘述了!
可视化结果:
plot(res)- 1
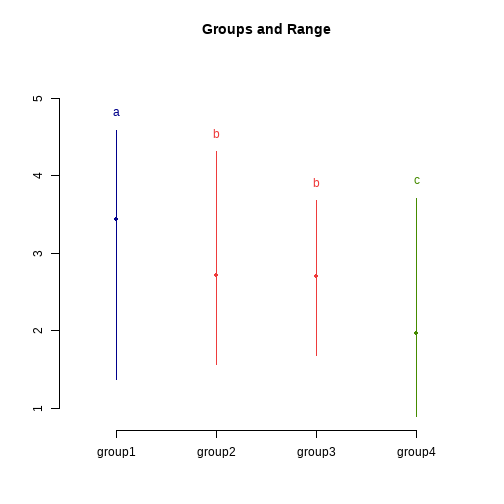
也是和LSD检验一样的!
本文首发于公众号:医学和生信笔记,完美观看体验请至公众号查看本文。
医学和生信笔记,专注R语言在临床医学中的使用,R语言数据分析和可视化。
-
相关阅读:
创新案例分享 | 建立校园大数据治理评价平台,高效提高教学效率与质量
Android studio 打包release版本 apk 换方式了?
【pwn】ez_pz_hackover_2016 --pwngdb和pwntools的结合,动态调试
国自然热点 | 一文了解铁死亡及其研究思路
水浸监测新方法,简单实用,你值得拥有!
使用人性化的Linux防火墙CFW阻止DDOS攻击
丁鹿学堂前端培训:node自动化启动和模块化路由
【Java从入门到精通 07】:面向对象编程(基础部分)
SwiftUI Swift iOS iPadOS 实现更改 App 图标
安装axios时报错
- 原文地址:https://blog.csdn.net/Ayue0616/article/details/127587930
