-
一张图介绍PRS的计算步骤
查看了一下博客和文献,把我的理解总结一下。
PRS是多基因风险评分,下面介绍一下它处理的步骤。

https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7612115/
1. 数据
1.1 基础数据(BASE DATA)
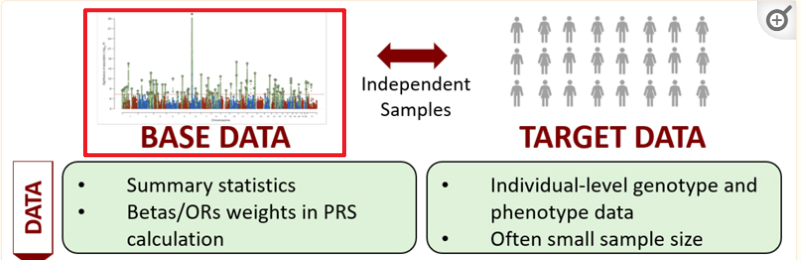
数据包括:- Summary statistics结果
- Betas或者ORs
这部分数据,主要是大样本得到的特定性状的GWAS结果,GWAS summary result,包括snp,染色体,物理位置,maf,effect(或者OR),P值等信息,类似:
连续性状:
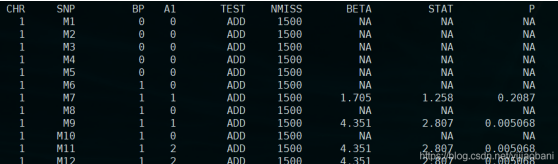
或者:

二分类性状:
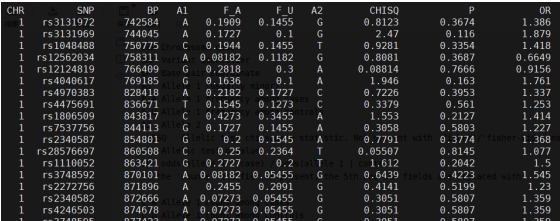
或者:

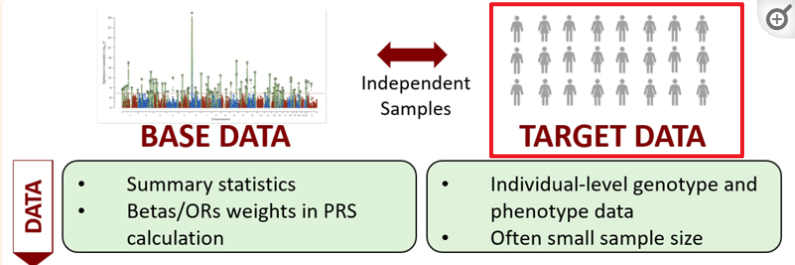
1.2 目标数据(TARGET DATA)
这部分数据就是我们自己搜集的数据,包括:
- 个体的基因型和表型
- 通常数据量不大,个体数据量不大,位点数据量也不大
2. 数据处理

这部分,包括:- 数据的质控,包括Base data的质控和Target data的质控
- 影响PGS的因素要注意,比如样本的重复、关系、以及群体的结构
- 获得目标的SNP位点
具体而言,在进行处理数据前,需要确定性状的遗传特性h2snp 要大于0.05,文件中确定定位基因,对maf和去填充准确性得分进行质控,两个数据基因组版本一致,对于基因型不匹配的可以通过flip翻转,重复的snp删除,性染色体删除,重复样本删除,对于基础数据和目标数据中有交叉的个人删除,亲缘关系近的也删除,确保基础数据和目标数据独立,等操作。
3. PRS计算

包括根据LD去调整,比如修剪(clumping),然后计算Beta校正值,以及调整P值,计算PRS值。具体的做法:
- 对于Beta和OR值,应该对其进行矫正,可以通过LASSO或者岭回归进行收缩(shrinkage),另外,通过P值进行筛选SNP。
- 根据LD去质控SNP,保证中选的SNP独立
4. 测试

这部分,主要是在目标数据中进行测试,查看计算的PRS和实际的表型匹配度如何,计算准确性。5. 验证或者预测

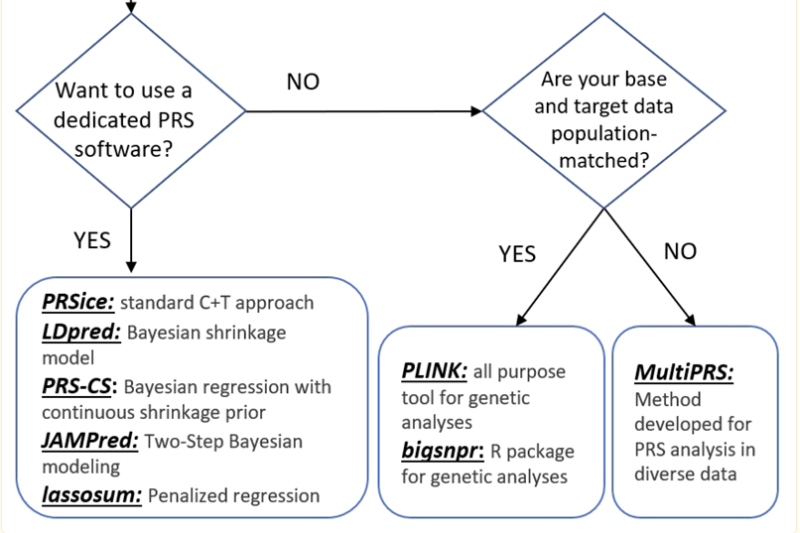
通过测试集的验证,就可以选出PRS模型,进行大样本的预测。6. 相关的软件
- plink
- biqsnpr,一个R包
- PRSice,应用最广泛,通过C+T的策略
- LDpred,通过贝叶斯收缩的模型
- PRS-CS
- JAMPred
- Lassosum
-
相关阅读:
leetcode 286 墙与门
第三篇 部署方式--单机部署
负环判断——虫洞(裸题)
2023 年 API 安全状况
Buffer Pool详解
ECMAScript13 中11个令人惊叹的 JavaScript 新特性
RocketMQ 消费者分类与分组
c++ 对象和类
one-hot和Embedding
【Spark NLP】第 15 章:聊天机器人
- 原文地址:https://blog.csdn.net/yijiaobani/article/details/127439431