-
R语言---PCA/tSNE/UMAP降维计算
目前,需要将不同材料的基因型数据降维,使用线性降维方法PCA,非线性降维方法tSNE和UMAP。
使用的输入输入数据是不同材料基因型的相似性矩阵,期望获得多个SNP,不同材料的降维情况。1. PCA计算
PCA(principal component analysis)是一种
线性降维方法,通常分为R-mode和Q-mode,数据列为variable,行为unit;在R语言中计算PCA,常用princomp和prcomp函数。(1) princomp函数
princomp函数是基于协方差(covariance) 或者相关矩阵(correlation) 提取的特征(eigen)并进行特征值分解。
princomp函数适用行数大于列数的数据(即 units数目大于variables数目),否则报错:> x=t(mtcars) > dim(x) [1] 11 32 > princomp(x) Error in princomp.default(x) : 'princomp' can only be used with more units than variables- 1
- 2
- 3
- 4
- 5
- 6
默认参数为:
## Default S3 method: princomp(x, cor = FALSE, scores = TRUE, covmat = NULL, subset = rep_len(TRUE, nrow(as.matrix(x))), fix_sign = TRUE, ...)- 1
- 2
- 3
在R中键入
?princomp,会发现该函数推荐我们使用prcomp:The calculation is done using eigen on the correlation or covariance matrix, as determined by cor. This is done for compatibility with the S-PLUS result. A preferred method of calculation is to use svd on x, as is done in prcomp.- 1
- 2
- 3
princomp PCA结果:
pca_iris = princomp(iris[,1:4])$scores[,1:2] plot(pca_iris, t='n') text(pca_iris, labels=iris$Species,col=colors[iris$Species])- 1
- 2
- 3

(2) prcomp函数
prcomp函数的计算是基于奇异值分解的(singular value decomposition,svd),对于R/Q-mode数据都可以,不会报错,因此比上面的函数更好用。
默认参数为:## Default S3 method: prcomp(x, retx = TRUE, center = TRUE, scale. = FALSE, tol = NULL, rank. = NULL, ...)- 1
- 2
- 3
该函数有两个参数比较重要:
scale. = TRUE/FALSE (default FALSE) center = TRUE/FALSE (default TRUR)- 1
- 2
scale.这个参数通常设置为TRUE,因为这样可以对数据框进行scale(zscore标准化),防止因为数据的数量级不同对结果产生影响。当然了这个参数设置FALSE,但传入的是预先scale的数据是不影响结果的。
实际情况,还是要看啊,具体并不能确定哪个更好(scale. =T/F)。使用自带的
iris数据框计算举例:## prcomp method:简单出图版本 pca.res <- prcomp(iris[,1:4])$x[,1:2] plot(pca.res, t='n') text(pca.res, labels=iris$Species,col=colors[iris$Species])- 1
- 2
- 3
- 4
## prcomp method:计算percentage pca.res <- prcomp(iris[,1:4]) summary(pca.res) pca.plot=as.data.frame(pca.res$x[,1:2]) colnames(pca.plot)=c("PC1","PC2") ## calculate percentage percent = round(pca.res$sdev^2/sum(pca.res$sdev^2), 4) pc1r=percent[1] pc2r=percent[2] plot(pca.plot, t='n',xlab=paste("PC1","(",round(pc1r * 100, 2),"%)"), ylab=paste("PC2","(",round(pc2r * 100, 2),"%)")) text(pca.plot, labels=iris$Species,col=colors[iris$Species])- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13

2. tSNE计算
tSNE(t-distributed stochastic neighbor embedding)是一种
非线性降维方法,t-SNE本质是将高维数据映射到低维空间,保留数据的局部特性。(1)使用Rtsne包
## 使用前需要先下载Rtsne require(Rtsne) ## 默认参数如下: ## Default S3 method: Rtsne( X, dims = 2, initial_dims = 50, perplexity = 30, theta = 0.5, check_duplicates = TRUE, pca = TRUE, partial_pca = FALSE, max_iter = 1000, verbose = getOption("verbose", FALSE), is_distance = FALSE, Y_init = NULL, pca_center = TRUE, pca_scale = FALSE, normalize = TRUE, stop_lying_iter = ifelse(is.null(Y_init), 250L, 0L), mom_switch_iter = ifelse(is.null(Y_init), 250L, 0L), momentum = 0.5, final_momentum = 0.8, eta = 200, exaggeration_factor = 12, num_threads = 1, ... )- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
perplexity 默认为30,该参数有限制,perplexity参数值必须小于(nrow(data) - 1 )/ 3:numeric; Perplexity parameter (should not be bigger than 3 * perplexity < nrow(X) - 1, see details for interpretation)- 1
theta默认为0.5,设置该值越小越准确,但是计算时间长,因此多数设置为0.0.pca默认为TRUE表示是否对输入的原始数据进行PCA分析,然后使用PCA得到的topN主成分进行后续分析,默认采用top50的主成分进行后续分析,当然也可以通过initial_dims参数修改这个值。
为保持结果一致,需要设置随机种子。显然下面这两个参数是设置pca计算的,与pca计算参数含义相同:
pca_center = TRUE, pca_scale = FALSE,- 1
- 2
详细参数选择请参考:
https://distill.pub/2016/misread-tsne/- 1
应用举例:
set.seed(1) # 设置随机数种子 require(Rtsne) ## Rtsne package iris.data <- iris[, grep("Sepal|Petal", colnames(iris))] iris.labels <- iris[, "Species"] test=unique(iris.data) ## remove duplicates, else error occurs tsne_test = Rtsne( test, dims = 2, theta = 0, pca=T, ## 调用pca结果,计算快。 pca_center = TRUE, pca_scale = F, perplexity = 30, ## 30 verbose = F) # 进行t-SNE降维分析 tsne_result = as.data.frame(tsne_test$Y) dim(tsne_result) colnames(tsne_result) = c("tSNE1","tSNE2") plot(tsne_result,col=as.factor(iris.labels), pch=20,cex=0.7) text(tsne_result,labels=iris$Species, col=colors[iris$Species])- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21

(2)使用tsne包
还存在另一个R包tsne,也可以进行tsne计算,只不过这个貌似没有pca参数系列,只是原始值计算,所以速度较慢。
默认参数:tsne(X, initial_config = NULL, k = 2, initial_dims = 30, perplexity = 30, max_iter = 1000, min_cost = 0, epoch_callback = NULL, whiten = TRUE, epoch=100)- 1
- 2
- 3
应用举例(iris数据):
colors = rainbow(length(unique(iris$Species))) names(colors) = unique(iris$Species) ecb = function(x,y){ plot(x,t='n'); text(x,labels=iris$Species, col=colors[iris$Species]) } set.seed(1) ## 设定随机数 tsne_iris = tsne(iris[,1:4], epoch_callback = ecb, perplexity=50)- 1
- 2
- 3
- 4
- 5

要想保持结果一致,需要设定随机数种子,否则会不同。
Rtsne中默认先进行pca计算,然后作为Rtsne输入,运算速度更快;
tsne中未使用pca,迭代较慢。个人偏向Rtsne。3. UMAP计算
UMAP(uniform manifold approximation and projection for dimension reduction)是一种非线性降维方法,同时考虑了局部距离和整体。
默认参数:require(umap) ## umap params custom.config = umap.defaults ## 修改部分参数: custom.config$random_state = 123 ## 设定随机数 custom.config$n_neighbors =15 ## 设定邻接数目- 1
- 2
- 3
- 4
- 5
- 6
- 7
全部默认参数为:
> custom.config umap configuration parameters n_neighbors: 15 n_components: 2 metric: euclidean n_epochs: 200 input: data init: spectral min_dist: 0.1 set_op_mix_ratio: 1 local_connectivity: 1 bandwidth: 1 alpha: 1 gamma: 1 negative_sample_rate: 5 a: NA b: NA spread: 1 random_state: NA transform_state: NA knn: NA knn_repeats: 1 verbose: FALSE umap_learn_args: NA- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
参数
min_dist: 0.1控制局部距离,
参数n_neighbors: 15控制整体,
参数random_state控制随机数以
iris数据为例:
代码如下:library(umap) iris.data <- iris[, grep("Sepal|Petal", colnames(iris))] iris.labels <- iris[, "Species"] head(iris.labels) iris.umap <- umap(iris.data) ## 使用默认参数: config=umap.defaults plot.data = as.data.frame(iris.umap$layout) colnames(plot.data)=c("umap1","umap2") head(plot.data) plot(plot.data,col=as.factor(iris.labels), pch=20,cex=0.7)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11

设置随机数为123,重新绘图:
代码为:library(umap) config.params = umap.defaults config.params$random_state=123 ## 设置随机数 iris.umap <- umap(iris.data, config = config.params) plot.data = as.data.frame(iris.umap$layout) colnames(plot.data)=c("umap1","umap2") head(plot.data) plot(plot.data,col=as.factor(iris.labels), pch=20,cex=0.7)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
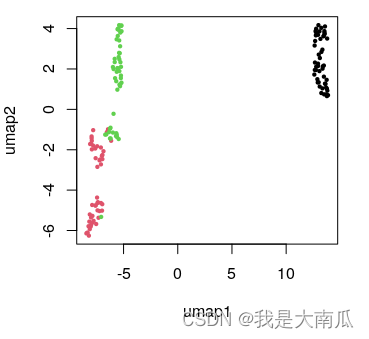 上面参数修改方法在umap函数内也行;
上面参数修改方法在umap函数内也行;iris.umap <- umap(iris.data, random_state=123) ## 直接函数内改变默认参数也可以- 1
但有一点要注意,即便后面参数相同,如果输入的数据改变顺序,结果也会不同:
代码如下:library(umap) iris.data <- iris[, grep("Sepal|Petal", colnames(iris))] ## 增加重新排序,使原始数据顺序不同,结果也会不同 iris.data <- iris.data[order(iris.data$Sepal.Length),] iris.labels <- iris[, "Species"] config.params = umap.defaults config.params$random_state=123 iris.umap <- umap(iris.data, random_state=123) plot.data = as.data.frame(iris.umap$layout) colnames(plot.data)=c("umap1","umap2") plot(plot.data,col=as.factor(iris.labels), pch=20,cex=0.7)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13

此外,可以进行波动数据映射到基础数据降维的umap结果中:iris.data <- iris[, grep("Sepal|Petal", colnames(iris))] iris.labels <- iris[, "Species"] library(umap) config.params = umap.defaults config.params$random_state=123 iris.umap <- umap(iris.data, random_state=123) plot.data = as.data.frame(iris.umap$layout) colnames(plot.data)=c("umap1","umap2") plot(plot.data,col=as.factor(iris.labels), pch=20,cex=0.7) iris.wnoise <- iris.data + matrix(rnorm(150*40, 0, 0.1), ncol=4) colnames(iris.wnoise) <- colnames(iris.data) head(iris.wnoise, 3) head(iris.data) iris.wnoise.umap <- predict(iris.umap, iris.wnoise) head(iris.wnoise.umap, 3) points(iris.wnoise.umap, col=as.factor(iris.labels), pch=4,cex=0.7)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
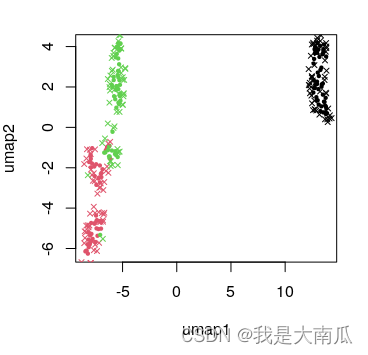
为保持结果的可重复性,umap结果需要设置随机种子,原始数据顺序也不能改变;
此外umap的输入数据可以为data,也可以是dist距离;
umap的knn也可以从已有的结果中提取,参考官方文档。4. 总结
(1)PCA中(princomp和prcomp),推荐使用
prcomp,使用范围更广;
(2)tSNE中(Rtsne和tsne)推荐Rtsne,参数更明确,可以选择pca模式(默认为pca模式),计算更快。二者均需要额外设定随机种子确保结果可重复;
(3)UMAP中参数设定可以通过外部参数设置,或者函数内设置,可以尝试更多参数,选择合适的结果。总之,方法无好坏,数据特性不同,则适用不同方法。实际应用起来可能就需要多尝试了,结合实际问题选择。
本文也只是粗浅的过一遍,实际背后原理的这些,还得继续学习。参考:
https://zhuanlan.zhihu.com/p/496823203 (PCA)
https://cloud.tencent.com/developer/article/1635055 (PCA)
https://stats.stackexchange.com/questions/268264/normalizing-all-the-variarbles-vs-using-scale-true-option-in-prcomp-in-r (PCA scale)
https://cloud.tencent.com/developer/article/1556202 (Rtsne)
https://stats.stackexchange.com/questions/222912/how-to-determine-parameters-for-t-sne-for-reducing-dimensions (Rtsne)
https://distill.pub/2016/misread-tsne/ (Rtsne parameters setting)
https://cran.r-project.org/web/packages/umap/vignettes/umap.html (R umap example) -
相关阅读:
es查询初学
数据库中的范式
net-java-php-python-大学生互助旅游网站修改计算机毕业设计程序
2022/7/27 考试总结
前端品优购项目准备工作
优思学院|质量工程师:他们的使命与职责
IDEA类和方法注释模板设置
计算机毕业设计jsp教师课堂教学评价系统Myeclipse开发mysql数据库web结构java编程计算机网页项目
基于Qt HTTP应用程序项目案例
猜年龄小游戏(while 和 if的运用)
- 原文地址:https://blog.csdn.net/cfc424/article/details/126853847
