-
2018 Journal of cheminformatics | 基于条件变分自编码器分子生成模型
2018 Journal of cheminformatics | Molecular generative model based on conditional variational autoencoder for de novo molecular design

Paper:https://jcheminf.biomedcentral.com/articles/10.1186/s13321-018-0286-7
Code:https://github.com/jaechanglim/CVAE基于条件变分自编码器分子生成模型
本论文由Jaechang Lim等人 2018年发表于化学信息学杂志,其主要工作是提出了一种基于条件变分自编码器的分子生成模型,用于从头设计分子。证明它可以用于生成具有五种靶标性质的类药物分子。并且能够在不改变其他属性的情况下调整单个属性。
模型
条件变分自动编码器 (CVAE)
首先,作者阐明 VAE 和 CVAE 之间的区别,它们的目标函数相互比较。VAE 的目标函数由下式给出:
E [ log P ( X ∣ z ) ] − D K L [ Q ( z ∣ X ) ∥ P ( z ) ] , E[logP(X|z)]−DKL[Q(z|X)∥P(z)],
E[logP(X∣z)]−DKL[Q(z∣X)∥P(z)],其中 E E E表示一个期望值, P P P和 Q Q Q为概率分布,DKL为Kullback-Leibler散度, X X X和 z z z分别表示数据和潜在空间。第一项和第二项通常分别称为重建误差和 KL 项。CVAE 与 VAE 的一个关键区别在于将条件信息嵌入到 VAE 的目标函数中,从而得到修改后的目标函数如下:
E [ log P ( X ∣ z , c ) ] − D K L [ Q ( z ∣ X , c ) ∥ P ( z ∣ c ) ] , E[logP(X|z,c)]−DKL[Q(z|X,c)∥P(z|c)],
E[logP(X∣z,c)]−DKL[Q(z∣X,c)∥P(z∣c)],其中c表示条件向量。条件向量c直接参与编码和解码过程。
CVAE 模型与联合训练的 VAE 模型的主要区别在于,分子特性直接合并到编码器和解码器中。两部分组成:
- 用于目标分子性质,
- 涉及分子结构和其他性质。
分子表示和模型构建
用 SMILES 代码表示分子,以利用专门处理文本和序列的最先进的深度学习技术。每个 SMILES 代码都被规范化为一个独特的分子表示。
数据
ZINC 数据集,
结果

CVAE 生成的分子,其条件向量由a阿司匹林和b达菲的五个目标特性组成。

由 CVAE 生成的分子,其条件向量由阿司匹林的五个目标特性组成,潜向量由阿司匹林的潜向量稍作修改。图中显示结果,从阿司匹林以这种方式产生的分子。它们看起来与阿司匹林非常相似,并且在 10% 的误差范围内也具有与阿司匹林相似的特性。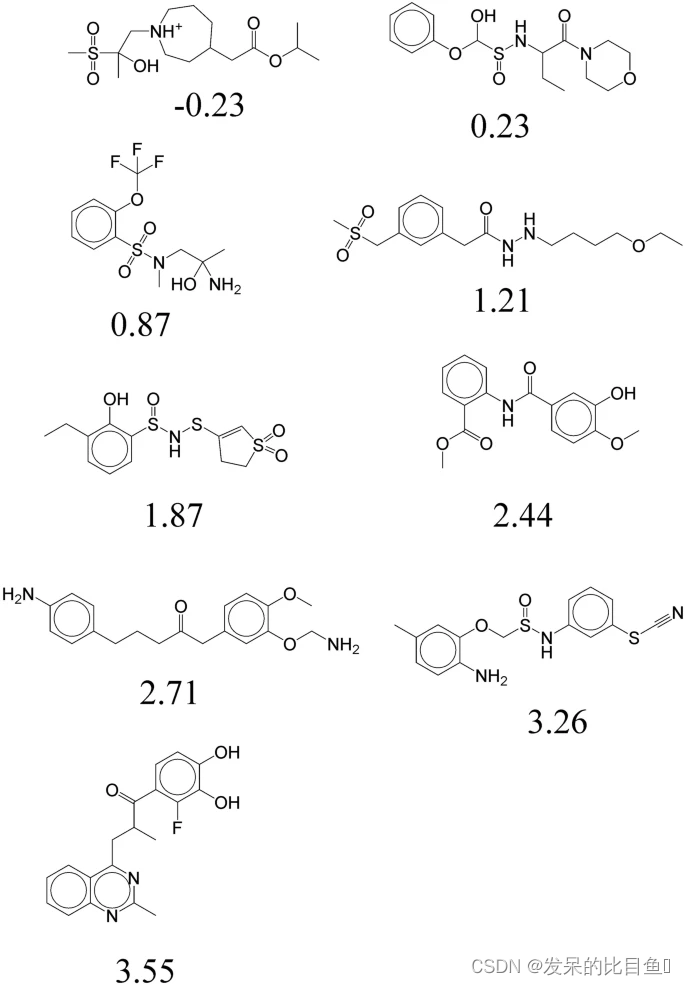

比较了从训练集中随机选择的 1000 个分子和 1000 个生成的分子的 LogP 和 TPSA 分布,这些分子的属性值超出了数据集的范围(朝向更大的值)。
目标属性的分布向更大的值移动,导致属性值超出范围的分子比例增加。
从测试集中随机选择的 1000 个分子的潜在向量的两个分量及其 MW、LogP 和 TPSA 值。具有相似性质的分子可能位于联合训练的 VAE 中潜在空间的同一区域周围。 -
相关阅读:
latex小节标题如何靠左显示
好用的问卷工具有什么?5款工具盘点
利用css var函数让你的组件样式输出规范样式API,可定制性更高;
以太坊合并的3位赢家和2位输家
剑指Offer 09.用两个栈实现队列
JAVA计算机毕业设计智能化车辆管理综合信息平台Mybatis+源码+数据库+lw文档+系统+调试部署
Flask 学习-96.Flask-SQLAlchemy 判断查询结果是否存在的几种方式
Kafka详解
C语言进阶——程序环境和预处理
护眼灯显色指数怎么选?台灯显色指数Ra95和98哪个好
- 原文地址:https://blog.csdn.net/weixin_42486623/article/details/126700604