-
计算机智能专题-遗传算法(1不带约束的)
前言:
遗传算法是一种群智能优化算法,它主要解决的是排列组合问题。通过模仿遗传学中染色体的交叉、变异、并通过“优选”,使父代产生更优的“子代”,在不断的“优胜略汰”中,最终得到最优的组合。更新ing,,,,
本篇文章讲的是:遗传算法的整数编码,原理较为简单可以用作入门。
第二篇遗传算法“实数编码”,是模仿二进制交叉和编译,适用范围更广泛。
一、基本概念
1.编码:
>>生物学定义:DNA中遗传信息在一个长链上按一定的模式排列
>>计算机学定义:将实际问题抽象成"染色体",所建立的数学模型,具体来讲,实际问题中的排列组合问题,并不是涉及染色体,要使用遗传算法解决问题需要进行模型转化。
2.解码:
>>生物学定义: 从遗传DNA到表现型的映射
>>计算机学定义:实际问题通过“编码”过程映射为染色体并使用遗产算法处理之后,需要重新映射到实际问题中,来获得真实的自由组合,这个过程叫做解码。
3.交叉
>>生物学定义:在两个染色体的某一相同位置处DNA被切断,其前后两串分别交叉组合形成两个新的染色体。
>>计算机学定义:在实际的操作中会模仿这样的操作,对编码后的数据进行交叉。
4.变异:
>>生物学定义:很小概率产生基因突变,产生出新的染色体,然后表现出新的性状
5.适应度
度量某个物种对于生存环境的适应程度。对于生存化境适应程度较高的物种将获得更多的繁殖机会,反之机会较少,甚至灭绝。
6.选择
指定以一定的概率从种群中选择若干个体进行"优胜略汰"。
二、基本流程

三、具体实例
3.1一个连续问题的实列
实现遗传算法求解:
(x−5)(x−10)(x−6)(x−16)+21
=-x^4+37*x^3-476*x^2+2540*x-4779
的最大值,其中x为0到31之间的整数,
f = lambda x:-x**4+37*x**3-476*x**2+2540*x-4779函数图像如下图所示:

这里做下说明,对于给出的这个实际问题,可以通过遍历循环得到0到31中满足要求的值,这里通过简单的示例,解释用遗传算法解决这个问题。
1. 参数初始化
>>遗传算法进化迭代次数epochs,这个根据遗传算迭代收敛的情况而定。
>>种群个体数N,设N=8。这个根据经验设置,作为超参数调试。
>>个体的编码长度M=5,这个根据实际问题的需要求而来,一般设置为最少能够用二进制表示的数码范围。[0,31]32位<=
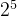 .
.2. 种群初始化
这里已经隐含了编码过程,十进制到二进制。
对于这个编码过程做简单的解释:
后面的遗传算法使用,需要进行染色体的交叉、编译操作,而二进制作为和染色体DNA最接近的数制要比十进制更符合操作习惯,将十进制转化为二进制后,二进制的每一位代表一个染色片段,而“交叉”“变异”的操作是以染色体片段为单位进行的。下面结合一个例子讲解种群初始化的过程。
初始化代码如下,产生一个N*M的矩阵,每一行代表一个个体。
- def __init__(self,N=8,M=5):
- _groups = np.zeros([N,M],int)
- for index in range(0,N):
- for _index in range(0,M):
- _groups[index,_index] = np.random.choice([0,1])
- self._groups = _groups

说明:
在这N*M的矩阵中,每一行是一个1*M的向量,这代表一个十进制个体的二进制表示。所以后面应用时还需要设计一个解码器,以便经过遗传算法操作过后的染色体编码可以映射到实际问题中,
这里的解码器,是将二进制转化为十进制的函数:Binary2Decimal
- def Binary2Decimal(self,Binary,include_negative=False):
- '''
- fct: 将二进制转化为十进制
- :param Binary: 输入的二进制数组
- :param include_negative: 最高位是不是 符号位
- :return:
- '''
- sum = 0
- bit_flag = -1 if include_negative==True else 1
- for index in range(1,len(Binary)):
- sum = sum + Binary[-index]*2**(index-1)
- if bit_flag==-1:
- if Binary[0]==1:
- sum = sum * bit_flag
- else:
- sum = sum + 0
- else:
- sum = sum + Binary[0]*2**(index)
- return sum
3. 优选
优选的方法有很多,本文中的问题是:求函数的最大值,所以优化的对象是函数值,最简单的方案是:比较种群中个体间的函数值的大小,根据值的大小进行优胜略汰。
具体的分析:
计算得到的值存在几种情况:
1.全为正、2.全为负、3.有正有负、4.值一样
优选的方式需要考虑这几种情况,通常常用的几种优选的方式有:“赌轮盘”、“锦标赛”、“小生境”
>>赌轮盘:2021.12.23晚上更新一点
按照某个标准,计算得到每一个个体的得分,然后将每个个体分布在一个圆盘上面,个体所占圆心角的大小取决于个体得分所占总分的比例。

这样盘的模型就制作好了,接下来就按照“运气”进行筛选。转动红色的指针,指针指到哪里,哪一个个体就被"优选"到下一个轮回。一般需要筛选出几个个体,就转几次轮盘。
这样做的理论依据:
可以通过设置 评价函数 ,使较优的值的评价分数较高,这样相应的它在轮盘中所占的圆心角就越大,被选择的机会就越大,参与繁殖的机会就越大,这也符合自然法则中的优胜略汰。但是通过这种转轮盘的方式,没有直接选择最好的那一个,也给了劣势群体的一些机会,说不定有些:个体的得分不高,但是他的后代可能产生更优的性状,这种几率发生的比较低,但也是自然法则中的一个现象,所以赌轮盘这种优选方式需要考虑这种情况。
赌轮盘制作函数:
1.将初始化的二进制值,通过函数Binary2Decimal转化为十进制
2.十进制直接计算得到函数值
3. 拿着所有值减去最小值,并加以上一个数
4. 计算所有的个体的的占比,用于制作轮盘
- def calculate_suitability(self,input,error=0.01):
- y_get = list(map(lambda x:f(x),input))
- y_get_ = y_get-np.min(y_get)+error #让最小的那个数值 不为零
- out_data = [y_get__/np.sum(y_get_) for y_get__ in y_get_]
- return out_data
摇轮盘函数:
这个函数自行理解,关于需要优选的次数:为了保证后代的种群的数量保持不变,所以有几个个体就需要摇几次。当然个体所占圆心角较大的被优选的机会更多,可以是一个个体被多次优选到。
- def optimize_choose(self,input_Data):
- out_data_index = []
- for index in range(len(input_Data)): # 需要优选的次数
- random_value = np.random.rand(1) # 随机轮盘指针位置
- sum_value = 0
- for _index in range(len(input_Data)): # 手动转到对应位置
- sum_value = sum_value + input_Data[_index]
- if random_value <= sum_value:
- out_data_index.append(_index)
- break
- else:
- continue
- return out_data_index
>>锦标赛:2021.12.24早上更新一点
锦标赛制,保留最好的那个一个,或者择优录取,是人类绝大数情况下进行社交活动中所采用的较为公平的优选方式,是否得到“优选”,完全取决于个体的实力,而不考虑个体的社会关系。一般的做法,将个体按照评价标准进行打分,按照打分从高到低排序,然后“优选”取前N个个体。
>>"小生境":
小生境优选,可以说综合了赌轮盘和锦标赛,考虑个体在种群中的“社会关系”,避免陷入局部最优。这个后面有机会,单独开一个博客来讲。
4. 交叉-变异 (2021.12.27更新)
后代之间的基因融合(交叉、变异)的基本原理如下图所示。

这里是简单的原理描述,一些超参数的设置解释如下:
1. 关于交叉的起始位置一般是随机的,通过设置随机函数来实现
2. 关于交叉的长度也可以按照经验来指定,或者如上图所示的
3. 值得注意的是:交叉规则也完全可以由经验来设计
4. 关于变异的位置,这里也采用随机的位置。
5. 变异的长度一般是一位
6.交叉是子代之间一定进行的,但是变异却是按照概率发生的。
一个具体的交叉变异函数代码如下:
- def cross_variation(self,last_groups, opt_chose):
- '''
- :fct 对优选的后代进行交叉、变异
- :param last_groups: 上一代种群中的所有个体
- :param opt_chose: 经过优选 算法产生的"优秀个体的索引",
- 用于从上一代种群中筛选出优秀的个体进行下一步的繁殖
- :return:进行交叉变异后的下一代
- '''
- opt_chose_data = [] # 优选的后的结果
- for opt_flag in opt_chose:
- opt_chose_data.append(last_groups[opt_flag,:])
- cross_Data = copy.deepcopy(opt_chose_data)
- # 分组 因为交叉和变异,需要两两进行,所以要进行分组
- index_groups = list(range(0,len(last_groups)))
- np.random.shuffle(index_groups) #分组是随机的,按顺序排完之后,再次打乱
- # 染色体 交叉,
- # 1. 交叉的位置是随机的
- for index in range(0,int(len(index_groups)/2)):
- index = index*2
- # 交叉的起始位置 是随机的
- variation_site = np.random.choice(list(range(0,np.shape(last_groups)[-1]))[1:])
- variation_data = opt_chose_data[index_groups[index]][variation_site:] # 保存 中间变量
- cross_Data[index_groups[index]][variation_site:] = opt_chose_data[index_groups[index+1]][variation_site:]
- cross_Data[index_groups[index + 1]][variation_site:] = variation_data
- # 染色体 变异
- # 2. 变异是否 发生是随机的
- variation_Data = copy.deepcopy(cross_Data)
- variation_value = 1/np.shape(last_groups)[-1] #变异发生的概率
- for index in range(len(variation_Data)):
- variation_random_value = np.random.rand(1) #当前变异发生的概率
- if variation_random_value>=variation_value:
- # 变异发生的位置
- variation_site = np.random.choice(list(range(0, np.shape(last_groups)[-1]))[1:])
- # 变异的表示:0->1: 1->0
- variation_Data[index][variation_site] = 1 - variation_Data[index][variation_site]
- return np.array(variation_Data)
该函数中做一下解释:
关于变异的发生概率一般设置为染色体长度的倒数,在繁殖中,会产生一个随机数,若随机数的概率大于变异发生概率时,就让其变异。
5. 遗传循环
遗传循环:包括以下几个步骤
>>解码
>>计算生存适应度
>>优选
>>交叉、变异
>>解码,求输出值
函数如下:
- def run(self):
- init_groups = self._groups
- data_collect = []
- for epochs in range(100):
- y_group = [self.Binary2Decimal(Binary,False) for Binary in init_groups]
- y_suit = self.calculate_suitability(y_group)
- opt_chose = self.optimize_choose(y_suit)
- cross_variation_data = self.cross_variation(init_groups, opt_chose)
- init_groups = copy.deepcopy(cross_variation_data)
- y_get = list(map(lambda x: f(self.Binary2Decimal(x,True)), init_groups))
- data_collect.append(np.max(y_get))
- print('epoch: %d 当前最优值为%f'%(epochs,np.max(data_collect)))
结果

汇总代码:
- import numpy as np
- import copy
- '''目标函数'''
- # f = lambda x:−x^4+37x^3−476x^2+2540x−4779 −(x−5)(x−10)(x−6)(x−16)+21
- f = lambda x:-x**4+37*x**3-476*x**2+2540*x-4779
- class genetic_algorithms():
- def __init__(self,N=8,M=5):
- _groups = np.zeros([N,M],int)
- for index in range(0,N):
- for _index in range(0,M):
- _groups[index,_index] = np.random.choice([0,1])
- self._groups = _groups
- def Binary2Decimal(self,Binary,include_negative=False):
- '''
- fct: 将二进制转化为十进制
- :param Binary: 输入的二进制数组
- :param include_negative: 最高位是不是 符号位
- :return:
- '''
- sum = 0
- bit_flag = -1 if include_negative==True else 1
- for index in range(1,len(Binary)):
- sum = sum + Binary[-index]*2**(index-1)
- if bit_flag==-1:
- if Binary[0]==1:
- sum = sum * bit_flag
- else:
- sum = sum + 0
- else:
- sum = sum + Binary[0]*2**(index)
- return sum
- # 适宜度的设计 很重要
- # 会出现负数,负数怎么处理
- # 这里将 负数变成正数 进行处理
- def calculate_suitability(self,input,error=0.01):
- y_get = list(map(lambda x:f(x),input))
- y_get_ = y_get-np.min(y_get)+error #让最小的那个数值 不为零
- out_data = [y_get__/np.sum(y_get_) for y_get__ in y_get_]
- return out_data
- def optimize_choose(self,input_Data):
- out_data_index = []
- for index in range(len(input_Data)): # 需要优选的次数
- random_value = np.random.rand(1) # 随机轮盘指针位置
- sum_value = 0
- for _index in range(len(input_Data)): # 手动转到对应位置
- sum_value = sum_value + input_Data[_index]
- if random_value <= sum_value:
- out_data_index.append(_index)
- break
- else:
- continue
- return out_data_index
- def cross_variation(self,last_groups, opt_chose):
- '''
- :fct 对优选的后代进行交叉、变异
- :param last_groups: 上一代种群中的所有个体
- :param opt_chose: 经过优选 算法产生的"优秀个体的索引",
- 用于从上一代种群中筛选出优秀的个体进行下一步的繁殖
- :return:进行交叉变异后的下一代
- '''
- opt_chose_data = [] # 优选的后的结果
- for opt_flag in opt_chose:
- opt_chose_data.append(last_groups[opt_flag,:])
- cross_Data = copy.deepcopy(opt_chose_data)
- # 分组 因为交叉和变异,需要两两进行,所以要进行分组
- index_groups = list(range(0,len(last_groups)))
- np.random.shuffle(index_groups) #分组是随机的,按顺序排完之后,再次打乱
- # 染色体 交叉,
- # 1. 交叉的位置是随机的
- for index in range(0,int(len(index_groups)/2)):
- index = index*2
- # 交叉的起始位置 是随机的
- variation_site = np.random.choice(list(range(0,np.shape(last_groups)[-1]))[1:])
- variation_data = opt_chose_data[index_groups[index]][variation_site:] # 保存 中间变量
- cross_Data[index_groups[index]][variation_site:] = opt_chose_data[index_groups[index+1]][variation_site:]
- cross_Data[index_groups[index + 1]][variation_site:] = variation_data
- # 染色体 变异
- # 2. 变异是否 发生是随机的
- variation_Data = copy.deepcopy(cross_Data)
- variation_value = 1/np.shape(last_groups)[-1] #变异发生的概率
- for index in range(len(variation_Data)):
- variation_random_value = np.random.rand(1) #当前变异发生的概率
- if variation_random_value>=variation_value:
- # 变异发生的位置
- variation_site = np.random.choice(list(range(0, np.shape(last_groups)[-1]))[1:])
- # 变异的表示:0->1: 1->0
- variation_Data[index][variation_site] = 1 - variation_Data[index][variation_site]
- return np.array(variation_Data)
- def run(self):
- init_groups = self._groups
- data_collect = []
- for epochs in range(100):
- y_group = [self.Binary2Decimal(Binary,False) for Binary in init_groups]
- y_suit = self.calculate_suitability(y_group)
- opt_chose = self.optimize_choose(y_suit)
- cross_variation_data = self.cross_variation(init_groups, opt_chose)
- init_groups = copy.deepcopy(cross_variation_data)
- y_get = list(map(lambda x: f(self.Binary2Decimal(x,True)), init_groups))
- data_collect.append(np.max(y_get))
- print('epoch: %d 当前最优值为%f'%(epochs,np.max(data_collect)))
- # print('epoch: %d 当前最优值为%f'%(epochs,np.max(y_get)))
- if __name__=='__main__':
- obj = genetic_algorithms(N=8,M=5)
- obj.run()
-
相关阅读:
1.1_4操作系统的运行机制与体系结构
云计算对象存储服务
ASP.NET Core 6.0对热重载的支持
Algorithm Review 5
【Vue域名动态配置】
机器人过程自动化(RPA)入门 3. 顺序、流程图和控制流程
Spring Boot 自动注入失败的原因
【排序14:存在重复元素】
linux部署Django项目
Python大作业——爬虫+可视化+数据分析+数据库(可视化篇)
- 原文地址:https://blog.csdn.net/weixin_44503976/article/details/126654578