-
RNA-seq 详细教程:实验设计(2)
学习目标
-
了解设置重复对于 RNA-seq分析的重要性 -
了解生物重复次数、测序深度和鉴定到的差异表达基因之间的关系 -
了解如何设计 RNA-seq实验,以避免批次效应
1. 注意事项
了解
RNA提取和RNA-seq文库制备实验过程中的步骤,有助于设计RNA-seq实验,但有一些特殊的注意事项需要明确:-
重复次数和类型 -
避免混淆 -
处理批次效应
2. 重复
实验重复可以通过技术重复或生物学重复来实现,如下图:

Klaus B., EMBO J (2015) 34: 2727-2730 -
技术重复
使用相同的生物样本重复实验步骤,以准确测量技术差异并在分析过程中将其去除。
-
生物学重复
使用相同条件下的不同生物样本来衡量样本间的差异。
在微阵列时代,技术重复被认为是必要的;然而,当前的
RNA-seq技术,技术差异远低于生物差异,因此不需要技术重复。相反,生物重复对于差异表达分析是绝对必要的。对于差异表达分析,生物学重复越多,对生物学变异的估计就越好,我们对平均表达水平的估计也就越精确。因此,数据可以进行更准确的建模并识别更多差异表达的基因。

Liu, Y., et al., Bioinformatics (2014) 30(3): 301–304 如上图所示,生物重复比测序深度更重要,测序深度是每个样本测序的读数总数。该图显示了测序深度和重复次数对鉴定出的差异表达基因数的关系。与增加测序深度相比,重复次数的增加往往会得到更多的差异表达基因。因此,通常更多的重复比更高的测序深度更好,但需要注意的是,检测低表达的差异表达基因和执行异构体水平(可变剪切)的差异表达分析需要更高的深度。
下面列出了一些关于重复和测序深度的建议,用于实验规划:
-
通用建议:
-
ENCODE 建议每个样本有 3000 万个 SE reads。 -
如果有大量的重复 (>3),每个样本 1500 万次 reads通常就足够了。 -
如果可能,进行更多的生物重复。 -
通常建议读取长度 >= 50 bp
-
-
含有低表达基因:
-
同样,重复比测序深度更有作用。 -
深度更深,至少有 30-60 百万 reads,具体取决于表达水平。
-
-
异构体水平的差异表达:
-
新亚型的深度应该更大(每个样本 > 6000 万 reads)。 -
对 RNA质量进行质控。
-
-
其他类型的
RNA分析(内含子保留、small RNA-Seq等):-
取绝于具体的分析
-
★
总之,尽量做生物学重复。
”3. Confound
Confounding是指:无法区分结果是由什么原因导致的。例如,我们知道性别对基因表达有很大影响,如果我们所有的对照组小鼠都是雌性而所有处理组小鼠都是雄性,那么我们的治疗效果就会被性别混淆。我们无法区分是处理的作用和性别的作用。

-
如何避免:
-
如果可能,确保每种情况下的动物都是相同的性别、年龄和批次。 -
如果不可能,则确保在不同条件下平均分配动物。

deconfound 4. 批次效应
批次效应是
RNA-seq分析的一个重要问题,仅由批次效应就能导致显著的表达差异。
Hicks SC, et al., bioRxiv (2015) -
如何确定是否有批次效应
-
是否所有的 RNA提取都是在同一天进行的? -
是否所有的文库构建都是在同一天进行的? -
是否同一个人对所有样品进行了 RNA提取与文库制备? -
是否对所有样品使用了相同的试剂? -
是否在同一地点进行 RNA提取与文库制备?
如果任何一个答案是“否”,那么就存在批次效应。
5. 建议
-
如果可能,以避免分批的方式设计实验。 -
如果无法避免分批:
-
不要按批次混淆实验:

Hicks SC, et al., bioRxiv (2015) -
跨批次拆分不同样本组的重复。重复次数越多越好(超过 2 个)。
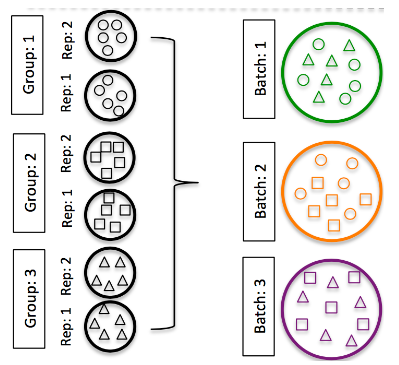
Hicks SC, et al., bioRxiv (2015) -
请务必在实验数据中包含批次信息。在分析过程中,如果没有混淆,可以回归出由于批次引起的变异,所以有这些信息,它不会影响结果。

欢迎Star -> Github 学习目录( <-点击跳转)
国内链接 -> 学习目录( <-点击跳转)
本文由 mdnice 多平台发布
-
-
相关阅读:
会员权益营销中,等级会员的五种权益设置
如何在 Ubuntu 上启用自动更新
Git 基础使用
【LeetCode算法系列题解】第71~75题
echarts+高德地图结合开发
大数据Flink(九十六):DML:Deduplication
J2EE从入门到入土02.Set及Map集合解析
【运维心得】如何进行应用日志分析?
从0到1配置TensorRT环境
V8垃圾回收
- 原文地址:https://blog.csdn.net/swindler_ice/article/details/128056008